Los ARN potenciadores (eRNA) representan una clase de moléculas de ARN no codificantes relativamente largas (50-2000 nucleótidos) transcritas a partir de la secuencia de ADN de las regiones potenciadoras . Se detectaron por primera vez en 2010 mediante el uso de técnicas de todo el genoma, como RNA-seq y ChIP-seq . [1] [2] Los eRNA se pueden subdividir en dos clases principales: eRNA 1D y eRNA 2D, que difieren principalmente en términos de su tamaño, estado de poliadenilación y direccionalidad transcripcional. [3] La expresión de un eRNA determinado se correlaciona con la actividad de su potenciador correspondiente en los genes diana. [4] Cada vez hay más pruebas que sugieren que los eRNA desempeñan un papel activo en la regulación transcripcional en cis y en trans , y aunque sus mecanismos de acción siguen sin estar claros, se han propuesto algunos modelos. [3]
Los potenciadores como sitios de transcripción extragénica se descubrieron inicialmente en estudios de todo el genoma que identificaron a los potenciadores como regiones comunes de unión de la ARN polimerasa II (ARN pol II) y la transcripción de ARN no codificante . [1] [2] Se encontró que el nivel de interacción ARN pol II-potenciador y la formación de transcripción de ARN eran altamente variables entre estos estudios iniciales. Usando picos de firma de cromatina explícitos, se encontró que una proporción significativa (~70%) de sitios de inicio de transcripción de ARN Pol II extragénicos se superponían a los sitios potenciadores en macrófagos murinos . [5] De 12.000 potenciadores neuronales en el genoma del ratón , se encontró que casi el 25% de los sitios se unían a ARN Pol II y generaban transcripciones . [6] En estudios paralelos, se identificaron 4.588 sitios de unión de ARN Pol II extragénicos de alta confianza en macrófagos murinos estimulados con el mediador inflamatorio lipopolisacárido para inducir la transcripción. [2] Estos eRNAs, a diferencia de los ARN mensajeros (ARNm), carecían de modificación por poliadenilación , eran generalmente cortos y no codificantes, y se transcribían bidireccionalmente. Estudios posteriores revelaron la transcripción de otro tipo de eRNAs, generados a través de transcripción unidireccional, que eran más largos y contenían una cola de poli A. [7] Además, los niveles de eRNA se correlacionaron con los niveles de ARNm de genes cercanos , lo que sugiere el potencial papel regulador y funcional de estas moléculas de ARN potenciador no codificante . [1]
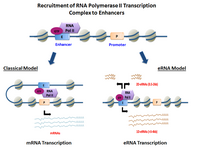
Los eRNA se transcriben a partir de secuencias de ADN situadas aguas arriba y aguas abajo de las regiones potenciadoras extragénicas . [8] Anteriormente, varios potenciadores modelo han demostrado la capacidad de reclutar directamente la ARN Pol II y los factores de transcripción generales y formar el complejo de preiniciación (PIC) antes del sitio de inicio de la transcripción en el promotor de los genes . En ciertos tipos de células , los potenciadores activados han demostrado la capacidad de reclutar la ARN Pol II y también de proporcionar una plantilla para la transcripción activa de sus secuencias locales . [2] [1]
Dependiendo de la direccionalidad de la transcripción , las regiones potenciadoras generan dos tipos diferentes de transcripciones no codificantes , 1D-eRNA y 2D-eRNA. La naturaleza del complejo de preiniciación y los factores de transcripción específicos reclutados al potenciador pueden controlar el tipo de eRNA generados. Después de la transcripción , la mayoría de los eRNA permanecen en el núcleo . [9] En general, los eRNA son muy inestables y se degradan activamente por el exosoma nuclear . No todos los potenciadores se transcriben, y los potenciadores no transcritos superan en gran medida a los transcritos en el orden de magnitud de docenas de miles en cada tipo de célula determinado . [5]
En la mayoría de los casos, la transcripción unidireccional de regiones potenciadoras genera eRNA largos (>4 kb) y poliadenilados. Los potenciadores que generan eRNA poliA+ tienen una proporción H3K4me1 /me3 más baja en su firma de cromatina que los eRNA 2D. [7] Los eRNA poliA+ son distintos de los politranscritos multiexónicos largos (meRNA) que se generan por iniciación de la transcripción en potenciadores intragénicos . Estos ARN largos no codificantes, que reflejan con precisión la estructura del gen huésped excepto por el primer exón alternativo , muestran un potencial de codificación pobre. [10] Como resultado, los eRNA poliA+ 1D pueden representar un grupo mixto de verdaderos ARN con plantilla potenciadora y ARN multiexónicos.
La transcripción bidireccional en los sitios potenciadores genera ARNm no poliadenilados y relativamente más cortos (0,5-2 kb). Los potenciadores que generan ARNm poliA tienen una firma de cromatina con una proporción H3K4me1/me3 más alta que los ARNm 1D. En general, la transcripción potenciadora y la producción de ARNm bidireccionales demuestran una fuerte correlación de la actividad potenciadora en la transcripción génica . [11]
Arner et al. [12] identificaron 65.423 potenciadores transcritos (que producen ARNe) entre 33 tipos de células diferentes en diferentes condiciones y diferentes momentos de estimulación. La transcripción de potenciadores generalmente precedió a la transcripción de factores de transcripción que, a su vez, generalmente precedieron a la transcripción de genes mediante ARN mensajero (ARNm).
Carullo et al. [13] examinaron un tipo particular de célula, neuronas (de cultivos neuronales primarios). Exhibieron 28,492 potenciadores putativos que generaban eRNAs. Estos eRNAs a menudo se transcribieron desde ambas hebras del ADN potenciador en direcciones opuestas. Carullo et al. [13] utilizaron estas neuronas cultivadas para examinar el momento de los eRNA potenciadores específicos en comparación con los ARNm de sus genes objetivo. Las neuronas cultivadas se activaron y el ARN se aisló de esas neuronas a los 0, 3.75, 5, 7.5, 15, 30 y 60 minutos después de la activación. En estas condiciones experimentales, encontraron que 2 de los 5 potenciadores del gen temprano inmediato (IEG) FOS , es decir, el potenciador 1 de FOS y el potenciador 3 de FOS, se activaron e iniciaron la transcripción de sus eRNAs (eRNA1 y eRNA3). El eRNA1 y el eRNA3 de FOS aumentaron significativamente en 7,5 minutos, mientras que el mRNA de FOS solo aumentó 15 minutos después de la estimulación. Se observaron patrones similares en los IEG FOSb y NR4A1 , lo que indica que, en muchos IEG, la inducción del eRNA precede a la inducción del mRNA en respuesta a la activación neuronal.
Si bien algunos potenciadores pueden activar sus promotores objetivo en sus genes objetivo sin transcribir eRNA, la mayoría de los potenciadores activos transcriben eRNA durante la activación de sus promotores objetivo. [14]
Las funciones del ARNe descritas a continuación se han descrito en diversos sistemas biológicos, a menudo demostradas con un pequeño número de pares específicos de genes potenciadores-objetivos. No está claro hasta qué punto las funciones del ARNe descritas aquí se pueden generalizar a la mayoría de los ARNe.
.jpg/440px-Regulation_of_transcription_in_metazoans_(animals).jpg)
Los bucles cromosómicos que se muestran en la figura, que llevan un potenciador al promotor de su gen objetivo, pueden ser dirigidos y formados por el ARNe transcrito a partir del potenciador después de que éste se activa.
Un ARN potenciador transcrito (eRNA) que interactúa con el complejo de proteínas mediadoras (ver Figura), especialmente la subunidad 12 del mediador ( MED12 ), parece ser esencial en la formación del bucle cromosómico que lleva al potenciador a una asociación cercana con el promotor del gen objetivo del potenciador en el caso de cinco genes estudiados por Lai et al. [15] [16] [17] Hou y Kraus, [18] describen otros dos estudios que informan resultados similares. Arnold et al. [19] revisan otros 5 casos donde el eRNA es activo en la formación del bucle potenciador-promotor.
Un eRNA bien estudiado es el eRNA del potenciador que interactúa con el promotor del gen del antígeno prostático específico (PSA). [20] El eRNA del PSA está fuertemente regulado al alza por el receptor de andrógenos . El eRNA del PSA alto tiene entonces un efecto dominó. El eRNA del PSA se une y activa el complejo proteico del factor de elongación de transcripción positiva P-TEFb que luego puede fosforilar la ARN polimerasa II (RNAP II), iniciando su actividad en la producción de ARNm . P-TEFb también puede fosforilar el factor de elongación negativo NELF (que pausa la ARNP II dentro de los 60 nucleótidos después de que comienza la iniciación del ARNm). El NELF fosforilado se libera de la ARNP II, lo que permite que la ARNP II tenga una progresión productiva del ARNm (ver Figura). El eRNA del PSA regulado al alza aumenta así la expresión de 586 genes sensibles al receptor de andrógenos. La eliminación del eRNA del PSA o la eliminación de un conjunto de nucleótidos del eRNA del PSA provoca una disminución de la presencia de ARN polimerasa II fosforilada (activa) en estos genes, lo que provoca una reducción de su transcripción.
La proteína NELF , factor de elongación negativo, también puede liberarse de su interacción con la ARN polimerasa II mediante la interacción directa con algunos ARNm. Schaukowitch et al. [21] demostraron que los ARNm de dos genes tempranos inmediatos (IEG) interactuaron directamente con la proteína NELF para liberar a NELF de la ARN polimerasa II que se detuvo en los promotores de estos dos genes, lo que permitió que estos dos genes se expresaran.
Además, los eRNA parecen interactuar con hasta otras 30 proteínas. [19] [17] [18]

Las nociones de que no todos los potenciadores se transcriben al mismo tiempo y que la transcripción de eRNA se correlaciona con la actividad específica del potenciador apoyan la idea de que los eRNA individuales tienen funciones biológicas distintas y relevantes. [3] Sin embargo, todavía no hay consenso sobre la importancia funcional de los eRNA. Además, los eRNA pueden degradarse fácilmente a través de exosomas y desintegración mediada por sinsentidos , lo que limita su potencial como importantes reguladores transcripcionales. [22] Hasta la fecha, se han propuesto cuatro modelos principales de la función de eRNA, [3] cada uno respaldado por diferentes líneas de evidencia experimental .
Dado que varios estudios han demostrado que la RNA Pol II se puede encontrar en una gran cantidad de regiones extragénicas , es posible que los eRNA simplemente representen el producto de una transcripción aleatoria “con fugas” y no tengan importancia funcional. [5] Por lo tanto, la actividad no específica de la RNA Pol II permitiría el ruido transcripcional extragénico en sitios donde la cromatina ya está en un estado abierto y transcripcionalmente competente. Esto explicaría incluso la expresión de eRNA específica de tejido [23] ya que los sitios abiertos también son específicos de tejido.
La transcripción génica mediada por la ARN polimerasa II induce una apertura local del estado de la cromatina a través del reclutamiento de las acetiltransferasas de histonas y otros modificadores de histonas que promueven la formación de eucromatina . Se propuso que la presencia de estas enzimas también podría inducir una apertura de la cromatina en las regiones potenciadoras , que suelen estar presentes en lugares distantes pero que pueden reclutarse para dirigirse a genes diana a través de la formación de bucles en el ADN . [24] En este modelo, los eRNA se expresan, por tanto, en respuesta a la transcripción de la ARN polimerasa II y, por tanto, no tienen ninguna función biológica .
Mientras que los dos modelos anteriores implicaban que los eRNA no eran funcionalmente relevantes, este mecanismo establece que los eRNA son moléculas funcionales que exhiben actividad cis . En este modelo, los eRNA pueden reclutar localmente proteínas reguladoras en su propio sitio de síntesis. En apoyo de esta hipótesis, se cree que las transcripciones que se originan a partir de potenciadores aguas arriba del gen de la ciclina D1 sirven como adaptadores para el reclutamiento de las acetiltransferasas de histonas . Se encontró que el agotamiento de estos eRNA condujo al silenciamiento transcripcional de la ciclina D1 . [9]
El último modelo implica la regulación transcripcional por eRNAs en ubicaciones cromosómicas distantes . A través del reclutamiento diferencial de complejos proteicos , los eRNAs pueden afectar la competencia transcripcional de loci específicos . Evf-2 representa un buen ejemplo de este eRNA transregulador , ya que puede inducir la expresión de Dlx2, que a su vez puede aumentar la actividad de los potenciadores Dlx5 y Dlx6 . [25] Los eRNAs que actúan en trans también podrían funcionar en cis , y viceversa.
La detección de eRNAs es bastante reciente (2010) y ha sido posible gracias al uso de técnicas de investigación de todo el genoma, como la secuenciación de ARN ( RNA-seq ) y la inmunoprecipitación-secuenciación de cromatina ( ChIP-seq ). [1] La RNA-seq permite la identificación directa de eRNAs haciendo coincidir la transcripción detectada con la secuencia potenciadora correspondiente mediante análisis bioinformáticos . [26] [4] La ChIP-seq representa una forma menos directa de evaluar la transcripción potenciadora , pero también puede proporcionar información crucial, ya que las marcas de cromatina específicas se asocian con potenciadores activos . [27] Aunque algunos datos siguen siendo controvertidos, el consenso en la literatura es que la mejor combinación de modificaciones postraduccionales de histonas en potenciadores activos está formada por H2AZ , H3K27ac y una alta proporción de H3K4me1 sobre H3K4me3 . [27] [28] [29] Los experimentos de ChIP también se pueden realizar con anticuerpos que reconocen la ARN Pol II , que se puede encontrar en sitios de transcripción activa . [5] La detección experimental de eRNA se complica por su baja estabilidad endógena conferida por la degradación de exosomas y la descomposición mediada por sinsentido . [22] Un estudio comparativo mostró que los ensayos de enriquecimiento para ARN con capuchón y nacientes (con estrategias como la ejecución continua de núcleos y la selección de tamaño) podrían capturar más eRNA en comparación con la secuenciación de ARN canónica . [30] Estos ensayos incluyen Global/Precision Run-on con selección de capuchón (GRO/PRO-cap), capped-small RNA-seq (csRNA-seq), Native Elongating Transcript-Cap Analysis of Gene Expression (NET-CAGE) y Precision Run-On sequencing (PRO-seq). [31] Sin embargo, el hecho de que los eRNA tienden a expresarse a partir de potenciadores activos podría hacer que su detección sea una herramienta útil para distinguir entre potenciadores activos e inactivos .
La evidencia de que los eRNA causan efectos posteriores en la eficiencia de la activación del potenciador y la transcripción génica sugiere sus capacidades funcionales y su importancia potencial. Se ha demostrado que el factor de transcripción p53 se une a regiones potenciadoras y genera eRNA de una manera dependiente de p53 . [32] En el cáncer , p53 desempeña un papel central en la supresión tumoral , ya que se ha demostrado que las mutaciones del gen aparecen en el 50% de los tumores . [33] Se ha demostrado que estas regiones potenciadoras unidas a p53 (p53BER) interactúan con múltiples dianas génicas locales y distales implicadas en la proliferación y supervivencia celular . Además, se ha demostrado que los eRNA generados por la activación de p53BER son necesarios para la transcripción eficiente de los genes diana de p53 , lo que indica el probable papel regulador importante de los eRNA en la supresión tumoral y el cáncer . En general, se ha demostrado que las mutaciones en eRNA muestran un comportamiento fenotípico similar en la oncogénesis en comparación con el ARN codificador de proteínas. [34]
Las variaciones en los potenciadores se han relacionado con enfermedades humanas, pero actualmente no es posible aplicar un enfoque terapéutico para manipular la actividad de los potenciadores . Con la aparición de los ARNm como componentes importantes de la actividad de los potenciadores , herramientas terapéuticas potentes como el ARNi pueden proporcionar vías prometedoras para atacar la alteración de la expresión génica.