La proteína FAM214A , también conocida como familia de proteínas con similitud de secuencia 214, A (FAM214A), es una proteína que, en los seres humanos, está codificada por el gen FAM214A . FAM214A es un gen con función desconocida que se encuentra en el locus q21.2-q21.3 del cromosoma 15 (humano) . [5] El producto proteico de este gen tiene dos dominios conservados, uno de función desconocida (DUF4210) y otro llamado Chromosome_Seg. [6] Aunque la función de la proteína FAM214A no está caracterizada, se ha predicho que tanto DUF4210 como Chromosome_Seg desempeñan un papel en la segregación cromosómica durante la meiosis. [7]
El gen FAM214A se encuentra en la cadena de ADN negativa (ver Sentido (biología molecular) ) del cromosoma 15 entre la posición 52.873.514 y 53.002.014; por lo que el gen tiene una longitud de 97.303 pares de bases (pb). [5] [8] [9] FAM214A ha sido etiquetado previamente con otros dos alias, conocidos como KIAA1370 y FLJ10980. [5] Se predice que el gen FAM214A contiene 12 exones que comprenden la transcripción final de ARNm de 4231 pb después de que se ha producido la transcripción . [10] Es este producto de ARNm el que luego se traduce en la proteína FAM214A final con la ayuda de la secuencia promotora y los factores de transcripción . El promotor de la secuencia de ARNm de FAM214A fue predicho y analizado por el programa El Dorado en Genomatix. [11] Este promotor tiene una longitud de 601 pares de bases y abarca una porción del 5' UTR. [11]
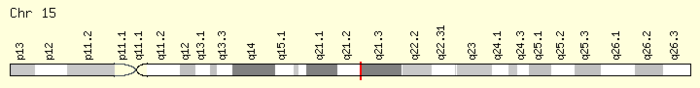
Según varias fuentes, como BioGPS y Expression Atlas, se considera que FAM214A se expresa de forma ubicua (o casi) en niveles bajos. [12] [13] [14] Como se puede ver en la imagen de BioGPS a continuación, hay un nivel de expresión significativamente mayor en células y tejidos relacionados con el sistema inmunitario, lo que sugiere un papel inmunitario; sin embargo, no ha habido evidencia in situ específica que respalde esta afirmación. Se han recopilado datos de expresión de varios estudios realizados en una amplia gama de genes, por lo tanto, algunos de los datos son contradictorios por naturaleza.
La función de la proteína FAM214A en humanos aún es desconocida; Sin embargo, hay tres asociaciones de términos funcionales que incluyen "proceso biológico", "componente celular" y "función molecular", que describen la función de esta proteína en The Gene Ontology que predicen las implicaciones de su función primaria in vivo . [15] [16] El producto proteico de FAM214A consta de 1076 aminoácidos (aa), se ha predicho que tiene una masa molecular de 121.700 daltons y tiene un punto isoeléctrico alrededor de pH 7,7. [6] [17] [18] Se predice que esta proteína permanecerá en el núcleo después de la transcripción basándose en su falta de secuencia de péptido señal y las predicciones del programa PSORTII. [19] Debido al empalme alternativo, se han observado otras dos isoformas (Q32MH5-2 y Q32MH5-3). Difieren ligeramente del producto primario. [20] La isoforma 2 tiene cuatro aminoácidos diferentes desde las bases 960-960 y le falta el final de la secuencia desde las bases 964-1076. [20] La isoforma 3 tiene siete aminoácidos adicionales añadidos al comienzo de la secuencia después de la metionina. [20]
Después de ser traducida, se predice que la proteína FAM214A permanecerá en el núcleo por más de un tipo de subprograma en PSORT II. [19] Esta proteína tiene una señal pat4, una de las dos señales de localización nuclear (NLS) "clásicas", que comienza en el residuo 709. [21] Aunque no tiene la segunda NLS "clásica", pat7, ni la NLS bipartita "no clásica", aún se predice que será dirigida al núcleo por la puntuación NCNN. [21] [22] Esta puntuación predice si la proteína es dirigida al núcleo o al citoplasma según la secuencia de aminoácidos. [21] [22] Para la proteína FAM214A, la puntuación NCNN predijo la localización nuclear con un 94,1% de certeza. [21] [22] Con base en esta información, PSORT genera una predicción general de la localización subcelular de la proteína. Para FAM214A, los valores previstos fueron 69,6% para el núcleo en comparación con 13,0% para las mitocondrias, 8,7% para el citoplasma y 4,3% para las vesículas secretoras y el retículo endoplásmico. [19]
Es muy probable que esta proteína no sufra una cantidad significativa de modificaciones postraduccionales debido a la falta de la secuencia de péptido señal predicha por NetNGlyc y NetOGlyc en el servidor web ExPASy. [24] [25] Esto se debe a que gran parte de la maquinaria intracelular que realiza modificaciones postraduccionales requiere que la proteína se mueva a través de orgánulos como el retículo endoplasmático y el aparato de Golgi . Sin una secuencia de péptido señal, la proteína generalmente no sale del núcleo, lo que fue predicho por PSORT II como se describió anteriormente. [19]
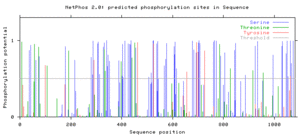
Se realizó un análisis SAPS de esta proteína en comparación con la base de datos swp23s.q, que indicó la presencia de una cantidad anormalmente grande de aminoácidos de serina y una cantidad anormalmente pequeña de aminoácidos de alanina en esta proteína. [17] Según un artículo de revisión de Fayard et al., la quinasa dependiente de fosfoinosítido 2 (PDK2) es una quinasa de serina/treonina que es importante para regular el ciclo celular. Debido a que la proteína FAM214A tiene una mayor cantidad de grupos de serina de lo que se considera normal, existe la posibilidad de que la PDK2 tenga un efecto importante en esta proteína. [26] Para determinar si realmente se predijo que la cantidad excesiva de serinas estaría fosforilada, la secuencia de la proteína se ejecutó a través del programa NetPhos desde el servidor web ExPASy. [23] Este programa predijo la fosforilación de 69 serinas, 14 treoninas y 9 tirosinas. [23] Según el análisis SAPS anterior, hay un total de 134 serinas, lo que indica que se prevé que aproximadamente la mitad se fosforilarán in vivo . A la derecha se muestra un diagrama de las predicciones de fosforilación.
El programa NetCorona en ExPASy predijo otro tipo de modificación postraduccional para la proteína FAM214A. [27] El programa predijo un único sitio de escisión entre la posición 214 y 215 en la secuencia de la proteína FAM214A después de la traducción. [27]
Se predicen varios sitios de unión de factores de transcripción para la secuencia promotora FAM214A. [11] Algunos de los que tienen la mayor confianza predicha se proporcionan en la siguiente tabla. [11]
Posibles factores de transcripción que se prevé que se unan a la secuencia promotora de FAM214A
La única proteína predicha según STRING para interactuar con la proteína FAM214A se llama MFSD6L . Esta proteína pertenece a la superfamilia de facilitadores principales y se predice que es una proteína transmembrana. Al igual que FAM214A, la función de esta proteína aún no se ha caracterizado a través de experimentación o investigación. [28] [29] Debido a que esta proteína MFSD6L es la única interacción de la proteína FAM214A predicha con alguna certeza, la secuencia para ella se ejecutó a través del programa PSORT II. Los datos del subprograma NLS predijeron la presencia de una sola secuencia NLS pat4 y dos pat7, lo que indica una posible localización nuclear. [19] [21] Por otro lado, la puntuación NCNN predijo la localización citoplasmática con un 94,1 % de certeza, lo que dejó la puntuación general PSORT II en 39,1 % membrana plasmática, 39,1 % retículo endoplásmico, 4,3 % vacuolar, 4,3 % vesículas del sistema secretor, 4,3 % Golgi, 4,3 % mitocondrial y 4,3 % nuclear. [21] [22] Esto es contradictorio ya que hay tres señales de localización nuclear totales, pero esto puede deberse al hecho de que la naturaleza transmembrana significativa de la proteína MFSD6L puede estar causando problemas con estas predicciones. [21]
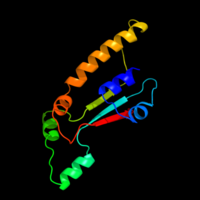
La estructura secundaria de la proteína FAM214A consta de varias hélices alfa y láminas beta , como lo predijeron Biology Workbench y P rotein H omology/analog Y Recognition Engine (PHYRE). [30] [31] El programa PHYRE predice que el 66 por ciento de la estructura secundaria de FAM214A está desordenada y, por lo tanto, no se puede analizar y convertir en una predicción de estructura terciaria. [30] Sin embargo, fue capaz de predecir aproximadamente el 10 por ciento de la estructura de la proteína con una significancia del 95 por ciento. [30] El diagrama para esto se muestra a la izquierda. [30]
Se ha encontrado un único gen parálogo en el cromosoma 9 del Homo sapiens y se denomina FAM214B (familia con similitud de secuencia, B). [32] FAM214B, aunque se considera un parálogo , tiene una secuencia proteica significativamente diferente de la de FAM214A. Cuando se compararon los dos entre sí en BLAST del NCBI, la única similitud significativa observada fue dentro de los últimos 200 aminoácidos (donde se encuentran los dominios DUF4210 y Chromosome_Seg). [33] Aunque la similitud entre FAM214A y B es baja, estas dos proteínas están en la misma familia de proteínas y contienen los mismos dos dominios conservados . [7] [34]
La proteína FAM214A tiene una cantidad significativa de ortólogos en una gran cantidad de grupos taxonómicos, incluidos Mammalia , Aves , Reptilia , Amphibia , Actinopterygii , Echinoidea , Insecta , Trematoda , Crustacea , Tricoplacia, Anthozoa y Eurotiomycetes . [35] Esto indica que la proteína FAM214A está bien conservada en Eukaryotes , pero no parece estar conservada en Bacteria o Archaea . En todos los ortólogos, la región más conservada estaba cerca del final de la proteína donde están los dominios conservados (ver a continuación). Se encontraron ortólogos para la proteína humana FAM214A en Tuber melanosporum , Talaromyces stipitatus y Aspergillus nidulans , que divergieron hace aproximadamente 1215 millones de años.
Ortólogos de la proteína FAM214A
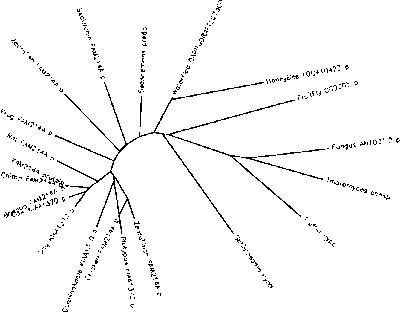
El programa CLUSTALW de Biology Workbench generó un árbol filogenético sin raíz de 20 ortólogos para demostrar la relación evolutiva entre FAM214A y sus ortólogos. [31]
Dentro de la proteína FAM214A, hay tres regiones bien conservadas. Estas incluyen una región bien conservada cerca del extremo n de la proteína y dos dominios conservados que incluyen el Dominio de Función Desconocida 4210 (DUF4210) y un dominio Chromosome_Seg cerca del extremo c . [7] A continuación, se muestra un diagrama esquemático de estas tres regiones. No se predice que la región bien conservada cerca del extremo n de la proteína contenga ningún dominio o motivo conocido; sin embargo, el sitio de escisión predicho por NetCorona anteriormente se encuentra dentro de esta región y está bien conservado en la mayoría de las proteínas ortólogas a FAM214A. [27] Los dos dominios conservados ubicados al final de esta proteína son la porción más importante del péptido según la historia evolutiva. Todos los organismos en la tabla de ortólogos anterior, excepto el ornitorrinco (al que le falta el dominio Chromosome_Seg), contienen ambos dominios conservados dentro de su secuencia de proteínas. [7]