El dominio efector de muerte ( DED ) es un dominio de interacción de proteínas que se encuentra solo en eucariotas y que regula una variedad de vías de señalización celular. [2] El dominio DED se encuentra en procaspasas inactivas ( cisteína proteasas ) y proteínas que regulan la activación de la caspasa en la cascada de apoptosis, como la proteína que contiene el dominio de muerte asociado a FAS ( FADD ). FADD recluta a la procaspasa 8 y la procaspasa 10 en un complejo de señalización inducida por muerte (DISC). Este reclutamiento está mediado por una interacción homotípica entre la procaspasa DED y un segundo DED que es el dominio efector de muerte en una proteína adaptadora que está directamente asociada con los receptores de TNF activados. La formación del complejo permite la activación proteolítica de la procaspasa en la forma de caspasa activa, lo que da como resultado el inicio de la apoptosis (muerte celular). Estructuralmente, el dominio DED es una subclase de motivo proteico conocido como pliegue de muerte y contiene 6 hélices alfa, que se parecen mucho a la estructura del dominio de muerte (DD).

DED es una subfamilia de la superfamilia DD (otros dominios reconocibles en esta superfamilia son: dominio de reclutamiento de caspasa (CARD) , dominio de pirina (PYD) y dominio de muerte (DD)). Las subfamilias se parecen estructuralmente entre sí, todas ellas (y DED en particular) están compuestas por un haz de 6 hélices alfa, pero divergen en las características de la superficie.
La estructura primaria completa de este dominio proteico no ha sido definida de manera consensual. Algunos estudios describen los residuos 2-184, pero los residuos C-terminales y N-terminales aún no han sido identificados. La presencia de aminoácidos que determinan la solubilidad y agregación a DED permitió identificar DED en diferentes proteínas, como la caspasa-8 y MC159. La estructura secundaria del dominio, como se dijo, está formada por 6 hélices alfa.
La estructura terciaria del dominio ha sido descrita a partir de la cristalización de la caspasa 8 en humanos. El método utilizado para describir la estructura fue la difracción de RAYOS X y la resolución obtenida es de 2,2 Å. [3] Los DED en esta proteína muestran un dímero unitario asimétrico , con su interfaz que contiene dos redes de enlaces de hidrógeno, que aparecen como una estructura filamentosa. La función de DED está determinada por su estructura. Hasta donde se sabe, las interacciones homotípicas que activan la caspasa y desencadenan la apoptosis están mediadas por contactos superficiales asimétricos entre socios (como DED1 y DED2 en el caso de la caspasa-8). [4] Los residuos que forman las superficies son típicamente aminoácidos cargados, pero también se puede observar un parche hidrofóbico corto en la superficie interactiva del dominio.
El dominio DED es más conocido por su papel en la apoptosis. Sin embargo, las proteínas que contienen DED también participan en otros procesos celulares, de modo que controlan las decisiones de vida y muerte de las células.
[5] La apoptosis es una muerte celular controlada y programada que confiere ventajas durante el ciclo de vida de un organismo. La vía extrínseca está dirigida por una familia de proteasas que se activan en respuesta a estímulos de muerte. Para conocer el papel de las DED en este proceso es importante observar la formación del complejo de señalización multiproteico que incluye la muerte celular (DISC).
DR4 , TRAIL-R2 y CD95 son receptores de muerte (miembros de la superfamilia de receptores de TNF ) que interactúan entre sí utilizando sus dominios de muerte intracelulares (DD). El DD de FADD, una proteína que contiene un DED, puede interactuar con estos DD descritos. Aquí, la función de FADD DED es crear una estructura estabilizada mediante la autoasociación de FADD.

[6] Estas interacciones están definidas por las hélices α1/α4 y α2/α3: los residuos Ser1, Val6, His9, Leu43, Asp44 y Glu51 de α1/α4 están en contacto con Thr21, Phe25, Lys33, Arg34, Glu37 y Glu51 de α2/α3 de la segunda molécula. Cada interacción involucra un área de 1062 Å 2 y contribuciones de cadenas laterales hidrofóbicas, enlaces de hidrógeno y puentes salinos. El homodímero final tiene una estructura orientada de modo que cada subunidad tiene los 2 sitios de interacción.
La procaspasa-8, también una proteína que contiene DED, tiene afinidad por la DED de FADD. Es por ello que son reclutadas a FADD como monómeros a través de sus DED. Esta interacción está definida por α1/α4 de la procaspasa-8 DED-A y FADD DED α2/α3 o α1/α4 de FADD DED y α2/α5 de la procaspasa-8 DED-B. La procaspasa-8 DED-B interactúa con FADD y DED-A media la formación de la cadena de la capasa-8, o viceversa. Sin embargo, en ambos casos la interacción lleva a crear un dímero entre procaspasas, lo que genera un cambio conformacional. Esta dimerización es esencial para crear el sitio activo; se libera una subunidad p12 y posteriormente se procesa a la subunidad pequeña p10. Las dos moléculas de procapasa-8 se asocian con estas subunidades p10 creando una muerte celular activa por proteasa-8. [7] [8]
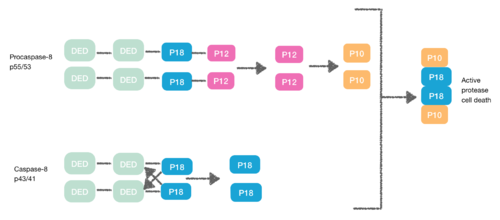
[9] Durante la creación del DISC, la procaspasa-8 también puede heterodimerizarse con otra proteína que contiene DED conocida como FLIP L. La pseudocaspasa de FLIP L tiene dos DED en tándem que son muy similares al extremo N de la capasa-8, pero en las que hay una mutación importante en el sitio activo (cisteína a tirosina).
Esta heterodimerización que se produce entre sus DED impide la homodimerización normal, por lo que la pseudocaspasa no puede activar la cascada apoptótica. La pseudocaspasa de FLIP L es más eficiente a la hora de inducir el cambio conformacional. Sin embargo, FLIP L no tiene suficiente actividad enzimática, por lo que no es posible la escisión entre los DED y p18. En consecuencia, es imposible crear la proteasa activa de muerte celular. [10]
La procaspasa-8 también puede heterodimerizarse con FLIP S , que también es una proteína que contiene DED. En este caso, la heterodimerización no activa directamente la procaspasa-8, ya que el cambio conformacional inicial no puede tener lugar en el dominio de caspasa de la procaspasa-8. [10]

Así es como la DED también puede inhibir la cascada de apoptosis, y la consecuencia es la necroptosis .
[11] Las caspasas son cisteína proteasas responsables del desmantelamiento de la célula durante la apoptosis .
Estas proteínas son zimógenos y se activan después de su escisión en sitios específicos dentro de la molécula.
Estructura:
Hay dos grupos de proteasas :
Los dos DED en tándem en el dominio pro de la caspasa inducen las interacciones proteína-proteína con otras proteínas como la FADD .
El estudio de las caspasas es importante porque no sólo controlan la apoptosis, sino que también la inhiben, según la necesidad de la célula. Los científicos han descubierto que son un mecanismo que puede regular la vida celular y es importante para las terapias contra el cáncer.
Las proteínas inhibidoras tipo FLICE (FLIP) son inhibidores celulares capaces de detener la señal de los receptores de muerte, que provocan la apoptosis celular.
Las primeras FLIP que se identificaron fueron expresadas por los virus γ-herpes por lo que se las denominó v-FLIP. Estas v-FLIP eran capaces de asociarse con el receptor en el complejo de señalización inductor de muerte (DISC) , bloqueando de esa manera la apoptosis mediada por CD95.
[12] Los vFLIP contienen predominantemente dos DED secuenciales, que son altamente homólogos al extremo N de la caspasa-8.
[10] Los homólogos celulares de v-FLIP generalmente se expresan en dos formas:
[12] Ambas formas de c-FLIP son reclutadas por el DISC CD95, donde se heterodimerizan con la caspasa-8. c-FLIP ha estado involucrado en vías de señalización alternativas, conectando el receptor CD95 con las vías NF-κB , JNK y MAPK .
PEA-15 (Fosfoproteína Enriquecida en Astrocitos-15 kDa) también conocida como PED (Fosfoproteína Enriquecida en Diabetes) es una proteína que contiene DED con efectos pleiotrópicos .
PED es una proteína pequeña, no catalítica, que consta de un dominio efector de muerte (DED) N-terminal y una cola C-terminal con estructura irregular. [13] PED/PEA-15 interactúa con varios tipos de proteínas con y sin DED, y su especificidad de unirse a estas proteínas está mediada por la fosforilación de dos residuos de serina en la cola C-terminal:
[13] La PEA-15 funciona como una proteína antiapoptótica DED en varias cascadas de señalización . En las vías mediadas por TNF α, CD95 y TRAIL, la PEA-15 actúa uniéndose e interrumpiendo las interacciones de FADD y caspasa-8.
[10] Además de la apoptosis, PEA-15 inhibe el transporte de glucosa mediado por insulina en las células musculares, por lo que una expresión de alto nivel del ARNm de PEA-15 se ha asociado con la diabetes mellitus tipo II .
Dominio efector de muerte que contiene unión al ADN (DEDD). Muestra capacidad de unión al ADN, localizándose en los nucléolos en sobreexpresión donde se asocia a una molécula llamada DEDAF (DED-associated factor) que potencia la apoptosis. Además bloquea la transcripción de la ARN polimerasa I uniéndose al ADN.
DEDD2 (FLAME-3) es un homólogo de DEDD que comparte un 48,5% de la secuencia de aminoácidos. Se ha observado que interactúa con c-FLIP y DEDD y que tiene un papel importante en la represión de la transcripción dependiente de la polimerasa II .
La proteína interactuante con huntingtina-1 (HIP-1) es una proteína que interactúa con la huntingtina (Htt) , otra proteína que cuando está mutada (con repeticiones de poliglutamina expandidas) forma agregados de proteínas en el cerebro de pacientes con enfermedad de Huntington (EH) .
[14] HIP-1 contiene un dominio pseudo efector de muerte (pDED) , por lo que la sobreexpresión de HIP-1 induce apoptosis en varias células al igual que las proteínas DED. Este tipo de apoptosis depende del pDED de HIP-1, y consiste en la activación de la caspasa-3, una enzima que se reduce cuando se expresa Htt de tipo salvaje, lo que sugiere que HIP-1 coopera con Htt en el patomecanismo de la enfermedad de Huntington.
[10] Mediante el cribado de dos híbridos en levaduras , se ha demostrado que HIP-1 interactúa con una proteína de 419 aminoácidos llamada HIPPI (interactuador de la proteína HIP-1). Experimentos posteriores han revelado que la presencia de HIPPI determina la apoptosis inducida por HIP-1 .
Proteína gigante asociada a FLICE. Contiene un dominio similar al de DED, pero la homología es muy débil y su función aún no está clara.
[7] Se ha demostrado que los complejos DED funcionan en etapas cruciales que controlan los procesos de vida y muerte celular. Este conocimiento es particularmente útil en terapia porque existen muchas patologías relacionadas con un control anormal de la vida celular.
La ausencia de apoptosis es una característica del cáncer. En algunos casos el gen que codifica la procaspasa-8 se encuentra silenciado por metilación, por lo que es necesario activar el gen mediante tratamientos epigenéticos para que la proteasa esté activa. En otros casos hay una sobreexpresión de FLIP, la molécula antiapoptótica que impide la formación de la caspasa activa. En este caso existen algunos agentes anticancerígenos que regulan a la baja la expresión de FLIP.
Sin embargo, la apoptosis anormal no es exclusiva del cáncer, existen otras patologías como la inflamación y enfermedades neurodegenerativas que también pueden ser tratadas con este tipo de terapéuticas.