
La dispersión biológica de ángulo pequeño es un método de dispersión de ángulo pequeño para el análisis de la estructura de materiales biológicos. La dispersión de ángulo pequeño se utiliza para estudiar la estructura de una variedad de objetos, como soluciones de macromoléculas biológicas, nanocompuestos, aleaciones y polímeros sintéticos. [1] La dispersión de rayos X de ángulo pequeño ( SAXS ) y la dispersión de neutrones de ángulo pequeño ( SANS ) son las dos técnicas complementarias conocidas conjuntamente como dispersión de ángulo pequeño (SAS). [2] SAS es un método análogo a la difracción de rayos X y neutrones , a la dispersión de rayos X de gran ángulo , así como a la dispersión de luz estática . A diferencia de otros métodos de dispersión de rayos X y neutrones, SAS proporciona información sobre los tamaños y formas de partículas cristalinas y no cristalinas. Cuando se utiliza para estudiar materiales biológicos, que muy a menudo se encuentran en solución acuosa, el patrón de dispersión es un promedio de orientación. [3] [4]
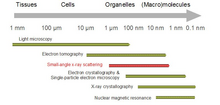
Los patrones SAS se recogen en pequeños ángulos de unos pocos grados. SAS es capaz de entregar información estructural en el rango de resolución entre 1 y 25 nm , y de distancias de repetición en sistemas parcialmente ordenados de hasta 150 nm de tamaño. La dispersión de ángulo ultrapequeño (USAS) puede resolver dimensiones aún mayores. La dispersión de ángulo pequeño de incidencia rasante (GISAS) es una técnica poderosa para estudiar capas de moléculas biológicas en superficies.
En aplicaciones biológicas, SAS se utiliza para determinar la estructura de una partícula en términos de tamaño y forma promedio de partícula. También se puede obtener información sobre la relación superficie - volumen . Normalmente, las macromoléculas biológicas están dispersas en un líquido. El método es preciso, en su mayoría no destructivo y normalmente requiere sólo un mínimo de preparación de la muestra. Sin embargo, las moléculas biológicas siempre son susceptibles al daño por radiación .
En comparación con otros métodos de determinación de estructuras, como la RMN en solución o la cristalografía de rayos X , SAS permite superar algunas restricciones. Por ejemplo, la RMN en solución se limita al tamaño de la proteína, mientras que el SAS se puede utilizar tanto para moléculas pequeñas como para conjuntos multimoleculares grandes. La RMN de estado sólido sigue siendo una herramienta indispensable para determinar la información a nivel atómico de macromoléculas superiores a 40 kDa o muestras no cristalinas como las fibrillas de amiloide. La determinación de la estructura mediante cristalografía de rayos X puede tardar varias semanas o incluso años, mientras que las mediciones de SAS tardan días. SAS también se puede combinar con otras técnicas analíticas como la cromatografía de exclusión por tamaño para estudiar muestras heterogéneas. [5] Sin embargo, con SAS no es posible medir las posiciones de los átomos dentro de la molécula.

Conceptualmente, los experimentos de dispersión de ángulo pequeño son simples: la muestra se expone a rayos X o neutrones y un detector registra la radiación dispersada. Como las mediciones SAS se realizan muy cerca del haz primario ("ángulos pequeños"), la técnica necesita un haz de rayos X o de neutrones altamente colimado o enfocado . La dispersión biológica de rayos X de ángulo pequeño se realiza a menudo en fuentes de radiación de sincrotrón , porque las moléculas biológicas normalmente se dispersan débilmente y las soluciones medidas están diluidas . El método biológico SAXS se beneficia de la alta intensidad de los haces de fotones de rayos X proporcionados por los anillos de almacenamiento de sincrotrón . La curva de dispersión de rayos X o neutrones (intensidad versus ángulo de dispersión) se utiliza para crear un modelo de baja resolución de una proteína, como se muestra aquí en la imagen de la derecha. Además, se pueden utilizar los datos de dispersión de rayos X o neutrones y ajustar dominios separados (estructuras de rayos X o RMN ) en la "envoltura SAXS".
En un experimento de dispersión, una solución de macromoléculas se expone a rayos X (con una longitud de onda λ típicamente de alrededor de 0,15 nm) o neutrones térmicos ( λ ≈0,5 nm). La intensidad dispersada I(s) se registra como una función de la transferencia de momento s ( s=4πsinθ/λ , donde 2θ es el ángulo entre la radiación incidente y la dispersada). De la intensidad de la solución se resta la dispersión procedente únicamente del disolvente. Las posiciones y orientaciones aleatorias de las partículas dan como resultado una distribución de intensidad isotrópica que, para partículas monodispersas que no interactúan, es proporcional a la dispersión de una sola partícula promediada en todas las orientaciones. La dispersión neta de partículas es proporcional a la diferencia al cuadrado en la densidad de longitud de dispersión ( densidad de electrones para los rayos X y densidad nuclear/de espín para los neutrones) entre la partícula y el disolvente: el llamado contraste. El contraste se puede variar en la dispersión de neutrones usando mezclas de H 2 O/ D 2 O o deuteración selectiva para obtener información adicional. [1] El contenido informativo de los datos SAS se ilustra aquí en la figura de la derecha, que muestra patrones de dispersión de rayos X de proteínas con diferentes pliegues y masas moleculares . En ángulos pequeños (resolución de 2-3 nm), las curvas son funciones de s que decaen rápidamente y están determinadas esencialmente por la forma de las partículas, que difieren claramente. A una resolución media (2 a 0,5 nm) las diferencias ya son menos pronunciadas y por encima de una resolución de 0,5 nm todas las curvas son muy similares. [6] Por lo tanto, SAS contiene información sobre las características estructurales generales (forma, estructura cuaternaria y terciaria), pero no es adecuado para el análisis de la estructura atómica.
Las primeras aplicaciones se remontan a finales de la década de 1930, cuando los principios fundamentales de SAXS se desarrollaron en el trabajo fundamental de Guinier tras sus estudios sobre aleaciones metálicas. En la primera monografía sobre SAXS de Guinier y Fournet ya se demostró que el método no sólo proporciona información sobre los tamaños y formas de las partículas, sino también sobre la estructura interna de sistemas desordenados y parcialmente ordenados.
En la década de 1960, el método se volvió cada vez más importante en el estudio de macromoléculas biológicas en solución, ya que permitía obtener información estructural de baja resolución sobre la forma general y la estructura interna en ausencia de cristales. En la década de 1970 se produjo un gran avance en los experimentos SAXS y SANS, gracias a la disponibilidad de radiación sincrotrón y fuentes de neutrones, estas últimas allanaron el camino para la variación del contraste mediante el intercambio de disolventes de H 2 O por D 2 O y métodos de deuteración específicos. Se descubrió que los estudios de dispersión en solución proporcionan, con una mínima inversión de tiempo y esfuerzo, información útil sobre la estructura de los sistemas bioquímicos no cristalinos. Además, SAXS/SANS también hizo posible investigaciones en tiempo real de interacciones intermoleculares, incluido el ensamblaje y cambios conformacionales a gran escala en ensamblajes macromoleculares .
La principal dificultad de SAS como método estructural es extraer la información estructural tridimensional del objeto a partir de los datos experimentales unidimensionales. Hasta ahora, a partir de los datos experimentales sólo se determinaban directamente los parámetros generales de las partículas (p. ej. volumen, radio de giro) de las macromoléculas, mientras que el análisis en términos de modelos tridimensionales se limitaba a cuerpos geométricos simples (p. ej. elipsoides, cilindros, etc.). .) o se realizó mediante prueba y error ad hoc. La microscopía electrónica se utilizó a menudo como limitación en la construcción de modelos de consenso. En la década de 1980, los avances en otros métodos estructurales llevaron a una disminución del interés de los bioquímicos en los estudios SAS, que extraían conclusiones estructurales a partir de sólo un par de parámetros generales o se basaban en modelos de prueba y error.
La década de 1990 trajo un gran avance en los métodos de análisis de datos SAXS/SANS, que abrió el camino para el modelado ab initio confiable de complejos macromoleculares, incluida la determinación detallada de la forma y la estructura de dominio y la aplicación de técnicas de refinamiento de cuerpos rígidos. Este progreso fue acompañado por nuevos avances en la instrumentación, lo que permitió lograr resoluciones de tiempo sub-ms en fuentes SR de tercera generación en los estudios del plegamiento de proteínas y ácidos nucleicos. [1]
En 2005 se inició un proyecto de cuatro años. Iniciativa de dispersión de rayos X de ángulo pequeño para Europa ( SAXIER) con el objetivo de combinar métodos SAXS con otras técnicas analíticas y crear software automatizado para analizar rápidamente grandes cantidades de datos . El proyecto creó una infraestructura SAXS europea unificada, utilizando los métodos más avanzados disponibles. [7]
En un experimento SAS de buena calidad, se miden varias soluciones con diferentes concentraciones de la macromolécula bajo investigación. Al extrapolar las curvas de dispersión medidas en concentraciones variables a una concentración cero, se puede obtener una curva de dispersión que representa una dilución infinita. Entonces los efectos de la concentración no deberían afectar la curva de dispersión. El análisis de datos de la curva de dispersión extrapolada comienza con la inspección del inicio de la curva de dispersión en la región alrededor de s = 0 . Si la región sigue la aproximación de Guinier (también conocida como ley de Guinier ), la muestra no se agrega . Entonces la forma de la partícula en cuestión puede determinarse mediante varios métodos, algunos de los cuales se describen en la siguiente referencia. [1]
El primer paso suele ser calcular una transformada de Fourier de la curva de dispersión. La curva transformada se puede interpretar como una función de distribución de distancia dentro de una partícula. Esta transformación también ofrece el beneficio de regularizar los datos de entrada. [ cita necesaria ]
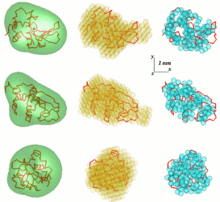
Un problema en el análisis de datos SAS es obtener una estructura tridimensional a partir de un patrón de dispersión unidimensional. Los datos de SAS no implican una solución única. Muchas proteínas diferentes, por ejemplo, pueden tener la misma curva de dispersión. La reconstrucción de la estructura 3D puede dar como resultado una gran cantidad de modelos diferentes. Para evitar este problema es necesario considerar una serie de simplificaciones.
Un método adicional consiste en combinar datos y modelos de dispersión de neutrones y rayos X de ángulo pequeño con el programa MONSA.
En el EMBL se han desarrollado intensamente programas informáticos de análisis SAS de libre acceso . En el primer enfoque general ab initio , una función de envolvente angular de la partícula r=F(ω) , donde ( r,ω ) son coordenadas esféricas, se describe mediante una serie de armónicos esféricos . Por tanto, la forma de baja resolución está definida por unos pocos parámetros (los coeficientes de esta serie) que se ajustan a los datos de dispersión. El enfoque se desarrolló e implementó en el programa informático SASHA ( Small Angle Scattering Shape Determination ). [8] [9] Se demostró que bajo ciertas circunstancias se puede extraer una envoltura única de los datos de dispersión. Este método sólo es aplicable a partículas globulares con formas relativamente simples y sin cavidades internas significativas. Para superar estas limitaciones, se desarrolló otro enfoque que utiliza diferentes tipos de búsquedas Monte-Carlo. DALAI_GA es un programa elegante que toma una esfera con un diámetro igual al tamaño máximo de partícula Dmax, determinado a partir de los datos de dispersión, y la llena con perlas. Cada perla pertenece a la partícula (índice = 1) o al disolvente (índice = 0). La forma se describe así mediante la cadena binaria de longitud M. A partir de una cadena aleatoria, un algoritmo genético busca un modelo que se ajuste a los datos. En la búsqueda, implementada en el programa DAMMIN , se imponen limitaciones de compacidad y conectividad. [10] [11] Si se conoce la simetría de las partículas, SASHA y DAMMIN pueden utilizarla como restricciones útiles. El procedimiento 'dar y recibir' SAXS3D y el programa SASMODEL , basado en elipsoides interconectados, son enfoques ab initio de Monte Carlo sin limitación en el espacio de búsqueda. [6]
Un enfoque que utiliza un conjunto de residuos ficticios (DR) y recocido simulado para construir un modelo DR localmente "compatible con cadena" dentro de una esfera de diámetro Dmax permite extraer más detalles de los datos SAXS. Este método está implementado en el programa GASBOR . [12] [13]
Los patrones de dispersión de soluciones de proteínas multidominio y complejos macromoleculares también se pueden ajustar utilizando modelos construidos a partir de estructuras de alta resolución ( RMN o rayos X ) de dominios o subunidades individuales, suponiendo que se conserva su estructura terciaria . Dependiendo de la complejidad del objeto, se emplean diferentes enfoques para la búsqueda global de la configuración óptima de subunidades que se ajusten a los datos experimentales.
Los modelos basados en Montecarlo contienen cientos o miles de parámetros y se requiere precaución para evitar una sobreinterpretación. Un enfoque común es alinear un conjunto de modelos resultantes de ejecuciones independientes de reconstrucción de formas para obtener un modelo promedio que conserve las características más persistentes y posiblemente también las más confiables (por ejemplo, usando el programa SUPCOMB ). [6] [14] [15]
Los aminoácidos de superficie desordenados (" bucles ") con frecuencia no se observan en los estudios cristalográficos y de RMN , y pueden faltar en los modelos informados. Dichos elementos desordenados contribuyen a la intensidad de la dispersión y sus ubicaciones probables se pueden encontrar arreglando la parte conocida de la estructura y agregando las partes faltantes para que se ajusten al patrón SAS de toda la partícula. Se amplió el enfoque de residuos ficticios y los algoritmos para agregar bucles o dominios faltantes se implementaron en el conjunto de programas CREDO . [6]
Recientemente, se propusieron algunos métodos que utilizan datos SAXS como restricciones. Los autores se propusieron mejorar los resultados de los métodos de reconocimiento de pliegues [16] y de predicción de novo de la estructura de proteínas [17] . Los datos SAXS proporcionan la transformada de Fourier del histograma de distancias de pares atómicos (función de distribución de pares) para una proteína determinada. Esto puede servir como una limitación estructural en los métodos utilizados para determinar el pliegue conformacional nativo de la proteína. El reconocimiento de hilos o pliegues supone que la estructura 3D está más conservada que la secuencia. Por tanto, secuencias muy divergentes pueden tener una estructura similar. Los métodos ab initio, por otra parte, desafían uno de los mayores problemas de la biología molecular: predecir el plegamiento de una proteína "desde cero", sin utilizar secuencias ni estructuras homólogas. Utilizando el "filtro SAXS", los autores pudieron purificar significativamente el conjunto de modelos de proteínas de novo. [17] Esto se demostró además mediante búsquedas de homología de estructuras . También se demostró que la combinación de puntuaciones SAXS con puntuaciones, utilizadas en métodos de subprocesamiento, mejora significativamente el rendimiento del reconocimiento de pliegues. [16] En un ejemplo, se demostró cómo se puede ensamblar una estructura terciaria aproximada de proteínas modulares a partir de estructuras de dominios de RMN de alta resolución, utilizando datos SAXS, limitando los grados de libertad de traducción. [18] Otro ejemplo muestra cómo los datos SAXS se pueden combinar con RMN, cristalografía de rayos X y microscopía electrónica para reconstruir la estructura cuaternaria de una proteína multidominio. [19]
Recientemente se propuso un método elegante para abordar el problema de las proteínas intrínsecamente desordenadas o multidominio con conectores flexibles . [20] Permite la coexistencia de diferentes conformaciones de una proteína, que en conjunto contribuyen al patrón de dispersión experimental promedio. Inicialmente, EOM (método de optimización de conjunto) genera un conjunto de modelos que cubren el espacio de configuración de proteínas. Luego se calcula la curva de dispersión para cada modelo. En el segundo paso, el programa selecciona subconjuntos de modelos de proteínas. La dispersión experimental promedio se calcula para cada subconjunto y se ajusta a los datos experimentales de SAXS. Si no se encuentra el mejor ajuste, los modelos se reorganizan entre diferentes subconjuntos y se realiza un nuevo cálculo de dispersión promedio y ajuste a los datos experimentales. Este método se ha probado en dos proteínas: la lisozima desnaturalizada y la proteína quinasa de Bruton . Dio algunos resultados interesantes y prometedores. [20]
Los recubrimientos de biomoléculas se pueden estudiar mediante rayos X de incidencia rasante y dispersión de neutrones. IsGISAXS ( dispersión de rayos X de ángulo pequeño con incidencia rasante ) es un programa de software dedicado a la simulación y análisis de GISAXS a partir de nanoestructuras. IsGISAXS solo abarca la dispersión de partículas de tamaño nanométrico, que están enterradas en un subsuelo de matriz o soportadas sobre un sustrato o enterradas en una capa delgada sobre un sustrato. También se maneja el caso de los agujeros. La geometría está restringida a un plano de partículas. La sección transversal de dispersión se descompone en términos de función de interferencia y factor de forma de partícula . Se pone énfasis en la geometría de incidencia rasante que induce un "efecto de refracción del haz". El factor de forma de la partícula se calcula dentro de la aproximación de Born de onda distorsionada (DWBA), comenzando como un estado no perturbado con interfaces nítidas o con el perfil perpendicular real del índice de refracción . Se encuentran disponibles varios tipos de formas geométricas simples con una descripción completa de las distribuciones de tamaño y forma en la aproximación de desacoplamiento (DA), en la aproximación monodispersa local (LMA) y también en la aproximación de correlación tamaño-espaciado (SSCA). Se consideran tanto sistemas desordenados de partículas definidos por su función de correlación de pares de partículas como cristales o paracristales bidimensionales. [21]