Una red bayesiana (también conocida como red de Bayes , red de Bayes , red de creencias o red de decisión ) es un modelo gráfico probabilístico que representa un conjunto de variables y sus dependencias condicionales a través de un gráfico acíclico dirigido (DAG). [1] Si bien es una de varias formas de notación causal , las redes causales son casos especiales de redes bayesianas. Las redes bayesianas son ideales para tomar un evento que ocurrió y predecir la probabilidad de que cualquiera de varias posibles causas conocidas fuera el factor contribuyente. Por ejemplo, una red bayesiana podría representar las relaciones probabilísticas entre enfermedades y síntomas. Dados los síntomas, la red se puede utilizar para calcular las probabilidades de la presencia de varias enfermedades.
Los algoritmos eficientes pueden realizar inferencias y aprendizaje en redes bayesianas. Las redes bayesianas que modelan secuencias de variables ( por ejemplo, señales de voz o secuencias de proteínas ) se denominan redes bayesianas dinámicas . Las generalizaciones de redes bayesianas que pueden representar y resolver problemas de decisión en condiciones de incertidumbre se denominan diagramas de influencia .
Formalmente, las redes bayesianas son grafos acíclicos dirigidos (DAG) cuyos nodos representan variables en el sentido bayesiano : pueden ser cantidades observables, variables latentes , parámetros desconocidos o hipótesis. Cada arista representa una dependencia condicional directa. Cualquier par de nodos que no estén conectados (es decir, ningún camino conecta un nodo con el otro) representan variables que son condicionalmente independientes entre sí. Cada nodo está asociado con una función de probabilidad que toma, como entrada, un conjunto particular de valores para las variables padre del nodo , y da (como salida) la probabilidad (o distribución de probabilidad, si corresponde) de la variable representada por el nodo. Por ejemplo, si los nodos padre representan variables booleanas , entonces la función de probabilidad podría representarse mediante una tabla de entradas, una entrada para cada una de las posibles combinaciones de padres. Se pueden aplicar ideas similares a grafos no dirigidos, y posiblemente cíclicos, como las redes de Markov .
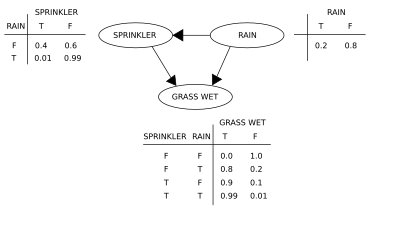
Utilicemos una ilustración para reforzar los conceptos de una red bayesiana. Supongamos que queremos modelar las dependencias entre tres variables: el aspersor (o, más apropiadamente, su estado, si está encendido o no), la presencia o ausencia de lluvia y si el césped está mojado o no. Observe que dos eventos pueden hacer que el césped se moje: un aspersor activo o la lluvia. La lluvia tiene un efecto directo en el uso del aspersor (es decir, cuando llueve, el aspersor generalmente no está activo). Esta situación se puede modelar con una red bayesiana (mostrada a la derecha). Cada variable tiene dos valores posibles, T (para verdadero) y F (para falso).
La función de probabilidad conjunta es, por la regla de la cadena de probabilidad ,
donde G = "Césped mojado (verdadero/falso)", S = "Aspersor encendido (verdadero/falso)" y R = "Lloviendo (verdadero/falso)".
El modelo puede responder preguntas sobre la presencia de una causa dada la presencia de un efecto (la llamada probabilidad inversa) como "¿Cuál es la probabilidad de que esté lloviendo, dado que el pasto está mojado?" utilizando la fórmula de probabilidad condicional y sumando todas las variables molestas :
Utilizando la expansión de la función de probabilidad conjunta y las probabilidades condicionales de las tablas de probabilidad condicional (CPT) indicadas en el diagrama, se puede evaluar cada término en las sumas del numerador y el denominador. Por ejemplo,
Luego, los resultados numéricos (subíndices de los valores de las variables asociadas) son
Para responder a una pregunta de intervención, como "¿Cuál es la probabilidad de que llueva, dado que mojamos el césped?", la respuesta está determinada por la función de distribución conjunta posterior a la intervención.
Se obtiene eliminando el factor de la distribución previa a la intervención. El operador do obliga a que el valor de G sea verdadero. La probabilidad de lluvia no se ve afectada por la acción:
Para predecir el impacto de activar el aspersor:
con el término eliminado, mostrando que la acción afecta a la hierba pero no a la lluvia.
Estas predicciones pueden no ser factibles dadas las variables no observadas, como en la mayoría de los problemas de evaluación de políticas. Sin embargo, el efecto de la acción aún se puede predecir, siempre que se cumpla el criterio de la puerta trasera. [2] [3] Establece que, si se puede observar un conjunto Z de nodos que d-separa [4] (o bloquea) todos los caminos de la puerta trasera de X a Y , entonces
Un camino de puerta trasera es aquel que termina con una flecha en X . Los conjuntos que satisfacen el criterio de puerta trasera se denominan "suficientes" o "admisibles". Por ejemplo, el conjunto Z = R es admisible para predecir el efecto de S = T sobre G , porque R d -separa el (único) camino de puerta trasera S ← R → G . Sin embargo, si no se observa S , ningún otro conjunto d -separa este camino y el efecto de encender el aspersor ( S = T ) sobre el césped ( G ) no se puede predecir a partir de observaciones pasivas. En ese caso, P ( G | do( S = T )) no se "identifica". Esto refleja el hecho de que, a falta de datos de intervención, la dependencia observada entre S y G se debe a una conexión causal o es espuria (dependencia aparente que surge de una causa común, R ). (véase la paradoja de Simpson )
Para determinar si se identifica una relación causal a partir de una red bayesiana arbitraria con variables no observadas, se pueden utilizar las tres reglas del " cálculo do " [2] [5] y probar si todos los términos do se pueden eliminar de la expresión de esa relación, confirmando así que la cantidad deseada es estimable a partir de datos de frecuencia. [6]
El uso de una red bayesiana puede ahorrar una cantidad considerable de memoria en comparación con las tablas de probabilidad exhaustivas, si las dependencias en la distribución conjunta son escasas. Por ejemplo, una forma sencilla de almacenar las probabilidades condicionales de 10 variables de dos valores como una tabla requiere espacio de almacenamiento para los valores. Si la distribución local de ninguna variable depende de más de tres variables principales, la representación de la red bayesiana almacena como máximo valores.
Una ventaja de las redes bayesianas es que es intuitivamente más fácil para un humano comprender (un conjunto disperso de) dependencias directas y distribuciones locales que distribuciones conjuntas completas.
Las redes bayesianas realizan tres tareas de inferencia principales:
Dado que una red bayesiana es un modelo completo de sus variables y sus relaciones, se puede utilizar para responder a preguntas probabilísticas sobre ellas. Por ejemplo, la red se puede utilizar para actualizar el conocimiento del estado de un subconjunto de variables cuando se observan otras variables (las variables de evidencia ). Este proceso de calcular la distribución posterior de las variables dada la evidencia se denomina inferencia probabilística. La distribución posterior proporciona una estadística suficiente universal para aplicaciones de detección, al elegir valores para el subconjunto de variables que minimicen alguna función de pérdida esperada, por ejemplo, la probabilidad de error de decisión. Por lo tanto, una red bayesiana se puede considerar un mecanismo para aplicar automáticamente el teorema de Bayes a problemas complejos.
Los métodos de inferencia exacta más comunes son: eliminación de variables , que elimina (por integración o suma) las variables no observadas que no son de consulta una por una distribuyendo la suma sobre el producto; propagación de árbol de camarillas , que almacena en caché el cálculo de modo que se puedan consultar muchas variables a la vez y se pueda propagar nueva evidencia rápidamente; y condicionamiento recursivo y búsqueda AND/OR, que permiten un equilibrio espacio-tiempo y coinciden con la eficiencia de la eliminación de variables cuando se utiliza suficiente espacio. Todos estos métodos tienen una complejidad que es exponencial en el ancho del árbol de la red. Los algoritmos de inferencia aproximada más comunes son el muestreo de importancia , la simulación MCMC estocástica , la eliminación de mini-bucket, la propagación de creencias en bucle , la propagación de creencias generalizadas y los métodos variacionales .
Para especificar completamente la red bayesiana y, por lo tanto, representar completamente la distribución de probabilidad conjunta , es necesario especificar para cada nodo X la distribución de probabilidad para X condicional a los padres de X. La distribución de X condicional a sus padres puede tener cualquier forma. Es común trabajar con distribuciones discretas o gaussianas ya que eso simplifica los cálculos. A veces solo se conocen las restricciones de la distribución; uno puede entonces usar el principio de máxima entropía para determinar una única distribución, la que tiene la mayor entropía dadas las restricciones. (De manera análoga, en el contexto específico de una red bayesiana dinámica , la distribución condicional para la evolución temporal del estado oculto se especifica comúnmente para maximizar la tasa de entropía del proceso estocástico implícito).
A menudo, estas distribuciones condicionales incluyen parámetros que son desconocidos y deben estimarse a partir de los datos, por ejemplo, mediante el enfoque de máxima verosimilitud . La maximización directa de la verosimilitud (o de la probabilidad posterior ) suele ser compleja dadas las variables no observadas. Un enfoque clásico para este problema es el algoritmo de maximización de expectativas , que alterna el cálculo de los valores esperados de las variables no observadas condicionales a los datos observados, con la maximización de la verosimilitud completa (o posterior) suponiendo que los valores esperados calculados previamente son correctos. En condiciones de regularidad moderada, este proceso converge en valores de máxima verosimilitud (o posterior máxima) para los parámetros.
Un enfoque bayesiano más completo para los parámetros consiste en tratarlos como variables adicionales no observadas y calcular una distribución posterior completa sobre todos los nodos, en función de los datos observados, para luego integrar los parámetros. Este enfoque puede ser costoso y dar lugar a modelos de gran dimensión, lo que hace que los enfoques clásicos de establecimiento de parámetros sean más manejables.
En el caso más simple, un experto especifica una red bayesiana y luego la utiliza para realizar inferencias. En otras aplicaciones, la tarea de definir la red es demasiado compleja para los humanos. En este caso, la estructura de la red y los parámetros de las distribuciones locales deben aprenderse a partir de los datos.
Aprender automáticamente la estructura gráfica de una red bayesiana (BN) es un desafío que se persigue dentro del aprendizaje automático . La idea básica se remonta a un algoritmo de recuperación desarrollado por Rebane y Pearl [7] y se basa en la distinción entre los tres patrones posibles permitidos en un DAG de 3 nodos:
Los primeros 2 representan las mismas dependencias ( y son independientes dados ) y, por lo tanto, son indistinguibles. Sin embargo, el colisionador puede identificarse de forma única, ya que y son marginalmente independientes y todos los demás pares son dependientes. Por lo tanto, si bien los esqueletos (los gráficos despojados de flechas) de estos tres tripletes son idénticos, la direccionalidad de las flechas es parcialmente identificable. La misma distinción se aplica cuando y tienen padres comunes, excepto que primero se deben condicionar esos padres. Se han desarrollado algoritmos para determinar sistemáticamente el esqueleto del gráfico subyacente y, luego, orientar todas las flechas cuya direccionalidad está dictada por las independencias condicionales observadas. [2] [8] [9] [10]
Un método alternativo de aprendizaje estructural utiliza la búsqueda basada en optimización. Requiere una función de puntuación y una estrategia de búsqueda. Una función de puntuación común es la probabilidad posterior de la estructura dados los datos de entrenamiento, como el BIC o el BDeu. El requisito de tiempo de una búsqueda exhaustiva que devuelve una estructura que maximiza la puntuación es superexponencial en el número de variables. Una estrategia de búsqueda local realiza cambios incrementales destinados a mejorar la puntuación de la estructura. Un algoritmo de búsqueda global como el Monte Carlo de cadena de Markov puede evitar quedar atrapado en mínimos locales . Friedman et al. [11] [12] analizan el uso de información mutua entre variables y la búsqueda de una estructura que la maximice. Lo hacen restringiendo el conjunto de candidatos padre a k nodos y buscando exhaustivamente en él.
Un método particularmente rápido para el aprendizaje exacto de BN es plantear el problema como un problema de optimización y resolverlo utilizando programación entera . Las restricciones de aciclicidad se agregan al programa entero (IP) durante la resolución en forma de planos de corte . [13] Este método puede manejar problemas con hasta 100 variables.
Para tratar problemas con miles de variables, es necesario un enfoque diferente. Uno de ellos es muestrear primero un ordenamiento y luego encontrar la estructura de red óptima con respecto a ese ordenamiento. Esto implica trabajar en el espacio de búsqueda de los posibles ordenamientos, lo cual es conveniente ya que es más pequeño que el espacio de las estructuras de red. Luego se muestrean y evalúan múltiples ordenamientos. Se ha demostrado que este método es el mejor disponible en la literatura cuando el número de variables es enorme. [14]
Otro método consiste en centrarse en la subclase de modelos descomponibles, para los cuales los MLE tienen una forma cerrada. De esta manera es posible descubrir una estructura consistente para cientos de variables. [15]
El aprendizaje de redes bayesianas con un ancho de árbol limitado es necesario para permitir una inferencia exacta y manejable, ya que la complejidad de la inferencia en el peor de los casos es exponencial en el ancho de árbol k (según la hipótesis del tiempo exponencial). Sin embargo, como propiedad global del grafo, aumenta considerablemente la dificultad del proceso de aprendizaje. En este contexto, es posible utilizar K-tree para un aprendizaje efectivo. [16]
Dados los datos y parámetros , un análisis bayesiano simple comienza con una probabilidad previa ( prior ) y una probabilidad para calcular una probabilidad posterior .
A menudo, la probabilidad previa depende a su vez de otros parámetros que no se mencionan en la probabilidad. Por lo tanto, la probabilidad previa debe reemplazarse por una probabilidad , y se requiere una probabilidad previa de los parámetros recién introducidos , lo que da como resultado una probabilidad posterior
Este es el ejemplo más simple de un modelo Bayes jerárquico .
El proceso puede repetirse; por ejemplo, los parámetros pueden depender a su vez de parámetros adicionales , que requieren su propio prior. Finalmente, el proceso debe terminar, con priores que no dependan de parámetros no mencionados.
Dadas las cantidades medidas, cada una con errores distribuidos normalmente y con una desviación estándar conocida ,
Supongamos que estamos interesados en estimar el . Un enfoque sería estimar el utilizando un enfoque de máxima verosimilitud ; dado que las observaciones son independientes, la verosimilitud se factoriza y la estimación de máxima verosimilitud es simplemente
Sin embargo, si las cantidades están relacionadas, de modo que, por ejemplo, los individuos mismos han sido extraídos de una distribución subyacente, entonces esta relación destruye la independencia y sugiere un modelo más complejo, por ejemplo,
con valores previos impropios , . Cuando , se trata de un modelo identificado (es decir, existe una solución única para los parámetros del modelo), y las distribuciones posteriores del individuo tenderán a moverse o a encogerse alejándose de las estimaciones de máxima verosimilitud hacia su media común. Esta contracción es un comportamiento típico en los modelos bayesianos jerárquicos.
Se debe tener cuidado al elegir valores a priori en un modelo jerárquico, en particular en el caso de variables de escala en niveles superiores de la jerarquía, como la variable del ejemplo. Los valores a priori habituales, como el de Jeffreys, a menudo no funcionan, porque la distribución posterior no será normalizable y las estimaciones realizadas minimizando la pérdida esperada serán inadmisibles .
Se han propuesto varias definiciones equivalentes de una red bayesiana. Para lo siguiente, sea G = ( V , E ) un grafo acíclico dirigido (DAG) y sea X = ( X v ), v ∈ V un conjunto de variables aleatorias indexadas por V .
X es una red bayesiana con respecto a G si su función de densidad de probabilidad conjunta (con respecto a una medida de producto ) se puede escribir como un producto de las funciones de densidad individuales, condicional a sus variables principales: [17]
donde pa( v ) es el conjunto de padres de v (es decir, aquellos vértices que apuntan directamente a v a través de un solo borde).
Para cualquier conjunto de variables aleatorias, la probabilidad de cualquier miembro de una distribución conjunta se puede calcular a partir de probabilidades condicionales utilizando la regla de la cadena (dado un ordenamiento topológico de X ) de la siguiente manera: [17]
Usando la definición anterior, esto se puede escribir como:
La diferencia entre las dos expresiones es la independencia condicional de las variables de cualquiera de sus no descendientes, dados los valores de sus variables padres.
X es una red bayesiana con respecto a G si satisface la propiedad local de Markov : cada variable es condicionalmente independiente de sus no descendientes dadas sus variables padre: [18]
donde de( v ) es el conjunto de descendientes y V \ de( v ) es el conjunto de no descendientes de v .
Esto se puede expresar en términos similares a la primera definición, como
El conjunto de padres es un subconjunto del conjunto de no descendientes porque el gráfico es acíclico .
En general, se sabe que aprender una red bayesiana a partir de datos es NP-hard . [19] Esto se debe en parte a la explosión combinatoria de enumeración de DAG a medida que aumenta el número de variables. Sin embargo, se pueden obtener conocimientos sobre una red bayesiana subyacente a partir de datos en tiempo polinomial centrándose en su estructura de independencia marginal: [20] mientras que las declaraciones de independencia condicional de una distribución modelada por una red bayesiana están codificadas por un DAG (de acuerdo con la factorización y las propiedades de Markov anteriores), sus declaraciones de independencia marginal (las declaraciones de independencia condicional en las que el conjunto condicionante está vacío) están codificadas por un gráfico simple no dirigido con propiedades especiales como intersección igual y números de independencia .
El desarrollo de una red bayesiana suele comenzar con la creación de un DAG G tal que X satisfaga la propiedad local de Markov con respecto a G . A veces se trata de un DAG causal . Se evalúan las distribuciones de probabilidad condicional de cada variable dadas sus variables parentales en G . En muchos casos, en particular en el caso en que las variables son discretas, si la distribución conjunta de X es el producto de estas distribuciones condicionales, entonces X es una red bayesiana con respecto a G . [21]
La manta de Markov de un nodo es el conjunto de nodos que consiste en sus padres, sus hijos y cualquier otro padre de sus hijos. La manta de Markov hace que el nodo sea independiente del resto de la red; la distribución conjunta de las variables en la manta de Markov de un nodo es conocimiento suficiente para calcular la distribución del nodo. X es una red bayesiana con respecto a G si cada nodo es condicionalmente independiente de todos los demás nodos de la red, dada su manta de Markov . [18]
Esta definición se puede hacer más general definiendo la separación "d" de dos nodos, donde d representa direccional. [2] Primero definimos la separación "d" de un sendero y luego definiremos la separación "d" de dos nodos en términos de eso.
Sea P un camino desde el nodo u hasta v . Un camino es un camino sin bucles y sin dirección (es decir, se ignoran todas las direcciones de los bordes) entre dos nodos. Entonces, se dice que P está d -separado por un conjunto de nodos Z si se cumple alguna de las siguientes condiciones:
Los nodos u y v están separados por d por Z si todos los caminos entre ellos están separados por d . Si u y v no están separados por d, están conectados por d.
X es una red bayesiana con respecto a G si, para cualesquiera dos nodos u , v :
donde Z es un conjunto que d -separa u y v . (La manta de Markov es el conjunto mínimo de nodos que d -separa el nodo v de todos los demás nodos).
Aunque las redes bayesianas se utilizan a menudo para representar relaciones causales , esto no tiene por qué ser así: una arista dirigida de u a v no requiere que X v sea causalmente dependiente de X u . Esto se demuestra por el hecho de que las redes bayesianas en los grafos:
son equivalentes: es decir, imponen exactamente los mismos requisitos de independencia condicional.
Una red causal es una red bayesiana con el requisito de que las relaciones sean causales. La semántica adicional de las redes causales especifica que si se provoca activamente que un nodo X se encuentre en un estado dado x (una acción escrita como do( X = x )), entonces la función de densidad de probabilidad cambia a la de la red obtenida al cortar los vínculos de los padres de X a X , y establecer X en el valor causado x . [2] Usando esta semántica, se puede predecir el impacto de las intervenciones externas a partir de los datos obtenidos antes de la intervención.
En 1990, mientras trabajaba en la Universidad de Stanford en grandes aplicaciones bioinformáticas, Cooper demostró que la inferencia exacta en redes bayesianas es NP-hard . [22] Este resultado impulsó la investigación sobre algoritmos de aproximación con el objetivo de desarrollar una aproximación manejable a la inferencia probabilística. En 1993, Paul Dagum y Michael Luby demostraron dos resultados sorprendentes sobre la complejidad de la aproximación de la inferencia probabilística en redes bayesianas. [23] Primero, demostraron que ningún algoritmo determinista manejable puede aproximar la inferencia probabilística dentro de un error absoluto ɛ < 1/2. Segundo, demostraron que ningún algoritmo aleatorio manejable puede aproximar la inferencia probabilística dentro de un error absoluto ɛ < 1/2 con una probabilidad de confianza mayor que 1/2.
Casi al mismo tiempo, Roth demostró que la inferencia exacta en redes bayesianas es de hecho #P-completa (y por lo tanto tan difícil como contar el número de asignaciones satisfactorias de una fórmula de forma normal conjuntiva (CNF)) y que la inferencia aproximada dentro de un factor 2 n 1− ɛ para cada ɛ > 0, incluso para redes bayesianas con arquitectura restringida, es NP-difícil. [24] [25]
En términos prácticos, estos resultados de complejidad sugirieron que, si bien las redes bayesianas eran representaciones ricas para aplicaciones de IA y aprendizaje automático, su uso en grandes aplicaciones del mundo real necesitaría ser moderado por restricciones estructurales topológicas, como redes Bayesianas ingenuas, o por restricciones en las probabilidades condicionales. El algoritmo de varianza acotada [26] desarrollado por Dagum y Luby fue el primer algoritmo de aproximación rápida demostrable para aproximar eficientemente la inferencia probabilística en redes bayesianas con garantías en la aproximación del error. Este poderoso algoritmo requería que la restricción menor en las probabilidades condicionales de la red bayesiana se alejara de cero y uno por donde era cualquier polinomio del número de nodos en la red, .
Entre los programas notables para redes bayesianas se incluyen:
El término red bayesiana fue acuñado por Judea Pearl en 1985 para enfatizar: [28]
A finales de la década de 1980, el Razonamiento Probabilístico en Sistemas Inteligentes de Pearl [30] y el Razonamiento Probabilístico en Sistemas Expertos de Napolitan [31] resumieron sus propiedades y las establecieron como un campo de estudio.
{{cite book}}: CS1 maint: location missing publisher (link){{cite book}}: CS1 maint: bot: original URL status unknown (link):También aparece como Heckerman, David (marzo de 1997). "Bayesian Networks for Data Mining". Minería de datos y descubrimiento de conocimiento . 1 (1): 79–119. doi :10.1023/A:1009730122752. S2CID 6294315.