La activación transcripcional controlada por tetraciclina es un método de expresión genética inducible donde la transcripción se activa o desactiva de forma reversible en presencia del antibiótico tetraciclina o uno de sus derivados (por ejemplo, doxiciclina ). [1]
La expresión génica controlada por la tetraciclina se basa en el mecanismo de resistencia al tratamiento con antibióticos de tetraciclina que se encuentra en las bacterias gramnegativas . En la naturaleza, el promotor P tet expresa TetR (el represor ) y TetA, la proteína que bombea el antibiótico de tetraciclina fuera de la célula. [2]
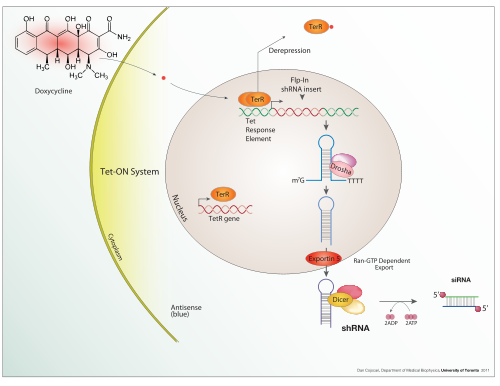
La diferencia entre Tet-On y Tet-Off no es si el transactivador activa o desactiva un gen, como su nombre podría sugerir, sino que ambas proteínas activan la expresión. La diferencia se relaciona con su respectiva respuesta a la tetraciclina o la doxiciclina (Dox, un análogo de la tetraciclina más estable); Tet-Off activa la expresión en ausencia de Dox, mientras que Tet-On se activa en presencia de Dox.
Los dos sistemas de expresión inducible más utilizados para la investigación de la biología de células eucariotas se denominan Tet-Off y Tet-On. [3] El sistema Tet-Off para controlar la expresión de genes de interés en células de mamíferos fue desarrollado por los profesores Hermann Bujard y Manfred Gossen en la Universidad de Heidelberg y publicado por primera vez en 1992. [4]
El sistema Tet-Off utiliza la proteína transactivadora de tetraciclina (tTA) , que se crea fusionando una proteína , TetR (represor de tetraciclina), que se encuentra en la bacteria Escherichia coli , con el dominio de activación de otra proteína, VP16 , que se encuentra en el virus del herpes simple . [5]
La proteína tTA resultante es capaz de unirse al ADN en secuencias operadoras TetO específicas . En la mayoría de los sistemas Tet-Off, varias repeticiones de dichas secuencias TetO se colocan aguas arriba de un promotor mínimo, como el promotor CMV. La totalidad de varias secuencias TetO con un promotor mínimo se denomina elemento de respuesta a la tetraciclina (TRE), porque responde a la unión de la proteína transactivadora de tetraciclina tTA mediante el aumento de la expresión del gen o genes aguas abajo de su promotor.
En un sistema Tet-Off, la expresión de genes controlados por TRE puede ser reprimida por la tetraciclina y sus derivados. Estos se unen al tTA y lo vuelven incapaz de unirse a las secuencias de TRE, impidiendo así la transactivación de los genes controlados por TRE.
Un sistema Tet-On funciona de manera similar, pero de manera opuesta. Mientras que en un sistema Tet-Off, el tTA es capaz de unirse al operador solo si no está unido a la tetraciclina o a uno de sus derivados, como la doxiciclina, en un sistema Tet-On, la proteína rtTA es capaz de unirse al operador solo si está unida a una tetraciclina. Por lo tanto, la introducción de doxiciclina en el sistema inicia la transcripción del producto genético. El sistema Tet-On a veces se prefiere al Tet-Off por su capacidad de respuesta más rápida.
Los sistemas de expresión Tet-Off también se utilizan para generar ratones transgénicos que expresan condicionalmente el gen de interés.
El transactivador Tet-On Advanced (también conocido como rtTA2 S -M2) es una versión alternativa de Tet-On que muestra una expresión basal reducida y funciona a una concentración de Dox 10 veces menor que Tet-Off. Además, se considera que su expresión es más estable en células eucariotas debido a que está optimizada para codones humanos y utiliza tres dominios de activación transcripcional mínimos. Fue descubierto en 2000 como uno de los dos mutantes mejorados por H. Bujard y sus colegas después de una mutagénesis aleatoria de la parte represora Tet del gen transactivador. [6] Tet-On 3G (también conocido como rtTA-V10 [7] ) es similar a Tet-On Advanced pero se derivó de rtTA2 S -S2 en lugar de rtTA2 S -M2. También está optimizado para codones humanos y está compuesto por tres dominios de activación VP16 mínimos. Sin embargo, la proteína Tet-On 3G tiene cinco diferencias de aminoácidos en comparación con Tet-On Advanced que parecen aumentar aún más su sensibilidad a Dox. Tet-On 3G es sensible a 100 veces menos Dox y es siete veces más activo que el Tet-On original. [8]
Otros sistemas, como el sistema T-REx de Life Technologies, funcionan de una manera diferente. [9] El gen de interés está flanqueado por un promotor CMV aguas arriba y dos sitios TetO2. La expresión del gen de interés se reprime por la unión de alta afinidad de los homodímeros TetR a cada secuencia TetO2 en ausencia de tetraciclina. La introducción de tetraciclina da como resultado la unión de una tetraciclina en cada homodímero TetR seguida de la liberación de TetO2 por los homodímeros TetR. La desunión de los homodímeros TetR y TetO2 da como resultado la desrepresión del gen de interés.
Una versión modificada de T-REx es el circuito biológico sintético Linearizer , optimizado para ajustar la expresión génica en células eucariotas (levadura en ciernes, humanas, etc.). Al incorporar sitios TetO2 en el promotor que impulsa la expresión de TetR, crea una retroalimentación negativa , que garantiza una expresión homogénea (bajo nivel de ruido) y una respuesta lineal a la dosis de los análogos de tetraciclina. [10]
En los plásmidos más utilizados , el elemento de respuesta a la tetraciclina consta de siete repeticiones de la secuencia bacteriana TetO de 19 pb (TCCCTATCAGTGATAGAGA) separadas por secuencias espaciadoras (por ejemplo: ACGATGTCGAGTTTAC). Es el TetO el que es reconocido y unido por la porción TetR de Tet-On o Tet-Off. El TRE generalmente se coloca aguas arriba de un promotor mínimo que tiene una expresión basal muy baja en ausencia de Tet-Off (o Tet-On) unido.
El sistema Tet tiene ventajas sobre los sistemas de expresión génica condicional Cre , FRT y ER (receptor de estrógeno). En los sistemas Cre y FRT, la activación o inactivación del gen es irreversible una vez que se logra la recombinación, mientras que, en los sistemas Tet y ER, es reversible. El sistema Tet tiene un control muy estricto sobre la expresión, mientras que el sistema ER es algo permeable. [11] Sin embargo, el sistema Tet, que depende de la transcripción y la posterior traducción de un gen diana, no actúa tan rápido como el sistema ER, que estabiliza la proteína diana ya expresada tras la administración de hormonas. Además, dado que la secuencia tet-o de 19 pb está naturalmente ausente de las células de mamíferos, se cree que la pleiotropía se minimiza en comparación con los métodos hormonales de control. Cuando se utiliza el sistema Tet en un cultivo celular, es importante confirmar que cada lote de suero bovino fetal se prueba para confirmar que las tetraciclinas contaminantes están ausentes o son demasiado bajas para interferir con la inducibilidad.
El mecanismo de acción del efecto antibacteriano de las tetraciclinas se basa en alterar la traducción de proteínas en las bacterias, dañando así la capacidad de los microbios de crecer y repararse; sin embargo, la traducción de proteínas también se altera en las mitocondrias eucariotas, lo que produce efectos que pueden confundir los resultados experimentales. [12]