La ribozima del virus de la hepatitis delta (HDV) es un ARN no codificante que se encuentra en el virus de la hepatitis delta y que es necesario para la replicación viral y es el único virus humano conocido que utiliza la actividad de la ribozima para infectar a su huésped. [1] La ribozima actúa procesando las transcripciones de ARN a longitudes unitarias en una reacción de autoescisión durante la replicación del virus de la hepatitis delta, que se cree que se propaga mediante un mecanismo de doble círculo rodante. [2] [3] La ribozima es activa in vivo en ausencia de cualquier factor proteico y era el ARN de autoescisión natural más rápido conocido en el momento de su descubrimiento. [4]
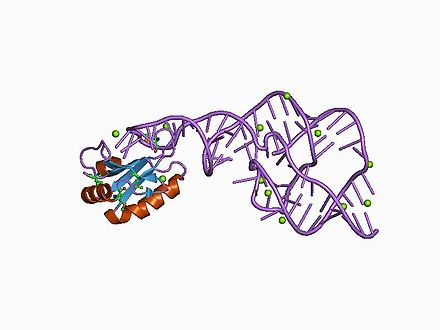
La estructura cristalina de esta ribozima se ha resuelto mediante cristalografía de rayos X y muestra cinco segmentos helicoidales conectados por un doble pseudonudo . [1]
Además del sentido (versión genómica), todos los virus HDV también tienen una versión antigenómica de la ribozima HDV. [5] Esta versión no es la secuencia complementaria exacta, pero adopta la misma estructura que la cadena sentido (genómica). Las únicas diferencias "significativas" entre los dos son un pequeño bulto en el vástago P4 y una unión J4/2 más corta. Tanto las ribozimas genómicas como las antigenómicas son necesarias para la replicación. [2]
La ribozima HDV está estructural y bioquímicamente relacionada con muchas otras ribozimas autoescindibles. Estas otras ribozimas a menudo se denominan ejemplos de ribozimas HDV debido a estas similitudes, aunque no se encuentran en los virus de la hepatitis delta. También se les puede denominar "similares a HDV" para indicar este hecho. [6]
Las ribozimas similares a HDV incluyen la ribozima CPEB3 de mamíferos , ribozimas theta en bacteriófagos, miembros de retrotransposones (por ejemplo, en el elemento R2 de ARN en insectos y en L1Tc y probablemente otros retrotransposones en tripanosomátidos) y secuencias de bacterias. [7] [8] [6] [9] [10] [11] La agrupación es probablemente el resultado de una evolución convergente : los deltavirus que se encuentran fuera de los humanos también poseen una ribozima DV, y ningún escenario de transferencia horizontal de genes propuesto todavía puede explicar esto. . [12] [13] [14]
La ribozima HDV cataliza la escisión del enlace fosfodiéster entre el nucleótido u oligonucleótido sustrato y el 5'-hidroxilo de la ribozima. En el virus de la hepatitis delta, esta secuencia de nucleótidos sustrato comienza con uridina y se conoce como U(-1); sin embargo, la identidad del nucleótido -1 no cambia significativamente la velocidad de catálisis. [1] Sólo existe un requisito por su naturaleza química, ya que, como lo muestran Perrotta y Been, la sustitución de la ribosa U(-1) por desoxirribosa suprime la reacción, lo que es consistente con la predicción de que el 2′-hidroxilo es el nucleófilo en la reacción química. [15] Por lo tanto, a diferencia de muchas otras ribozimas, como la ribozima cabeza de martillo , la ribozima HDV no tiene requisitos iniciales para la catálisis y requiere solo un único ribonucleótido -1 como sustrato para reaccionar eficientemente. [1]
Inicialmente, se creía que el nucleótido 75 de la ribozima, una citosina conocida como C75, era capaz de actuar como base general con el N3 de C75 extrayendo un protón del 2′-hidroxilo del nucleótido U(-1) para Facilitar el ataque nucleofílico al enlace fosfodiéster. [1] Sin embargo, aunque está bien establecido que el N3 de C75 tiene un pKa perturbado de su valor normal de 4,45 y está más cerca de aproximadamente 6,15 o 6,40, [16] [17] no es lo suficientemente neutral como para actuar como un catalizador básico. En cambio, se cree que el N3 del C75 actúa como un ácido de Lewis para estabilizar el 5'-hidroxilo saliente de la ribozima; esto está respaldado por su proximidad al 5′-hidroxilo en la estructura cristalina. [1] [18] La sustitución del nucleótido C75 por cualquier otro nucleótido suprime o perjudica sustancialmente la actividad de la ribozima, aunque esta actividad puede restaurarse parcialmente con imidazol, lo que implica aún más al C75 en la actividad catalítica. [19]
El C75 de la ribozima HDV ha sido objeto de varios estudios debido a su peculiar pKa. Los valores de pKa típicos para los nucleósidos libres oscilan entre 3,5 y 4,2; Estos valores de pKa más bajos son ácidos y es poco probable que se vuelvan básicos. Sin embargo, es probable que el entorno estructural dentro de la ribozima, que incluye una hendidura del sitio activo desolvatado, proporcione un potencial electrostático negativo que podría perturbar el pKa de la citosina lo suficiente como para actuar como un ácido de Lewis. [20] [21] [22] [23]

Además de la estabilización con ácido de Lewis del grupo saliente 5′-hidroxilo, ahora también se acepta que la ribozima HDV puede utilizar un ion metálico para ayudar en la activación del 2′-hidroxilo para atacar el nucleótido U(-1). Un ion magnesio en el sitio activo de la ribozima está coordinado con el nucleófilo 2'-hidroxilo y un oxígeno del fosfato escindible, y puede actuar como un ácido de Lewis para activar el 2'-hidroxilo. [19] [18] Además, es posible que el fosfato de U23 pueda actuar como un ácido de Lewis para aceptar un protón del 2′-hidroxilo con el magnesio sirviendo como ion coordinador. [24] Debido a que la ribozima HDV no requiere iones metálicos para tener actividad, no es una metaloenzima obligada, pero la presencia de magnesio en el sitio activo mejora significativamente la reacción de escisión. La ribozima HDV parece tener un requisito no específico para que se plieguen cantidades bajas de cationes divalentes, siendo activa en Mg 2+ , Ca 2+ , Mn 2+ y Sr 2+ . [1] En ausencia de iones metálicos, parece probable que el agua pueda reemplazar el papel del magnesio como ácido de Lewis.
Como está limitado por la rápida naturaleza de autoescisión de la ribozima HDV, los experimentos anteriores con ribonucleasa se realizaron en el producto 3 'de la autoescisión en lugar del precursor. [25] Sin embargo, se sabe que la secuencia flanqueante participa en la regulación de la actividad de autoescisión de la ribozima HDV. [26] [27] [28] Por lo tanto, la secuencia aguas arriba 5' del sitio de autoescisión se ha incorporado para estudiar la actividad de autoescisión resultante de la ribozima HDV. [26] Se han identificado dos estructuras alternativas.
La primera estructura inhibidora está plegada por una transcripción extendida (es decir, transcripción -30/99, las coordenadas se refieren al sitio de autoescisión) que se extiende desde 30 nt aguas arriba del sitio de escisión hasta 15 nt aguas abajo del extremo 3'. [26] La secuencia flanqueante secuestra la ribozima en una trampa cinética durante la transcripción y da como resultado una tasa de autoescisión extremadamente disminuida. [26] Esta estructura que previene la autoescisión incluye 3 tallos alternativos: Alt1, Alt2 y Alt3, que interrumpen la conformación activa. Alt1 es una interacción de largo alcance de 10 pb formada por un tramo inhibidor aguas arriba (-25/-15 nt) y el tramo aguas abajo (76/86 nt). [26] Alt1 altera la raíz P2 en la conformación activa en la que se propone que P2 tenga un papel activador tanto para la ribozima genómica como antigenómica. [26] [29] [30] Alt2 es una interacción entre la secuencia flanqueante aguas arriba y la ribozima, y Alt3 es una interacción ribozima-ribozima no nativa. [26]
La estructura secundaria de esta conformación inhibidora está respaldada por varios enfoques experimentales. [26] Primero, se realizó un sondeo directo mediante ribonucleasas y el modelado posterior mediante mfold 3.0 utilizando restricciones de los resultados del sondeo concuerda con la estructura propuesta. [26] En segundo lugar, se utilizó una serie de oligómeros de ADN complementarios a diferentes regiones de AS1/2 para rescatar la actividad de la ribozima; los resultados confirman las funciones inhibidoras de AS1/2. [26] En tercer lugar, el análisis mutacional introduce mutaciones simples/dobles fuera de la ribozima para garantizar que la actividad de la ribozima observada esté directamente asociada con la estabilidad de Alt1. [26] Se encuentra que la estabilidad de AS1 está inversamente relacionada con la actividad de autoescisión. [26]
La segunda estructura permisiva permite que la ribozima HDV se autoescinda cotranscripcionalmente y esta estructura incluye además la porción -54/-18 nt del transcrito de ARN. [26] El tramo inhibidor -24/-15 aguas arriba de la conformación inhibidora antes mencionada ahora está secuestrado en una horquilla P(-1) ubicada aguas arriba del sitio de escisión. [26] [31] [32] Sin embargo, el motivo P(-1) solo se encuentra en la secuencia genómica, lo que puede estar correlacionado con el fenómeno de que las copias genómicas del ARN del VHD son más abundantes en las células hepáticas infectadas. [26] [5] La evidencia experimental también respalda esta estructura alternativa. En primer lugar, se utiliza el mapeo estructural mediante ribonucleasa para sondear el fragmento -54/-1 en lugar del transcrito precursor completo debido a la naturaleza de escisión rápida de esta estructura, que concuerda con la horquilla local P(-1) (entre -54/ -40 y -18/-30 nt). [26] En segundo lugar, la conservación evolutiva se encuentra en P(-1) y la región de enlace entre P(-1) y P1 entre 21 aislados genómicos de ARN del VHD. [26]
Las propiedades especiales de la reacción de escisión de la ribozima HDV la convierten en una herramienta útil para preparar transcripciones de ARN con extremos 3' homogéneos, una alternativa a la transcripción de ARN con ARN polimerasa T7 que a menudo puede producir extremos heterogéneos o adiciones no deseadas. La versión de ADNc de la ribozima puede prepararse adyacente al ADNc de la secuencia de ARN diana y al ARN preparado a partir de la transcripción con la ARN polimerasa T7. La secuencia de ribozima se escindirá de manera eficiente sin requisitos posteriores, ya que el nucleótido -1 es invariante, dejando un fosfato cíclico 2′-3′ que puede eliminarse fácilmente mediante tratamiento con una fosfatasa o polinucleótido quinasa T4. [33] El ARN diana luego se puede purificar con purificación en gel.