
La desviación estándar de GC se produce cuando los nucleótidos guanina y citosina son sobreabundantes o subabundantes en una región particular de ADN o ARN . La desviación estándar de GC es también un método estadístico para medir la sobrerrepresentación de guanina en cadenas específicas. [1]
En condiciones de equilibrio (sin presión mutacional o selectiva y con nucleótidos distribuidos aleatoriamente dentro del genoma ) hay una frecuencia igual de las cuatro bases de ADN ( adenina , guanina , timina y citosina ) en ambas hebras simples de una molécula de ADN. [2] Sin embargo, en la mayoría de las bacterias (por ejemplo, E. coli ) y algunas arqueas (por ejemplo, Sulfolobus solfataricus ), las composiciones de nucleótidos son asimétricas entre la hebra líder y la hebra rezagada : la hebra líder contiene más guanina (G) y timina (T), mientras que la hebra rezagada contiene más adenina (A) y citosina (C). [2] Este fenómeno se conoce como sesgo GC y AT y las estadísticas correspondientes se definieron [2] como:
Desviación de GC = (G - C)/(G + C)
AT sesgado = (A − T)/(A + T)
El trabajo de Erwin Chargaff en 1950 demostró que, en el ADN, las bases guanina y citosina se encontraban en igual abundancia, y las bases adenina y timina se encontraban en igual abundancia. Sin embargo, no había igualdad entre la cantidad de un par en comparación con el otro. [3] El hallazgo de Chargaff se conoce como regla de Chargaff o regla de paridad 2 . [3] Tres años después, Watson y Crick utilizaron este hecho durante su derivación de la estructura del ADN, su modelo de doble hélice .
Un resultado natural de la regla de paridad 1, en el estado de equilibrio, en el que no hay sesgos de mutación y/o selección en ninguna de las dos cadenas de ADN, es que cuando hay una tasa de sustitución igual, los nucleótidos complementarios en cada cadena tienen cantidades iguales de una base dada y su complemento. [4] En otras palabras, en cada cadena de ADN la frecuencia de aparición de T es igual a A y la frecuencia de aparición de G es igual a C porque la tasa de sustitución es presumiblemente igual. Este fenómeno se conoce como regla de paridad 2. Por lo tanto, la segunda regla de paridad solo existe cuando no hay mutación o sustitución.
Cualquier desviación de la regla de paridad 2 dará como resultado una composición de bases asimétrica que discrimina la cadena líder (es decir, la cadena de ADN que se replica en la dirección hacia adelante) de la cadena rezagada. Esta asimetría se conoce como sesgo GC o AT. [2]
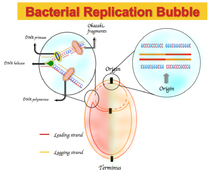
En algunos genomas bacterianos, hay un enriquecimiento de guanina sobre citosina y timina sobre adenina en la hebra líder y viceversa para la hebra rezagada. Los espectros de sesgo de la composición de nucleótidos varían de −1, que corresponde a G = 0 o A = 0, a +1, que corresponde a T = 0 o C = 0. [2] Por lo tanto, el sesgo GC positivo representa la riqueza de G sobre C y el sesgo GC negativo representa la riqueza de C sobre G. Como resultado, uno espera ver un sesgo GC positivo y un sesgo AT negativo en la hebra líder, y un sesgo GC negativo y un sesgo AT positivo en la hebra rezagada. [5] El sesgo GC o AT cambia de signo en los límites de los dos replicores , que corresponden al origen o término de la replicación del ADN. [2] [4] [5] Originalmente, esta composición asimétrica de nucleótidos se explicó como un mecanismo diferente utilizado en la replicación del ADN entre la cadena líder y la cadena rezagada. La replicación del ADN es semiconservativa y un proceso asimétrico en sí mismo. [6] Esta asimetría se debe a la formación de la horquilla de replicación y su división en cadenas líderes y rezagadas nacientes. La cadena líder se sintetiza de forma continua y yuxtapuesta a la cadena líder; la cadena rezagada se replica a través de fragmentos cortos de polinucleótido ( fragmentos de Okazaki ) en una dirección de 5' a 3'. [6]
Hay tres enfoques principales para calcular y demostrar gráficamente la desviación estándar del GC y sus propiedades.
La primera aproximación es la asimetría GC y AT. [2] Jean R. Lobry fue el primero en reportar, en 1996, [7] la presencia de asimetría compositiva en los genomas de tres bacterias: E. coli , Bacillus subtilis y Haemophilus influenzae . Las fórmulas originales en ese momento no se llamaban skew, sino desviación de [A] = [T] o [C] = [G]:
desviación de [A] = [T] como (A − T)/(A + T);
desviación de [C] = [G] como (C − G)/(C + G);
donde A, T, G y C representan la frecuencia de aparición de la base equivalente en una secuencia particular en una longitud definida. Se utiliza una estrategia de deslizamiento de ventana para calcular la desviación de C a través del genoma. En estos gráficos, una desviación positiva de C corresponde a la cadena rezagada y una desviación negativa de C corresponde a la cadena líder. [8] Además, el sitio donde cambia el signo de desviación corresponde al origen o terminal. El eje x representa las ubicaciones de los cromosomas trazadas de 5′ a 3′ y el eje y representa el valor de desviación. La principal debilidad de este método es su propiedad dependiente del tamaño de la ventana. Por lo tanto, la elección de un tamaño de ventana adecuado afecta en gran medida el resultado del gráfico. Se deben combinar otras técnicas con la desviación para identificar y localizar el origen de la replicación del ADN con mayor precisión.

El segundo enfoque se conoce como sesgo de GC acumulativo (sesgo de CGC). [9] Este método todavía utiliza la estrategia de ventana deslizante, pero aprovecha la suma de las ventanas adyacentes a partir de un inicio arbitrario. En este esquema, el genoma completo suele representarse gráficamente de 5' a 3' utilizando un inicio y una cadena arbitrarios. En el gráfico de sesgo de GC acumulativo, los picos corresponden a los puntos de conmutación (extremo u origen).
A diferencia del artículo anterior de Lobry, las implementaciones recientes de GC invierten la definición original y la redefinen como:
Sesgo GC = (G − C)/(G + C).
Con la definición invertida del sesgo GC, el valor máximo del sesgo acumulativo corresponde a la terminal y el valor mínimo corresponde al origen de la replicación.
El enfoque final es la curva Z. [ 10] A diferencia de los métodos anteriores, este método no utiliza la estrategia de ventana deslizante y se cree que tiene un mejor rendimiento en cuanto a la búsqueda del origen de replicación. [10] En este método, se investiga la frecuencia acumulada de cada base con respecto a la base al comienzo de la secuencia. La curva Z utiliza una representación tridimensional con los siguientes parámetros:
Donde , representa el exceso de purina sobre pirimidina, denota exceso de ceto sobre amino y muestra la relación entre los enlaces de hidrógeno débiles y fuertes . y los componentes pueden detectar por sí solos el origen de replicación y la composición asimétrica de las cadenas. Se debe utilizar una combinación de estos métodos para predecir el origen y la terminal de replicación, a fin de compensar su debilidad.
En la comunidad científica no hay consenso sobre el mecanismo que subyace al sesgo en la composición de nucleótidos dentro de cada cadena de ADN. Hay dos escuelas de pensamiento principales que explican el mecanismo detrás de la composición de nucleótidos específica de la cadena en las bacterias. [4]
El primero describe un sesgo y una presión mutacional asimétrica en cada cadena de ADN durante la replicación y la transcripción . [4] [11] Debido a la naturaleza asimétrica del proceso de replicación, una frecuencia mutacional desigual y una eficiencia de reparación del ADN durante el proceso de replicación pueden introducir más mutaciones en una cadena en comparación con la otra. [5] Además, el tiempo utilizado para la replicación entre las dos cadenas varía y puede conducir a una presión mutacional asimétrica entre la cadena líder y la rezagada. [12] Además de las mutaciones durante la replicación del ADN, las mutaciones transcripcionales pueden crear un sesgo en la composición de nucleótidos específica de la cadena. [5] La desaminación de la citosina y, en última instancia, la mutación de la citosina a timina en una cadena de ADN puede aumentar el número relativo de guanina y timina a citosina y adenina. [5] En la mayoría de las bacterias, la mayoría de los genes están codificados en la cadena líder. [4] Por ejemplo, la cadena líder en Bacillus subtilis codifica el 75% de los genes. [5] Además, se ha informado de un exceso de desaminación y conversión de citosina a timina en la cadena codificante en comparación con la cadena no codificante. [4] [5] [13] Una posible explicación es que la cadena no transcrita ( cadena codificante ) es monocatenaria durante el proceso de transcripción; por lo tanto, es más vulnerable a la desaminación en comparación con la cadena transcrita ( cadena no codificante ). [5] [14] Otra explicación es que la actividad de reparación de la deaminación durante la transcripción no ocurre en la cadena codificante. [5] Solo la cadena transcrita se beneficia de estos eventos de reparación de la deaminación.
La segunda escuela de pensamiento describe el mecanismo de sesgo de GC y AT como resultado de una diferencia en la presión selectiva entre las cadenas líder y rezagada. [4] [5] [14] El examen del genoma procariota muestra una preferencia en la posición del tercer codón para G sobre C y T sobre A. [5] Esta discriminación crea una composición de nucleótidos asimétrica, si la cadena codificante está distribuida de manera desigual entre las cadenas líder y rezagada, como en el caso de las bacterias. Además, se ha demostrado que los genes altamente transcritos, como las proteínas ribosómicas , se ubican principalmente en la cadena líder en bacterias. [5] Por lo tanto, un sesgo en la elección del codón de tercera posición de G sobre C puede conducir a un sesgo de GC. Además, algunas secuencias de señal son ricas en guanina y timina, como las secuencias chi , y estas secuencias podrían tener una mayor frecuencia de aparición en una cadena en comparación con la otra. [4] [5]
Tanto la presión mutacional como la selectiva pueden introducir asimetría en las cadenas de ADN de forma independiente. Sin embargo, la combinación y el efecto acumulativo de ambos mecanismos es la explicación más plausible para la asimetría de GC y AT. [4] [14]
Se ha demostrado que el sesgo GC es útil como indicador de la cadena líder de ADN, la cadena rezagada, el origen de replicación y la terminal de replicación. [2] [4] [5] La mayoría de las bacterias y arqueas contienen solo un origen de replicación de ADN. [2] El sesgo GC es positivo y negativo en la cadena líder y en la cadena rezagada respectivamente; por lo tanto, se espera ver un cambio en el signo del sesgo GC justo en el punto de origen y terminal de replicación de ADN. [4] El sesgo GC también se puede utilizar para estudiar los sesgos de la cadena y el mecanismo relacionado con ellos calculando el exceso de una base sobre su base complementaria en diferentes medios. [4] [5] [14] Métodos como el sesgo GC, el sesgo CGC y la curva Z son herramientas que pueden brindar la oportunidad de investigar mejor el mecanismo de replicación de ADN en diferentes organismos.