La notación de ácidos nucleicos actualmente en uso fue formalizada por primera vez por la Unión Internacional de Química Pura y Aplicada (IUPAC) en 1970. [1] Esta notación universalmente aceptada utiliza los caracteres romanos G, C, A y T para representar los cuatro nucleótidos que se encuentran comúnmente en los ácidos desoxirribonucleicos (ADN).
Dada la rápida expansión del papel de la secuenciación, síntesis y análisis genéticos en biología, algunos investigadores han desarrollado notaciones alternativas para respaldar aún más el análisis y la manipulación de datos genéticos. Estas notaciones generalmente aprovechan el tamaño, la forma y la simetría para lograr estos objetivos.
Los símbolos de bases degeneradas en bioquímica son una representación IUPAC [2] [3] de una posición en una secuencia de ADN que puede tener múltiples alternativas posibles. No deben confundirse con bases no canónicas porque cada secuencia particular tendrá de hecho una de las bases regulares. Se utilizan para codificar la secuencia de consenso de una población de secuencias alineadas y se utilizan, por ejemplo, en análisis filogenéticos para resumir en una sola secuencia múltiple o para búsquedas BLAST , aunque los símbolos degenerados IUPAC están enmascarados (ya que no están codificados).
En el sistema IUPAC, comúnmente utilizado, las nucleobases se representan con las primeras letras de sus nombres químicos: guanina, citosina, adenina y timina. [1] Esta abreviatura también incluye once caracteres de "ambigüedad" asociados con cada combinación posible de las cuatro bases del ADN. [4] Los caracteres de ambigüedad se diseñaron para codificar variaciones posicionales con el fin de informar errores de secuenciación del ADN , secuencias de consenso o polimorfismos de un solo nucleótido . La notación IUPAC, incluidos los caracteres de ambigüedad y las mnemotecnias sugeridas, se muestra en la Tabla 1.
A pesar de su amplia y casi universal aceptación, el sistema IUPAC tiene una serie de limitaciones, que se derivan de su dependencia del alfabeto romano. La escasa legibilidad de los caracteres romanos en mayúsculas, que se utilizan generalmente al mostrar datos genéticos, puede ser la principal de estas limitaciones. El valor de las proyecciones externas para distinguir letras ha sido bien documentado. [5] Sin embargo, estas proyecciones están ausentes en las letras mayúsculas, que en algunos casos solo se distinguen por sutiles pistas internas. Tomemos como ejemplo la C y la G mayúsculas utilizadas para representar la citosina y la guanina. Estos caracteres generalmente comprenden la mitad de los caracteres de una secuencia genética, pero se diferencian por una pequeña marca interna (dependiendo del tipo de letra). Sin embargo, estos caracteres romanos están disponibles en el conjunto de caracteres ASCII más comúnmente utilizado en las comunicaciones textuales, lo que refuerza la ubicuidad de este sistema.
Otra deficiencia de la notación IUPAC surge del hecho de que sus once caracteres de ambigüedad se han seleccionado de los caracteres restantes del alfabeto romano. Los autores de la notación se esforzaron por seleccionar caracteres de ambigüedad con mnemotecnia lógica. Por ejemplo, se utiliza S para representar la posibilidad de encontrar citosina o guanina en loci genéticos, los cuales forman fuertes interacciones de unión entre cadenas. Por el contrario, las interacciones más débiles de timina y adenina se representan con una W. Sin embargo, no se dispone de mnemotecnias convenientes para los otros caracteres de ambigüedad que se muestran en la Tabla 1. Esto ha hecho que los caracteres de ambigüedad sean difíciles de usar y puede explicar su aplicación limitada.
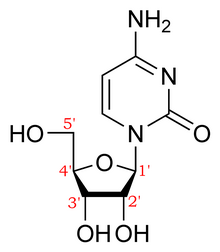
Las posiciones de los carbonos en el azúcar ribosa que forma la estructura principal de la cadena de ácidos nucleicos están numeradas y se utilizan para indicar la dirección de los ácidos nucleicos (5'->3' versus 3'->5'). Esto se conoce como direccionalidad . [3]
Los problemas de legibilidad asociados con los datos genéticos codificados por la IUPAC han llevado a los biólogos a considerar estrategias alternativas para la visualización de datos genéticos. Estos enfoques creativos para visualizar secuencias de ADN generalmente se han basado en el uso de símbolos distribuidos espacialmente y/o formas visualmente distintas para codificar secuencias extensas de ácidos nucleicos. Se han intentado notaciones alternativas para las secuencias de nucleótidos, sin embargo, la aceptación general ha sido baja. A continuación se resumen varios de estos enfoques.

En 1986, Cowin et al. describieron un nuevo método para visualizar secuencias de ADN conocido como proyección de pentagrama. [6] Su estrategia consistía en codificar nucleótidos como círculos en series de barras horizontales similares a las notas de un pentagrama musical. Como se ilustra en la Figura 1, cada espacio en el pentagrama de cinco líneas correspondía a una de las cuatro bases de ADN. La distribución espacial de los círculos hizo que fuera mucho más fácil distinguir bases individuales y comparar secuencias genéticas que los datos codificados por la IUPAC.
El orden de las bases (de arriba a abajo, G, A, T, C) se elige de manera que la cadena complementaria se pueda leer girando la proyección hacia abajo.
Zimmerman et al. adoptaron un enfoque diferente para visualizar los datos genéticos. [7] En lugar de basarse en círculos distribuidos espacialmente para resaltar las características genéticas, explotaron cuatro símbolos geométricamente diversos que se encuentran en una fuente de computadora estándar para distinguir las cuatro bases. Los autores desarrollaron una macro simple de WordPerfect para traducir los caracteres IUPAC en símbolos visualmente más distintivos.
Con la creciente disponibilidad de editores de fuentes, Jarvius y Landegren idearon un nuevo conjunto de símbolos genéticos, conocido como la fuente DNA Skyline, que utiliza bloques cada vez más altos para representar las diferentes bases de ADN. [8] Si bien recuerda a la proyección de pentagrama distribuida espacialmente de Cowin et al ., la fuente DNA Skyline es fácil de descargar y permite la traducción hacia y desde la notación IUPAC simplemente cambiando la fuente en la mayoría de las aplicaciones de procesamiento de texto estándar.

Los ambigramas (símbolos que transmiten un significado diferente cuando se observan en una orientación diferente) se han diseñado para reflejar las simetrías estructurales que se encuentran en la doble hélice del ADN. [9] Al asignar caracteres ambigráficos a bases complementarias (es decir, guanina: b, citosina: q, adenina: n y timina: u), es posible complementar secuencias de ADN simplemente rotando el texto 180 grados. [10] Una notación de ácido nucleico ambigráfica también facilita la identificación de palíndromos genéticos, como sitios de restricción de endonucleasas, como secciones de texto que se pueden rotar 180 grados sin cambiar la secuencia.
Un ejemplo de una notación de ácidos nucleicos ambigráfica es AmbiScript, una notación de ácidos nucleicos diseñada racionalmente que combina muchas de las características visuales y funcionales de sus predecesoras. [11] Su notación también utiliza caracteres desplazados espacialmente para facilitar la revisión visual y el análisis de datos genéticos. AmbiScript también fue diseñado para indicar posiciones ambiguas de nucleótidos mediante símbolos compuestos. Esta estrategia tenía como objetivo ofrecer una solución más intuitiva al uso de caracteres de ambigüedad propuestos por primera vez por la IUPAC. [4] Al igual que con las fuentes DNA Skyline de Jarvius y Landegren, las fuentes AmbiScript se pueden descargar y aplicar a datos de secuencias codificados por la IUPAC.
Los pares de bases de Watson y Crick se indican con un "•", un "-" o un "." (ejemplo: A•T o poly(rC)•2poly(rC)).
Los pares de bases de triple hélice de Hoogsteen se indican con un "*" o un ":" (ejemplo: C•G*G+, o T•A*T, o C•G*G, o T•A*A).