El código de barras de ADN de las algas se utiliza habitualmente para la identificación de especies yestudios filogenéticos . Las algas forman ungrupo filogenéticamente heterogéneo, lo que significa que la aplicación de un único código de barras/ marcador universal para la delimitación de especies no es viable, por lo que se aplican diferentes marcadores/códigos de barras para este objetivo en diferentes grupos de algas.
El código de barras de ADN de diatomeas es un método para la identificación taxonómica de diatomeas , incluso a nivel de especie . Se lleva a cabo utilizando ADN o ARN, seguido de la amplificación y secuenciación de regiones específicas y conservadas en el genoma de la diatomea, seguida de una asignación taxonómica.
Uno de los principales desafíos de la identificación de diatomeas es que a menudo se recolectan como una mezcla de diatomeas de varias especies. La codificación de barras de ADN es el proceso de identificación de especies individuales a partir de una muestra mixta de ADN ambiental (también llamado ADN ambiental extraido directamente del medio ambiente, como muestras de suelo o agua).
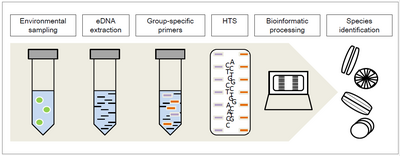
Un método recientemente aplicado es la codificación metabólica de ADN de diatomeas , que se utiliza para la evaluación de la calidad ecológica de ríos y arroyos debido a la respuesta específica de las diatomeas a condiciones ecológicas particulares. Como la identificación de especies a través de la morfología es relativamente difícil y requiere mucho tiempo y experiencia, [1] [2] la codificación metabólica de ADN mediante secuenciación de alto rendimiento (HTS) permite la asignación taxonómica y, por lo tanto, la identificación de la muestra completa con respecto a los cebadores específicos del grupo elegidos para la amplificación de ADN anterior .
Hasta ahora, ya se han desarrollado varios marcadores de ADN, principalmente dirigidos al ARNr 18S . [3] Utilizando la región hipervariable V4 del ADN de la subunidad pequeña ribosómica (ADNr SSU), se encontró que la identificación basada en ADN era más eficiente que el enfoque clásico basado en la morfología. [4] Otras regiones conservadas en los genomas que se utilizan con frecuencia como genes marcadores son la ribulosa-1-5-bisfosfato carboxilasa (rbcL), la citocromo oxidasa I (cox1, COI), [5] ITS [6] y 28S . [7] Se ha demostrado repetidamente que los datos moleculares obtenidos por la metacodificación de ADN ambiental de diatomeas reflejan con bastante fidelidad los índices bióticos de diatomeas basados en la morfología y, por lo tanto, proporcionan una evaluación similar del estado del ecosistema. [8] [9] Mientras tanto, las diatomeas se utilizan rutinariamente para la evaluación de la calidad ecológica en otros ecosistemas de agua dulce. [7] Junto con los invertebrados acuáticos , se consideran los mejores indicadores de perturbaciones relacionadas con las condiciones físicas, químicas o biológicas de los cursos de agua. Numerosos estudios están utilizando diatomeas bentónicas para biomonitoreo. [10] [11] [12] [13] Debido a que no se encontró un código de barras de ADN de diatomeas ideal, se ha propuesto que se utilicen diferentes marcadores para diferentes propósitos. De hecho, los genes altamente variables cox1, ITS y 28S se consideraron más adecuados para estudios taxonómicos, mientras que los genes 18S y rbcL más conservados parecen más apropiados para biomonitoreo.
La aplicación del concepto de código de barras de ADN a las diatomeas promete un gran potencial para resolver el problema de la identificación inexacta de las especies y facilitar así los análisis de la biodiversidad de muestras ambientales. [14]
Los métodos moleculares basados en la tecnología NGS casi siempre conducen a un mayor número de taxones identificados cuya presencia podría verificarse posteriormente mediante microscopía óptica. [4] Los resultados de este estudio proporcionan evidencia de que el código de barras de eDNA de diatomeas es adecuado para la evaluación de la calidad del agua y podría complementar o mejorar los métodos tradicionales. Stoeck et al. [15] también demostraron que el código de barras de eDNA proporciona una mayor comprensión de la diversidad de diatomeas u otras comunidades protistas y, por lo tanto, podría usarse para la proyección ecológica de la diversidad global. Otros estudios mostraron resultados diferentes. Por ejemplo, los inventarios obtenidos a partir del método basado en moléculas fueron más cercanos a los obtenidos por el método basado en la morfología cuando se enfocan las especies abundantes. [5]
La codificación metabólica del ADN también puede aumentar la resolución taxonómica y la comparabilidad entre regiones geográficas, lo que a menudo resulta difícil utilizando únicamente caracteres morfológicos. Además, la identificación basada en el ADN permite ampliar el rango de posibles bioindicadores, incluidos los grupos taxonómicos poco visibles que podrían ser altamente sensibles o tolerantes a factores estresantes particulares. De manera indirecta, los métodos moleculares también pueden ayudar a llenar los vacíos en el conocimiento de la ecología de las especies, al aumentar el número de muestras procesadas junto con una disminución en el tiempo de procesamiento (costo-efectividad), así como al aumentar la precisión y exactitud de la correlación entre la aparición de especies/MOTU y los factores ambientales. [16]
.jpg/440px-Naviculoid_diatom_(3074442495).jpg)
Actualmente no hay consenso sobre los métodos para la preservación y aislamiento de ADN, la elección de códigos de barras de ADN y cebadores de PCR, ni acuerdo sobre los parámetros de agrupamiento MOTU y su asignación taxonómica. [16] Los pasos de muestreo y moleculares deben estandarizarse a través de estudios de desarrollo. [5] Una de las principales limitaciones es la disponibilidad de códigos de barras de referencia para especies de diatomeas. La base de datos de referencia de taxones bioindicadores está lejos de estar completa a pesar de los esfuerzos constantes de numerosas iniciativas nacionales de códigos de barras, muchas especies aún carecen de información de códigos de barras. Además, la mayoría de los datos de metacodificación de barras existentes solo están disponibles localmente y geográficamente dispersos, lo que dificulta el desarrollo de herramientas útiles a nivel mundial. [16] Visco et al. [17] estimaron que no más del 30% de las especies de diatomeas europeas están actualmente representadas en bases de datos de referencia. Por ejemplo, existe una carencia importante para varias especies de las comunidades fennoscandianas (especialmente diatomeas acidófilas, como Eunotia incisa ). También se ha demostrado que la identificación taxonómica con códigos de barras de ADN no es precisa por encima del nivel de especie, por ejemplo para discriminar variedades (referencia faltante).
Otra limitación bien conocida de los códigos de barras para la identificación taxonómica es el método de agrupamiento utilizado antes de la asignación taxonómica: a menudo conduce a una pérdida masiva de información genética y la única forma fiable de evaluar los efectos de diferentes agrupamientos y diferentes procesos de asignación taxonómica sería comparar la lista de especies generada por diferentes procesos cuando se utiliza la misma base de datos de referencia. Esto aún no se ha hecho para la variedad de procesos utilizados en la evaluación molecular de las comunidades de diatomeas en Europa. [16] Las bases de datos validadas taxonómicamente, que incluyen comprobantes accesibles, también son cruciales para la identificación fiable de taxones mediante NGS. [18]
Además, a menudo se descubre que el sesgo de cebadores es una fuente importante de variación en los códigos de barras y la eficiencia de los cebadores de PCR puede diferir entre especies de diatomeas, es decir, algunos cebadores conducen a una amplificación preferencial de un taxón sobre otro. [16]
La inferencia de abundancia a partir de datos de metabarcoding se considera uno de los problemas más difíciles en el uso ambiental. [19] [20] El número de secuencias generadas por HTS no corresponde directamente al número de especímenes o biomasa y diferentes especies pueden producir diferentes cantidades de lecturas (por ejemplo, debido a diferencias en el tamaño del cloroplasto con el marcador rbcL). Vasselon et al. [21] crearon recientemente un factor de corrección de biovolumen cuando se utiliza el marcador rbcL. Por ejemplo, Achnanthidium minutissimum tiene un biovolumen pequeño y, por lo tanto, generará menos copias del fragmento rbcL (ubicado en el cloroplasto) que las especies más grandes. Este factor de corrección, sin embargo, requiere una calibración extensa con el biovolumen propio de cada especie y solo se ha probado en unas pocas especies hasta ahora. Las fluctuaciones del número de copias de genes para otros marcadores, como el marcador 18S, no parecen ser específicas de la especie, pero aún no se han probado.
Los marcadores de código de barras suelen combinar regiones hipervariables del genoma (para permitir la distinción entre especies) con regiones muy conservadas (para asegurar una especificidad para el organismo objetivo). Se han diseñado y utilizado con éxito varios marcadores de ADN, pertenecientes a los genomas nuclear, mitocondrial y de cloroplasto ( rbcL , COI , ITS + 5.8S , SSU , 18S ...), para la identificación de diatomeas con NGS. [22] [23] [6]
La región del gen 18S ha sido ampliamente utilizada como marcador en otros grupos de protistas [24] [25] y Jahn et al. [26] fueron los primeros en probar la región del gen 18S para el código de barras de diatomeas. Zimmerman et al. [7] propusieron un fragmento de 390-410 pb de largo del locus del gen 18S rRNA de 1800 pb de largo como marcador de código de barras para el análisis de muestras ambientales con HTS. y analiza su uso y limitaciones para la identificación de diatomeas. Este fragmento incluye la subunidad V4, que es la más grande y compleja de las regiones altamente variables dentro del locus 18S. [27] Destacaron que esta región hipervariable del gen 18S tiene un gran potencial para estudiar la diversidad de protistas a gran escala, pero tiene una eficiencia limitada para la identificación por debajo del nivel de especie o especies crípticas.
El gen rbcl se utiliza para estudios de taxonomía (Trobajo et al. 2009), cuyos beneficios incluyen que rara vez hay variación intragenómica y que son muy fáciles de alinear y comparar. Una biblioteca de referencia de acceso abierto, llamada R-Syst::diatom, incluye datos para dos códigos de barras (18S y rbcL). Se puede acceder libremente a ella a través de un sitio web. [28] Kermmarec et al. [5] también utilizaron con éxito el gen rbcL para la evaluación ecológica de las diatomeas. El marcador rbcL también se alinea y compara fácilmente.
Moniz y Kaczmarska [23] investigaron el éxito de amplificación de los marcadores SSU, COI e ITS2 y descubrieron que el fragmento ITS-2 + 5.8S de 300 a 400 pb proporcionó la mayor tasa de éxito de amplificación y una buena resolución de especies. Este marcador se utilizó posteriormente para separar especies morfológicamente definidas con una tasa de éxito del 99,5%. A pesar de este éxito de amplificación, Zimmerman et al. [7] criticaron el uso de ITS-2 debido a la heterogeneidad intraindividual. Se ha sugerido que los marcadores SSU [7] o rbcL (Mann et al., 2010) son menos heterogéneos entre individuos y, por lo tanto, más beneficiosos para distinguir entre especies.
Las diatomeas se utilizan rutinariamente como parte de un conjunto de herramientas de biomonitoreo que deben monitorearse como parte de la Directiva Marco Europea del Agua. [29] Las diatomeas se utilizan como un indicador de la salud del ecosistema en aguas dulces porque son ubicuas, se ven directamente afectadas por los cambios en los parámetros fisicoquímicos y muestran una mejor relación con las variables ambientales que otros taxones, por ejemplo, los invertebrados, lo que brinda una mejor imagen general de la calidad del agua. [30]

En los últimos años, los investigadores han desarrollado y estandarizado las herramientas para la metacodificación y secuenciación de diatomeas, para complementar la evaluación tradicional mediante microscopía, abriendo una nueva vía de biomonitoreo para sistemas acuáticos. [31] El uso de diatomeas bentónicas a través de un método de secuenciación de próxima generación para el biomonitoreo de ríos reveló un buen potencial en él. [5] Muchos estudios han demostrado que la metacodificación y la HTS (secuenciación de alto rendimiento) se pueden utilizar para estimar el estado de calidad y la diversidad en aguas dulces. Como parte de la Agencia de Medio Ambiente, Kelly et al. [32] ha desarrollado un enfoque de metacodificación basado en ADN para evaluar las comunidades de diatomeas en ríos para el Reino Unido. Vasselon et al. [33] compararon los enfoques morfológicos y HTS para diatomeas y encontraron que HTS dio una indicación confiable del estado de calidad para la mayoría de los ríos en términos del Índice de Sensibilidad a la Contaminación Específica (SPI). [34] También aplicaron la metacodificación de ADN de las comunidades de diatomeas a la red de monitoreo de ríos en la isla tropical Mayotte (DOM-TOM en francés).
Rimet et al. [35] también exploraron la posibilidad de utilizar HTS para evaluar la diversidad de diatomeas y demostraron que los índices de diversidad tanto del HTS como del análisis microscópico estaban bien correlacionados, aunque no eran perfectos.
Los códigos de barras y metacódigos de barras de ADN se pueden utilizar para establecer métricas e índices moleculares que potencialmente brindan conclusiones muy similares a las de los enfoques tradicionales sobre el estado ecológico y ambiental de los ecosistemas acuáticos. [36]
Las diatomeas se utilizan como herramienta de diagnóstico para el ahogamiento en las prácticas forenses. La prueba de diatomeas se basa en el principio de la inhalación de diatomeas desde el agua hasta los pulmones y su distribución y deposición alrededor del cuerpo. Los métodos de ADN se pueden utilizar para confirmar si la causa de la muerte fue efectivamente el ahogamiento y localizar el origen del ahogamiento. [37] La codificación de barras de ADN de diatomeas proporciona la oportunidad de analizar rápidamente la comunidad de diatomeas presente en un cuerpo y localizar el origen del ahogamiento e investigar si un cuerpo puede haber sido movido de un lugar a otro.
La metacodificación de diatomeas puede ayudar a delimitar especies crípticas que son difíciles de identificar mediante microscopía y ayudar a completar bases de datos de referencia comparando conjuntos morfológicos con datos de metacodificación de diatomeas. [35]
Las clorofitas poseen un linaje antiguo y taxonómicamente muy diverso (Fang et al. 2014), que incluye también a las plantas terrestres. Aunque se han descrito más de 14 000 especies basándose en criterios estructurales y ultraestructurales (Hall et al. 2010), su identificación morfológica suele ser limitada.
Se han propuesto varios códigos de barras para las clorofitas para la identificación basada en el ADN con el fin de evitar los problemas de la identificación morfológica. Aunque el gen codificante de la citocromo oxidasa I (COI, COX) (enlace) es un código de barras estándar para los animales, resultó ser insatisfactorio para las clorofitas porque el gen contiene varios intrones en este grupo de algas (Turmel et al. 2002). Los genes marcadores nucleares que se han utilizado para las clorofitas son SSU rDNA, LSU rDNA, rDNA ITS (Leliaert et al. 2014). [38]
Las macroalgas (una agrupación morfológica más que taxonómica) pueden ser muy difíciles de identificar debido a su morfología simple, plasticidad fenotípica y etapas alternativas del ciclo de vida. Por lo tanto, la sistemática e identificación de las algas han llegado a depender en gran medida de herramientas genéticas/moleculares como el código de barras del ADN . [39] [40] El gen SSU rADN es un código de barras de uso común para estudios filogenéticos sobre macroalgas. [41] Sin embargo, el SSU rADN es una región altamente conservada y generalmente carece de resolución para la identificación de especies.
En las últimas 2 décadas se han desarrollado ciertos estándares para el código de barras de ADN con el objetivo de identificar especies para cada uno de los principales grupos de macroalgas. [42] [39] [43] [44] [45] El gen de la subunidad I de la citocromo c oxidasa (COI) se usa comúnmente como código de barras para las algas rojas y marrones, mientras que tufA (factor de elongación de plástidos), rbcL (subunidad grande de la rubisco) e ITS ( espaciador de transcripción interna ) se usan comúnmente para las algas verdes. [41] [45] Estos códigos de barras suelen tener una longitud de 600-700 pb.
Los códigos de barras suelen diferir entre los tres grupos principales de macroalgas (rojas, verdes y marrones) porque su herencia evolutiva es muy diversa. [46] Las macroalgas son un grupo polifilético , lo que significa que dentro del grupo no todas comparten un ancestro común reciente, lo que dificulta encontrar un gen que se conserve entre todos pero que sea lo suficientemente variable para la identificación de las especies.
Adaptado de [40]
Puede encontrar información detallada sobre los códigos de barras de ADN de diferentes organismos aquí: