En bioinformática , GLIMMER (Gene Locator and Interpolated Markov ModelER) se utiliza para encontrar genes en ADN procariota . [1] "Es eficaz para encontrar genes en bacterias , arqueas y virus , y normalmente encuentra el 98-99% de todos los genes codificantes de proteínas relativamente largos ". [1] GLIMMER fue el primer sistema que utilizó el modelo interpolado de Markov [2] para identificar regiones codificantes. El software GLIMMER es de código abierto y lo mantienen Steven Salzberg , Art Delcher y sus colegas del Centro de Biología Computacional [3] de la Universidad Johns Hopkins . Los algoritmos y el software GLIMMER originales fueron diseñados por Art Delcher, Simon Kasif y Steven Salzberg y se aplicaron a la anotación del genoma bacteriano en colaboración con Owen White .
La primera versión de GLIMMER, "es decir, GLIMMER 1.0", se lanzó en 1998 y se publicó en el artículo Microbial gene identity using interpolated Markov model . [1] Los modelos de Markov se utilizaron para identificar genes microbianos en GLIMMER 1.0. GLIMMER considera las dependencias de la secuencia de composición local, lo que hace que GLIMMER sea más flexible y más potente en comparación con el modelo de Markov de orden fijo .
En el artículo Microbial gene identity using interpolated Markov models (Identificación de genes microbianos mediante modelos interpolados de Markov) se hizo una comparación entre el modelo de Markov interpolado utilizado por GLIMMER y el modelo de Markov de quinto orden . [1] "El algoritmo GLIMMER encontró 1680 genes de 1717 genes anotados en Haemophilus influenzae , donde el modelo de Markov de quinto orden encontró 1574 genes. GLIMMER encontró 209 genes adicionales que no estaban incluidos en los 1717 genes anotados, donde el modelo de Markov de quinto orden encontró 104 genes". [1]
La segunda versión de GLIMMER, es decir, GLIMMER 2.0, se lanzó en 1999 y se publicó en el artículo Identificación microbiana mejorada con GLIMMER . [4] Este artículo [4] proporciona mejoras técnicas significativas, como el uso del modelo de contexto interpolado en lugar del modelo de Markov interpolado y la resolución de genes superpuestos que mejoran la precisión de GLIMMER.
Se utilizan modelos de contexto interpolados en lugar del modelo de Markov interpolado , lo que brinda la flexibilidad de seleccionar cualquier base. En el modelo de Markov interpolado, la distribución de probabilidad de una base se determina a partir de las bases inmediatamente anteriores. Si la base inmediatamente anterior es una traducción de aminoácidos irrelevante , el modelo de Markov interpolado aún considera la base anterior para determinar la probabilidad de la base dada, mientras que el modelo de contexto interpolado que se utilizó en GLIMMER 2.0 puede ignorar las bases irrelevantes. Las predicciones de falsos positivos se incrementaron en GLIMMER 2.0 para reducir la cantidad de predicciones de falsos negativos. Los genes superpuestos también se resuelven en GLIMMER 2.0.
En el artículo Improved microbial identity with GLIMMER [4] se han realizado varias comparaciones entre GLIMMER 1.0 y GLIMMER 2.0 , que muestra mejoras en la versión posterior. "La sensibilidad de GLIMMER 1.0 oscila entre el 98,4 y el 99,7 %, con una media del 99,1 %, mientras que GLIMMER 2.0 tiene una sensibilidad del 98,6 al 99,8 %, con una media del 99,3 %. GLIMMER 2.0 es muy eficaz para encontrar genes de alta densidad. El parásito Trypanosoma brucei , responsable de causar la enfermedad del sueño africana , está siendo identificado por GLIMMER 2.0" [4]
La tercera versión de GLIMMER, "GLIMMER 3.0", se lanzó en 2007 y se publicó en el artículo Identifying organisms and endosymbiont DNA with Glimmer [Identificación de genes bacterianos y ADN endosimbionte con Glimmer] . [5] Este artículo describe varios cambios importantes realizados en el sistema GLIMMER, incluidos métodos mejorados para identificar regiones codificantes y codones de inicio . La puntuación de ORF en GLIMMER 3.0 se realiza en orden inverso, es decir, comienza desde el codón de terminación y retrocede hacia el codón de inicio. El escaneo inverso ayuda a identificar la porción codificante del gen con mayor precisión, que está contenida en la ventana de contexto de IMM. GLIMMER 3.0 también mejora los datos del conjunto de entrenamiento generados al comparar el ORF largo con la distribución universal de aminoácidos de genomas bacterianos muy dispares. "GLIMMER 3.0 tiene un resultado promedio de ORF largo del 57 % para varios organismos, mientras que GLIMMER 2.0 tiene un resultado promedio de ORF largo del 39 %". [5]
GLIMMER 3.0 reduce la tasa de predicciones de falsos positivos, que se incrementó en GLIMMER 2.0 para reducir la cantidad de predicciones de falsos negativos. "GLIMMER 3.0 tiene una precisión de predicción del sitio de inicio del 99,5 % para coincidencias de 3'5', mientras que GLIMMER 2.0 tiene un 99,1 % para coincidencias de 3'5'. GLIMMER 3.0 utiliza un nuevo algoritmo para escanear regiones codificantes, un nuevo módulo de detección del sitio de inicio y una arquitectura que integra todas las predicciones de genes en un genoma completo". [5]
Longitud mínima de descripción
El proyecto GLIMMER ayudó a introducir y popularizar el uso de modelos de longitud variable en biología computacional y bioinformática, que posteriormente se han aplicado a numerosos problemas, como la clasificación de proteínas y otros. El modelado de longitud variable fue iniciado originalmente por teóricos de la información y posteriormente aplicado ingeniosamente y popularizado en la compresión de datos (por ejemplo, la compresión Ziv-Lempel). La predicción y la compresión están íntimamente vinculadas mediante el uso de principios de longitud mínima de descripción . La idea básica es crear un diccionario de palabras frecuentes (motivos en secuencias biológicas). La intuición es que los motivos que aparecen con frecuencia tienen más probabilidades de ser más predictivos e informativos. En GLIMMER, el modelo interpolado es un modelo de mezcla de las probabilidades de estos motivos relativamente comunes. De manera similar al desarrollo de los HMM en biología computacional, los autores de GLIMMER se vieron conceptualmente influenciados por la aplicación previa de otra variante de modelos de Markov interpolados al reconocimiento de voz por parte de investigadores como Fred Jelinek (IBM) y Eric Ristad (Princeton). El algoritmo de aprendizaje en GLIMMER es diferente de estos enfoques anteriores.
GLIMMER se puede descargar desde la página de inicio de Glimmer (requiere un compilador de C++). Alternativamente, NCBI aloja una versión en línea [1].
Los pasos explicados anteriormente describen la funcionalidad básica de GLIMMER. Se han realizado varias mejoras en GLIMMER y algunas de ellas se describen en las siguientes subsecciones.
El sistema GLIMMER consta de dos programas. El primer programa, llamado build-imm, toma un conjunto de secuencias de entrada y genera el modelo de Markov interpolado de la siguiente manera.
Se calcula la probabilidad de cada base, es decir, A, C, G, T para todos los k-meros para 0 ≤ k ≤ 8. Luego, para cada k-mero , GLIMMER calcula el peso. La probabilidad de la nueva secuencia se calcula de la siguiente manera.
donde n es la longitud de la secuencia es el oligómero en la posición x. , la puntuación del modelo de Markov interpolado de orden se calcula como
"donde es el peso del k-mero en la posición x-1 en la secuencia S y es la estimación obtenida a partir de los datos de entrenamiento de la probabilidad de la base ubicada en la posición x en el modelo de orden x". [1]
La probabilidad de base dadas las i bases anteriores se calcula de la siguiente manera.
"El valor de asociado con puede considerarse como una medida de confianza en la precisión de este valor como una estimación de la probabilidad verdadera. GLIMMER utiliza dos criterios para determinar . El primero de ellos es la frecuencia de ocurrencia simple en la que el número de ocurrencias de la cadena de contexto en los datos de entrenamiento excede un valor de umbral específico, luego se establece en 1.0. El valor predeterminado actual para el umbral es 400, lo que brinda una confianza del 95%. Cuando no hay suficientes ocurrencias de muestra de una cadena de contexto, build-imm emplea criterios adicionales para determinar el valor. Para una cadena de contexto dada de longitud i, build-imm compara las frecuencias observadas de la siguiente base , , , con las probabilidades del modelo de Markov interpoladas calculadas previamente utilizando el siguiente contexto más corto, , , , . Utilizando una prueba, build-imm determina qué tan probable es que las cuatro frecuencias observadas sean consistentes con los valores IMM del siguiente contexto más corto". [1]
El segundo programa, llamado glimmer, utiliza este IMM para identificar genes putativos en un genoma completo. GLIMMER identifica todos los marcos de lectura abiertos que tienen una puntuación superior al umbral y comprueba si hay genes superpuestos. La resolución de genes superpuestos se explica en la siguiente subsección.
Las ecuaciones y la explicación de los términos utilizados anteriormente se tomaron del artículo 'Identificación de genes microbianos utilizando modelos de Markov interpolados [1]'
En GLIMMER 1.0, cuando dos genes A y B se superponen, se puntúa la región de superposición. Si A es más largo que B, si A tiene una puntuación más alta en la región de superposición y si mover el sitio de inicio de B no resuelve la superposición, entonces se rechaza B.
GLIMMER 2.0 proporcionó una mejor solución para resolver la superposición. En GLIMMER 2.0, cuando dos genes potenciales A y B se superponen, se puntúa la región de superposición. Supongamos que el gen A tiene una puntuación más alta, se consideran cuatro orientaciones diferentes.
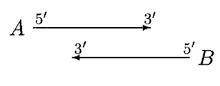
En el caso anterior, el desplazamiento de los sitios de inicio no elimina la superposición. Si A es significativamente más largo que B, entonces se rechaza B o, de lo contrario, tanto A como B se consideran genes, con una superposición dudosa.
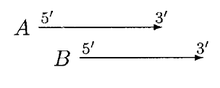
En el caso anterior, mover B puede resolver la superposición; A y B pueden considerarse genes no superpuestos, pero si B es significativamente más corto que A, entonces B se rechaza.

En el caso anterior, mover A puede resolver la superposición. A solo se mueve si la superposición es una fracción pequeña de A o, de lo contrario, B se rechaza.

En el caso anterior, se pueden mover tanto A como B. Primero movemos el inicio de B hasta que la región de superposición tenga una puntuación más alta para B. Luego movemos el inicio de A hasta que tenga una puntuación más alta. Luego movemos B nuevamente, y así sucesivamente, hasta que se elimine la superposición o no se puedan hacer más movimientos.
El ejemplo anterior se ha tomado del artículo 'Identificación de genes bacterianos y ADN endosimbionte con Glimmer' [5]
La señal del sitio de unión del ribosoma (RBS) se puede utilizar para encontrar la verdadera posición del sitio de inicio. Los resultados de GLIMMER se pasan como entrada al programa RBSfinder para predecir los sitios de unión del ribosoma. GLIMMER 3.0 integra el programa RBSfinder en la función de predicción de genes.
El software ELPH (que se determinó como altamente efectivo para identificar RBS en el artículo [5] ) se utiliza para identificar RBS y está disponible en este sitio web Archivado el 27 de noviembre de 2013 en Wayback Machine . El algoritmo de muestreo de Gibbs se utiliza para identificar motivos compartidos en cualquier conjunto de secuencias. Estas secuencias de motivos compartidos y su longitud se proporcionan como entrada a ELPH. Luego, ELPH calcula la matriz de peso de posición (PWM) que será utilizada por GLIMMER 3 para puntuar cualquier RBS potencial encontrado por RBSfinder. El proceso anterior se realiza cuando tenemos una cantidad sustancial de genes de entrenamiento. Si hay una cantidad inadecuada de genes de entrenamiento, GLIMMER 3 puede autoarrancarse para generar un conjunto de predicciones de genes que se pueden usar como entrada para ELPH. ELPH ahora calcula PWM y este PWM se puede volver a utilizar en el mismo conjunto de genes para obtener resultados más precisos para los sitios de inicio. Este proceso se puede repetir durante muchas iteraciones para obtener resultados de predicción de genes y PWM más consistentes.
Glimmer apoya los esfuerzos de anotación de genomas en una amplia gama de especies bacterianas, arqueales y virales. En un esfuerzo de reanotación a gran escala en el Banco de Datos de ADN de Japón (DDBJ, que refleja Genbank ), Kosuge et al. (2006) [6] examinaron los métodos de búsqueda de genes utilizados para 183 genomas. Informaron que de estos proyectos, Glimmer fue el buscador de genes para el 49%, seguido de GeneMark con el 12%, con otros algoritmos utilizados en el 3% o menos de los proyectos. (También informaron que el 33% de los genomas utilizaron "otros" programas, lo que en muchos casos significaba que no podían identificar el método. Excluyendo esos casos, Glimmer se utilizó para el 73% de los genomas para los que los métodos pudieron identificarse de manera inequívoca). Glimmer fue utilizado por el DDBJ para volver a anotar todos los genomas bacterianos en las Bases de Datos Internacionales de Secuencias de Nucleótidos. [7] Este grupo también lo está utilizando para anotar virus. [8] Glimmer es parte del proceso de anotación bacteriana en el Centro Nacional de Información Biotecnológica (NCBI), [9] que también mantiene un servidor web para Glimmer, [10] al igual que sitios en Alemania, [11] Canadá. [12]
Según Google Scholar, a principios de 2011, el artículo original de Glimmer (Salzberg et al., 1998) [1] ha sido citado 581 veces, y el artículo de Glimmer 2.0 (Delcher et al., 1999) [4] ha sido citado 950 veces.