La 5-metilcitosina es una forma metilada de la base de ADN citosina (C) que regula la transcripción genética y asume varias otras funciones biológicas. [1] Cuando la citosina está metilada, el ADN mantiene la misma secuencia, pero la expresión de los genes metilados puede alterarse (el estudio de esto es parte del campo de la epigenética ). La 5-metilcitosina está incorporada en el nucleósido 5-metilcitidina .
En la 5-metilcitosina, un grupo metilo está unido al quinto átomo del anillo de 6 átomos, contando en sentido antihorario desde el nitrógeno unido al NH en la posición de las seis en punto. Este grupo metilo distingue a la 5-metilcitosina de la citosina.
Mientras intentaba aislar la toxina bacteriana responsable de la tuberculosis , WG Ruppel aisló un nuevo ácido nucleico llamado ácido tuberculínico en 1898 del bacilo tuberculoso . [2] Se descubrió que el ácido nucleico era inusual, ya que contenía además de timina , guanina y citosina , un nucleótido metilado. En 1925, Johnson y Coghill detectaron con éxito una cantidad menor de un derivado de citosina metilada como producto de la hidrólisis del ácido tuberculínico con ácido sulfúrico . [3] [4] Este informe fue severamente criticado porque su identificación se basó únicamente en las propiedades ópticas del picrato cristalino , y otros científicos no lograron reproducir el mismo resultado. [5] Pero su existencia se demostró finalmente en 1948, cuando Hotchkiss separó los ácidos nucleicos del ADN del timo de ternera utilizando cromatografía en papel , mediante la cual detectó una citosina metilada única, bastante distinta de la citosina y el uracilo convencionales . [6] Después de siete décadas, resultó que también es una característica común en diferentes moléculas de ARN , aunque el papel preciso es incierto. [7]
La función de esta sustancia química varía significativamente entre especies: [8]
Mientras que la desaminación espontánea de la citosina forma uracilo , que es reconocido y eliminado por las enzimas de reparación del ADN, la desaminación de la 5-metilcitosina forma timina . Esta conversión de una base de ADN de citosina (C) a timina (T) puede dar lugar a una mutación de transición . [11] Además, la desaminación enzimática activa de la citosina o la 5-metilcitosina por la familia APOBEC de citosina deaminasas podría tener implicaciones beneficiosas en varios procesos celulares, así como en la evolución de los organismos. [12] Por otro lado, las implicaciones de la desaminación en la 5-hidroximetilcitosina siguen siendo menos conocidas.
El grupo NH2 se puede eliminar (desaminación) de la 5-metilcitosina para formar timina con el uso de reactivos como el ácido nitroso ; la citosina se desamina a uracilo (U) en condiciones similares. [ cita requerida ]
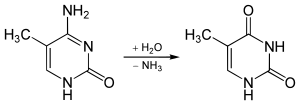
La 5-metilcitosina es resistente a la desaminación por tratamiento con bisulfito , que desamina los residuos de citosina. Esta propiedad se suele aprovechar para analizar los patrones de metilación de la citosina del ADN con secuenciación con bisulfito . [13]
Las marcas de 5mC se colocan en el ADN genómico a través de las metiltransferasas de ADN (DNMT). Hay 5 DNMT en humanos: DNMT1, DNMT2, DNMT3A, DNMT3B y DNMT3L, y en las algas y los hongos hay 3 más (DNMT4, DNMT5 y DNMT6). [14] DNMT1 contiene la secuencia de orientación de focos de replicación (RFTS) y el dominio CXXC que catalizan la adición de marcas de 5mC. RFTS dirige DNMT1 a loci de replicación de ADN para ayudar en el mantenimiento de 5mC en las hebras hijas durante la replicación de ADN, mientras que CXXC contiene un dominio de dedo de zinc para la adición de novo de metilación al ADN. [15] Se descubrió que DNMT1 era la metiltransferasa de ADN predominante en todo el tejido humano. [16] Principalmente, DNMT3A y DNMT3B son responsables de la metilación de novo , y DNMT1 mantiene la marca de 5mC después de la replicación. [1] Las DNMT pueden interactuar entre sí para aumentar la capacidad de metilación. Por ejemplo, 2 DNMT3L pueden formar un complejo con 2 DNMT3A para mejorar las interacciones con el ADN, facilitando la metilación. [17] Los cambios en la expresión de DNMT dan como resultado una metilación aberrante. La sobreexpresión produce un aumento de la metilación, mientras que la alteración de la enzima reduce los niveles de metilación. [16]

El mecanismo de adición es el siguiente: primero, un residuo de cisteína en el motivo PCQ de la DNMT crea un ataque nucleofílico en el carbono 6 sobre el nucleótido de citosina que se va a metilar. Luego, la S-adenosilmetionina dona un grupo metilo al carbono 5. Una base en la enzima DNMT desprotona el hidrógeno residual en el carbono 5 restaurando el doble enlace entre el carbono 5 y 6 en el anillo, produciendo el par de bases 5-metilcitosina. [15]
Después de que una citosina se metila a 5mC, se puede revertir a su estado inicial a través de múltiples mecanismos. La desmetilación pasiva del ADN por dilución elimina la marca gradualmente a través de la replicación por falta de mantenimiento por DNMT. En la desmetilación activa del ADN, una serie de oxidaciones lo convierte en 5-hidroximetilcitosina (5hmC), 5-formilcitosina (5fC) y 5-carboxilcitosina (5caC), y las dos últimas son finalmente escindidas por la ADN glicosilasa de timina (TDG), seguida de la reparación por escisión de bases (BER) para restaurar la citosina. [1] La eliminación de TDG produjo un aumento de 5fC de 2 veces sin ningún cambio estadísticamente significativo en los niveles de 5hmC, lo que indica que 5mC debe oxidarse iterativamente al menos dos veces antes de su desmetilación completa. [18] La oxidación ocurre a través de las dioxigenasas de la familia TET (translocación diez-once) ( enzimas TET ) que pueden convertir 5mC, 5hmC y 5fC en sus formas oxidadas. Sin embargo, la enzima tiene la mayor preferencia por 5mC y la velocidad de reacción inicial para las conversiones de 5hmC y 5fC con TET2 es 4,9-7,6 veces más lenta. [19] La TET requiere Fe(II) como cofactor, y oxígeno y α-cetoglutarato (α-KG) como sustratos, y el último sustrato se genera a partir de isocitrato por la enzima isocitrato deshidrogenasa (IDH). [20] Sin embargo, el cáncer puede producir 2-hidroxiglutarato (2HG) que compite con α-KG, reduciendo la actividad de TET y, a su vez, reduciendo la conversión de 5mC a 5hmC. [21]
En el cáncer, el ADN puede metilarse tanto en exceso, lo que se denomina hipermetilación , como en defecto, lo que se denomina hipometilación. [22] Las islas CpG que se superponen a los promotores de genes se metilan de novo , lo que da lugar a una inactivación aberrante de genes normalmente asociados a la inhibición del crecimiento de los tumores (un ejemplo de hipermetilación). [23] Al comparar el tejido tumoral y el normal, el primero tenía niveles elevados de las metiltransferasas DNMT1, DNMT3A y, principalmente, DNMT3B, todas ellas asociadas a los niveles anormales de 5mC en el cáncer. [16] Las secuencias repetidas en el genoma, incluido el ADN satélite, Alu y los elementos intercalados largos (LINE), a menudo se observan hipometiladas en el cáncer, lo que da lugar a la expresión de estos genes normalmente silenciados, y los niveles suelen ser marcadores significativos de la progresión tumoral. [22] Se ha planteado la hipótesis de que existe una conexión entre la hipermetilación y la hipometilación; La sobreactividad de las metiltransferasas del ADN que producen la metilación anormal de novo de 5mC puede compensarse mediante la eliminación de la metilación, un tipo de reparación epigenética. Sin embargo, la eliminación de la metilación es ineficiente y da como resultado un exceso de hipometilación en todo el genoma. También puede ocurrir lo contrario: la sobreexpresión de la hipometilación puede verse silenciada por la hipermetilación en todo el genoma. [22] Es probable que las capacidades distintivas del cáncer se adquieran a través de cambios epigenéticos que alteran el 5mC tanto en las células cancerosas como en el estroma asociado al tumor circundante dentro del microambiente tumoral. [24] Se ha informado que el fármaco anticanceroso cisplatino reacciona con el 5mC. [25]
La "edad epigenética" se refiere a la conexión entre la edad cronológica y los niveles de metilación del ADN en el genoma. [26] La combinación de los niveles de metilación del ADN, en conjuntos específicos de CpG llamados "CpG de reloj", con algoritmos que hacen una regresión de los niveles típicos de metilación colectiva del genoma en una edad cronológica dada, permite la predicción de la edad epigenética. Durante la juventud (0 a 20 años), los cambios en la metilación del ADN ocurren a un ritmo más rápido a medida que avanza el desarrollo y el crecimiento, y los cambios comienzan a desacelerarse a edades más avanzadas. Existen múltiples estimadores de la edad epigenética. El reloj de Horvath mide un conjunto multitejido de 353 CpG, la mitad de los cuales se correlacionan positivamente con la edad y la otra mitad negativamente, para estimar la edad epigenética. [27] El reloj de Hannum utiliza muestras de sangre de adultos para calcular la edad basándose en una base ortogonal de 71 CpG. [28] El reloj de Levine, conocido como DNAm PhenoAge, depende de 513 CpG y supera a los otros estimadores de edad en la predicción de la mortalidad y la esperanza de vida, pero muestra sesgo con los tejidos no sanguíneos. [29] Hay informes de estimadores de edad con el estado de metilación de solo un CpG en el gen ELOVL2. [30] La estimación de la edad permite predecir la esperanza de vida a través de las expectativas de condiciones relacionadas con la edad a las que pueden estar sujetos los individuos en función de sus marcadores de metilación de 5mC. [ cita requerida ]