El ADN del cloroplasto ( cpDNA ) es el ADN ubicado en los cloroplastos, que son orgánulos fotosintéticos ubicados dentro de las células de algunos organismos eucariotas. Los cloroplastos, como otros tipos de plástidos , contienen un genoma separado del del núcleo celular . La existencia del ADN del cloroplasto se identificó bioquímicamente en 1959, [1] y se confirmó mediante microscopía electrónica en 1962. [2] Los descubrimientos de que el cloroplasto contiene ribosomas [3] y realiza la síntesis de proteínas [4] revelaron que el cloroplasto es genéticamente semiautónomo. Las primeras secuencias completas del genoma del cloroplasto se publicaron en 1986, Nicotiana tabacum (tabaco) por Sugiura y colegas y Marchantia polymorpha (hepática) por Ozeki et al. [5] [6] Desde entonces, se ha secuenciado una gran cantidad de ADN de cloroplastos de varias especies .
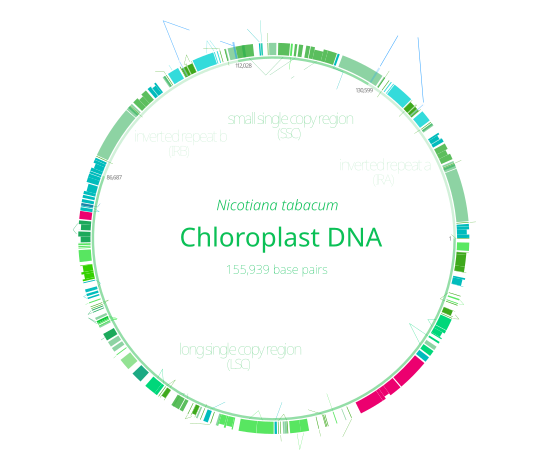
Los ADN de los cloroplastos son circulares y suelen tener una longitud de entre 120.000 y 170.000 pares de bases . [7] [8] [9] Pueden tener una longitud de contorno de alrededor de 30 a 60 micrómetros y una masa de alrededor de 80 a 130 millones de daltons . [10]
La mayoría de los cloroplastos tienen todo su genoma cloroplástico combinado en un único anillo grande, aunque los de las algas dinófitas son una notable excepción: su genoma está dividido en unos cuarenta plásmidos pequeños , cada uno de 2.000 a 10.000 pares de bases de longitud. [11] Cada minicírculo contiene de uno a tres genes, [11] pero también se han encontrado plásmidos en blanco, sin ADN codificante .
Durante mucho tiempo se ha pensado que el ADN del cloroplasto tiene una estructura circular, pero algunas evidencias sugieren que el ADN del cloroplasto generalmente adopta una forma lineal. [12] Se ha observado que más del 95 % del ADN del cloroplasto en los cloroplastos del maíz tiene una forma lineal ramificada en lugar de círculos individuales. [11]
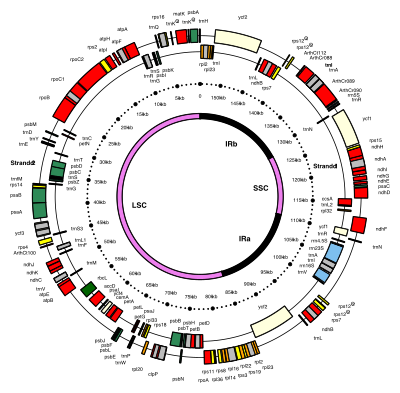
Muchos ADN de cloroplastos contienen dos repeticiones invertidas , que separan una sección de copia única larga (LSC) de una sección de copia única corta (SSC). [9]
Las repeticiones invertidas varían enormemente en longitud, desde 4.000 a 25.000 pares de bases de longitud cada una. [11] Las repeticiones invertidas en plantas tienden a estar en el extremo superior de este rango, cada una con una longitud de 20.000 a 25.000 pares de bases. [9] [13] Las regiones de repetición invertida generalmente contienen tres genes de ARN ribosómico y dos genes de ARNt , pero se pueden expandir o reducir para contener tan solo cuatro o hasta más de 150 genes. [11] Si bien un par dado de repeticiones invertidas rara vez son completamente idénticas, siempre son muy similares entre sí, aparentemente como resultado de una evolución concertada . [11]
Las regiones de repetición invertida están altamente conservadas entre las plantas terrestres y acumulan pocas mutaciones. [9] [13] Existen repeticiones invertidas similares en los genomas de las cianobacterias y los otros dos linajes de cloroplastos ( glaucophyta y rhodophyceæ ), lo que sugiere que son anteriores al cloroplasto, [11] aunque algunos ADN de cloroplastos como los de los guisantes y algunas algas rojas [11] han perdido desde entonces las repeticiones invertidas. [13] [14] Otros, como el alga roja Porphyra, invirtieron una de sus repeticiones invertidas (convirtiéndolas en repeticiones directas). [11] Es posible que las repeticiones invertidas ayuden a estabilizar el resto del genoma del cloroplasto, ya que los ADN de cloroplastos que han perdido algunos de los segmentos de repetición invertida tienden a reorganizarse más. [14]
Cada cloroplasto contiene alrededor de 100 copias de su ADN en las hojas jóvenes, cifra que se reduce a 15-20 copias en las hojas más viejas. [15] Por lo general, se encuentran empaquetadas en nucleoides que pueden contener varios anillos de ADN de cloroplasto idénticos. Se pueden encontrar muchos nucleoides en cada cloroplasto. [10]
Aunque el ADN del cloroplasto no está asociado con histonas verdaderas , [16] en las algas rojas se ha encontrado una proteína de cloroplasto similar a una histona (HC) codificada por el ADN del cloroplasto que empaqueta firmemente cada anillo de ADN del cloroplasto en un nucleoide . [17]
En las algas rojas primitivas , los nucleoides del ADN del cloroplasto están agrupados en el centro de un cloroplasto, mientras que en las plantas verdes y las algas verdes , los nucleoides están dispersos por todo el estroma . [17]
Se han secuenciado más de 5000 genomas de cloroplastos y son accesibles a través de la base de datos de genomas de orgánulos del NCBI. [18] Los primeros genomas de cloroplastos se secuenciaron en 1986, del tabaco ( Nicotiana tabacum ) [19] y la hepática ( Marchantia polymorpha ). [20] La comparación de las secuencias genéticas de la cianobacteria Synechocystis con las del genoma del cloroplasto de Arabidopsis proporcionó la confirmación del origen endosimbiótico del cloroplasto. [21] [22] También demostró el alcance significativo de la transferencia genética del ancestro cianobacteriano al genoma nuclear.
En la mayoría de las especies de plantas, el genoma del cloroplasto codifica aproximadamente 120 genes. [23] [24] Los genes codifican principalmente componentes centrales de la maquinaria fotosintética y factores involucrados en su expresión y ensamblaje. [25] En las especies de plantas terrestres, el conjunto de genes codificados por el genoma del cloroplasto está bastante conservado. Esto incluye cuatro ARN ribosómicos , aproximadamente 30 ARNt , 21 proteínas ribosómicas y 4 subunidades del complejo ARN polimerasa codificado por plástidos que están involucrados en la expresión de genes de plástidos. [25] La subunidad grande Rubisco y 28 proteínas tilacoides fotosintéticas están codificadas dentro del genoma del cloroplasto. [25]
Con el tiempo, muchas partes del genoma del cloroplasto se transfirieron al genoma nuclear del huésped, [7] [8] [26] un proceso llamado transferencia génica endosimbiótica . Como resultado, el genoma del cloroplasto se reduce considerablemente en comparación con el de las cianobacterias de vida libre. Los cloroplastos pueden contener entre 60 y 100 genes, mientras que las cianobacterias a menudo tienen más de 1500 genes en su genoma. [27] Los parásitos Pilostyles incluso han perdido sus genes de plástidos para ARNt . [28] Por el contrario, solo hay unos pocos casos conocidos en los que los genes se han transferido al cloroplasto desde varios donantes, incluidas las bacterias. [29] [30] [31]
La transferencia de genes endosimbióticos es la forma en que conocemos los cloroplastos perdidos en muchos linajes cromalveolados . Incluso si un cloroplasto finalmente se pierde, los genes que donó al núcleo del huésped anterior persisten, lo que proporciona evidencia de la existencia del cloroplasto perdido. Por ejemplo, mientras que las diatomeas (un heterocontofito ) ahora tienen un cloroplasto derivado de algas rojas , la presencia de muchos genes de algas verdes en el núcleo de la diatomea proporciona evidencia de que el ancestro de las diatomeas (probablemente el ancestro de todos los cromalveolados también) tuvo un cloroplasto derivado de algas verdes en algún momento, que posteriormente fue reemplazado por el cloroplasto rojo. [32]
En las plantas terrestres, entre el 11 y el 14 % del ADN de sus núcleos se puede rastrear hasta el cloroplasto, [33] hasta el 18 % en Arabidopsis , lo que corresponde a unos 4500 genes codificadores de proteínas. [34] Ha habido algunas transferencias recientes de genes del ADN del cloroplasto al genoma nuclear en plantas terrestres. [8]
De las aproximadamente tres mil proteínas que se encuentran en los cloroplastos, aproximadamente el 95% de ellas están codificadas por genes nucleares. Muchos de los complejos proteicos del cloroplasto consisten en subunidades tanto del genoma del cloroplasto como del genoma nuclear del huésped. Como resultado, la síntesis de proteínas debe coordinarse entre el cloroplasto y el núcleo. El cloroplasto está en su mayor parte bajo control nuclear, aunque los cloroplastos también pueden emitir señales que regulan la expresión genética en el núcleo, denominadas señalización retrógrada . [35]
La síntesis de proteínas en los cloroplastos depende de una ARN polimerasa codificada por el genoma del propio cloroplasto, que está relacionada con las ARN polimerasas que se encuentran en las bacterias. Los cloroplastos también contienen una misteriosa segunda ARN polimerasa que está codificada por el genoma nuclear de la planta. Las dos ARN polimerasas pueden reconocer y unirse a diferentes tipos de promotores dentro del genoma del cloroplasto. [36] Los ribosomas de los cloroplastos son similares a los ribosomas bacterianos. [37]
La edición de ARN es la inserción, eliminación y sustitución de nucleótidos en una transcripción de ARNm antes de su traducción a proteína. El entorno altamente oxidativo dentro de los cloroplastos aumenta la tasa de mutación, por lo que se necesitan reparaciones posteriores a la transcripción para conservar las secuencias funcionales. El editosoma del cloroplasto sustituye C -> U y U -> C en lugares muy específicos de la transcripción. Esto puede cambiar el codón de un aminoácido o restaurar un pseudogén no funcional agregando un codón de inicio AUG o eliminando un codón de terminación UAA prematuro. [38]
El editosoma reconoce y se une a la secuencia cis aguas arriba del sitio de edición. La distancia entre el sitio de unión y el sitio de edición varía según el gen y las proteínas involucradas en el editosoma. Cientos de proteínas PPR diferentes del genoma nuclear están involucradas en el proceso de edición del ARN. Estas proteínas consisten en aminoácidos repetidos de 35 meros, cuya secuencia determina el sitio de unión cis para la transcripción editada. [38]
Las plantas terrestres basales, como las hepáticas, los musgos y los helechos, tienen cientos de sitios de edición diferentes, mientras que las plantas con flores suelen tener entre treinta y cuarenta. Las plantas parásitas, como Epifagus virginiana, muestran una pérdida de la edición de ARN, lo que da lugar a una pérdida de la función de los genes de la fotosíntesis. [39]
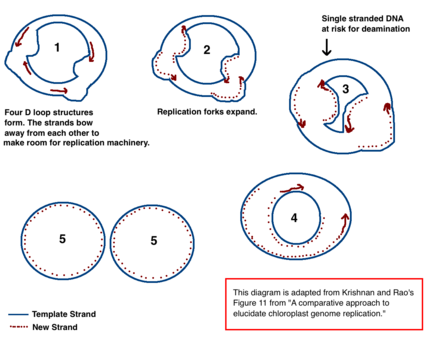
El mecanismo de replicación del ADN del cloroplasto (cpDNA) no se ha determinado de manera concluyente, pero se han propuesto dos modelos principales. Los científicos han intentado observar la replicación del cloroplasto mediante microscopía electrónica desde la década de 1970. [40] [41] Los resultados de los experimentos de microscopía llevaron a la idea de que el ADN del cloroplasto se replica utilizando un bucle de doble desplazamiento (D-loop). A medida que el D-loop se mueve a través del ADN circular, adopta una forma intermedia theta, también conocida como intermediario de replicación de Cairns, y completa la replicación con un mecanismo de círculo rodante. [40] [12] La replicación comienza en puntos de origen específicos. Se abren múltiples horquillas de replicación , lo que permite que la maquinaria de replicación replique el ADN. A medida que continúa la replicación, las horquillas crecen y finalmente convergen. Las nuevas estructuras de cpDNA se separan, creando cromosomas cpDNA hijos.
Además de los primeros experimentos de microscopía, este modelo también está respaldado por las cantidades de desaminación observadas en el cpDNA. [40] La desaminación ocurre cuando se pierde un grupo amino y es una mutación que a menudo resulta en cambios de bases. Cuando se desamina la adenina, se convierte en hipoxantina (H). La hipoxantina puede unirse a la citosina y, cuando se replica el par de bases HC, se convierte en un GC (por lo tanto, un cambio de base A → G). [42]

En el cpDNA, hay varios gradientes de desaminación A → G. El ADN se vuelve susceptible a eventos de desaminación cuando es monocatenario. Cuando se forman las horquillas de replicación, la hebra que no se copia es monocatenaria y, por lo tanto, corre el riesgo de desaminación A → G. Por lo tanto, los gradientes en la desaminación indican que las horquillas de replicación probablemente estaban presentes y la dirección en la que se abrieron inicialmente (el gradiente más alto es probablemente el más cercano al sitio de inicio porque fue monocatenario durante la mayor cantidad de tiempo). [40] Este mecanismo sigue siendo la teoría principal en la actualidad; sin embargo, una segunda teoría sugiere que la mayor parte del cpDNA es en realidad lineal y se replica a través de recombinación homóloga. Además, sostiene que solo una minoría del material genético se mantiene en cromosomas circulares, mientras que el resto está en estructuras ramificadas, lineales u otras estructuras complejas. [40] [12]
Uno de los principales modelos en competencia para el cpDNA afirma que la mayor parte del cpDNA es lineal y participa en estructuras de recombinación homóloga y replicación similares al bacteriófago T4 . [12] Se ha establecido que algunas plantas tienen cpDNA lineal, como el maíz, y que aún más contienen estructuras complejas que los científicos aún no comprenden; [12] sin embargo, la visión predominante hoy es que la mayor parte del cpDNA es circular. Cuando se realizaron los experimentos originales en cpDNA, los científicos notaron estructuras lineales; sin embargo, atribuyeron estas formas lineales a círculos rotos. [12] Si las estructuras ramificadas y complejas observadas en los experimentos con cpDNA son reales y no artefactos de ADN circular concatenado o círculos rotos, entonces un mecanismo de replicación de bucle D es insuficiente para explicar cómo se replicarían esas estructuras. [12] Al mismo tiempo, la recombinación homóloga no explica los múltiples gradientes A → G observados en los plastomas. [40] Esta deficiencia es una de las mayores de la teoría de la estructura lineal.
El movimiento de tantos genes del cloroplasto hacia el núcleo significa que muchas proteínas del cloroplasto que se suponía que debían traducirse en el cloroplasto ahora se sintetizan en el citoplasma. Esto significa que estas proteínas deben ser dirigidas de vuelta al cloroplasto y ser importadas a través de al menos dos membranas del cloroplasto. [43]
Curiosamente, alrededor de la mitad de los productos proteicos de los genes transferidos ni siquiera se dirigen de nuevo al cloroplasto. Muchos se convirtieron en exaptaciones , asumiendo nuevas funciones como participar en la división celular , el enrutamiento de proteínas e incluso la resistencia a las enfermedades . Unos pocos genes del cloroplasto encontraron nuevos hogares en el genoma mitocondrial ; la mayoría se convirtieron en pseudogenes no funcionales , aunque unos pocos genes de ARNt todavía funcionan en la mitocondria . [27] Algunos productos proteicos del ADN del cloroplasto transferidos se dirigen a la vía secretora [27] (aunque muchos plástidos secundarios están delimitados por una membrana más externa derivada de la membrana celular del huésped y, por lo tanto, topológicamente fuera de la célula, porque para llegar al cloroplasto desde el citosol , hay que cruzar la membrana celular , como si se dirigiera al espacio extracelular . En esos casos, las proteínas dirigidas al cloroplasto viajan inicialmente a lo largo de la vía secretora). [44]
Debido a que la célula que adquiere un cloroplasto ya tenía mitocondrias (y peroxisomas , y una membrana celular para la secreción), el nuevo huésped del cloroplasto tuvo que desarrollar un sistema de orientación de proteínas único para evitar que las proteínas del cloroplasto se enviaran al orgánulo equivocado . [43]
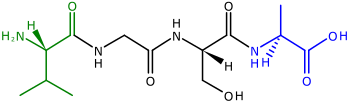
Los polipéptidos , precursores de las proteínas , son cadenas de aminoácidos . Los dos extremos de un polipéptido se denominan extremo N , o amino , y extremo C , o carboxilo . [45] Para muchas (pero no todas) [46] proteínas de cloroplasto codificadas por genes nucleares , se añaden péptidos de tránsito escindibles a los extremos N de los polipéptidos, que se utilizan para ayudar a dirigir el polipéptido al cloroplasto para su importación [43] [47] (los péptidos de tránsito N-terminales también se utilizan para dirigir polipéptidos a las mitocondrias de las plantas ). [48] Las secuencias de tránsito N-terminales también se denominan presecuencias [43] porque están ubicadas en el extremo "delantero" de un polipéptido: los ribosomas sintetizan polipéptidos desde el extremo N hasta el extremo C. [45]
Los péptidos de tránsito del cloroplasto presentan una enorme variación en longitud y secuencia de aminoácidos . [47] Pueden tener de 20 a 150 aminoácidos de longitud [43] —una longitud inusualmente larga, lo que sugiere que los péptidos de tránsito son en realidad colecciones de dominios con diferentes funciones. [47] Los péptidos de tránsito tienden a estar cargados positivamente , [43] ricos en aminoácidos hidroxilados como serina , treonina y prolina , y pobres en aminoácidos ácidos como ácido aspártico y ácido glutámico . [47] En una solución acuosa , la secuencia de tránsito forma una espiral aleatoria. [43]
Sin embargo, no todas las proteínas del cloroplasto incluyen un péptido de tránsito escindible en el extremo N. [43] Algunas incluyen la secuencia de tránsito dentro de la parte funcional de la proteína misma. [43] Algunas tienen su secuencia de tránsito adjunta a su extremo C. [ 49] La mayoría de los polipéptidos que carecen de secuencias de orientación en el extremo N son los que se envían a la membrana externa del cloroplasto , más al menos uno enviado a la membrana interna del cloroplasto . [43]
Después de que un polipéptido de cloroplasto se sintetiza en un ribosoma en el citosol , la energía del ATP se puede utilizar para fosforilar o agregar un grupo fosfato a muchos (pero no a todos) de ellos en sus secuencias de tránsito. [43] La serina y la treonina (ambas muy comunes en las secuencias de tránsito de cloroplastos, que representan el 20-30% de la secuencia) [50] son a menudo los aminoácidos que aceptan el grupo fosfato . [48] [50] La enzima que lleva a cabo la fosforilación es específica para los polipéptidos de cloroplasto e ignora los destinados a las mitocondrias o los peroxisomas . [50]
La fosforilación cambia la forma del polipéptido, [50] lo que facilita que las proteínas 14-3-3 se unan al polipéptido. [43] [51] En las plantas, las proteínas 14-3-3 solo se unen a las preproteínas del cloroplasto. [48] También se une a la proteína de choque térmico Hsp70 que evita que el polipéptido se pliegue prematuramente. [43] Esto es importante porque evita que las proteínas del cloroplasto asuman su forma activa y realicen sus funciones de cloroplasto en el lugar equivocado: el citosol . [48] [51] Al mismo tiempo, tienen que mantener la forma suficiente para que puedan ser reconocidas e importadas al cloroplasto. [48]
La proteína de choque térmico y las proteínas 14-3-3 juntas forman un complejo guía citosólico que facilita la importación del polipéptido del cloroplasto al cloroplasto. [43]
Alternativamente, si el péptido de tránsito de una preproteína de cloroplasto no está fosforilado, una preproteína de cloroplasto puede unirse a una proteína de choque térmico o a Toc159. Estos complejos pueden unirse al complejo TOC en la membrana externa del cloroplasto utilizando energía GTP . [43]
El complejo TOC , o translocón en la membrana externa del cloroplasto , es un conjunto de proteínas que importan preproteínas a través de la envoltura externa del cloroplasto . Se han identificado cinco subunidades del complejo TOC: dos proteínas de unión a GTP Toc34 y Toc159, el túnel de importación de proteínas Toc75, más las proteínas Toc64 [43] y Toc12. [46]
Las tres primeras proteínas forman un complejo central que consta de un Toc159, cuatro o cinco Toc34 y cuatro Toc75 que forman cuatro agujeros en un disco de 13 nanómetros de diámetro. El complejo central completo pesa alrededor de 500 kilodaltons . Las otras dos proteínas, Toc64 y Toc12, están asociadas con el complejo central pero no forman parte de él. [46]
![Toc34 de una planta de guisante. Toc34 tiene tres moléculas casi idénticas (mostradas en tonos de verde ligeramente diferentes), cada una de las cuales forma un dímero con una de sus moléculas adyacentes. Parte de un sitio de unión de la molécula GDP está resaltada en rosa.[52]](http://upload.wikimedia.org/wikipedia/commons/thumb/e/e4/TOC34.png/350px-TOC34.png)
Toc34 es una proteína integral en la membrana externa del cloroplasto que está anclada a ella por su cola C-terminal hidrofóbica [53] . [43] [51] Sin embargo, la mayor parte de la proteína, incluido su gran dominio de unión al trifosfato de guanosina (GTP), se proyecta hacia el estroma. [51]
El trabajo de Toc34 es capturar algunas preproteínas de cloroplasto en el citosol y entregárselas al resto del complejo TOC. [43] Cuando GTP , una molécula de energía similar al ATP se une a Toc34, la proteína se vuelve mucho más capaz de unirse a muchas preproteínas de cloroplasto en el citosol . [43] La presencia de la preproteína de cloroplasto hace que Toc34 rompa GTP en guanosina difosfato (GDP) y fosfato inorgánico . Esta pérdida de GTP hace que la proteína Toc34 libere la preproteína de cloroplasto, entregándola a la siguiente proteína TOC. [43] Toc34 luego libera la molécula de GDP agotada, probablemente con la ayuda de un factor de intercambio de GDP desconocido . Un dominio de Toc159 podría ser el factor de intercambio que lleva a cabo la eliminación de GDP. La proteína Toc34 puede entonces tomar otra molécula de GTP y comenzar el ciclo nuevamente. [43]
La Toc34 se puede desactivar mediante fosforilación . Una proteína quinasa que se desplaza por la membrana externa del cloroplasto puede usar ATP para agregar un grupo fosfato a la proteína Toc34, impidiéndole recibir otra molécula de GTP , inhibiendo así su actividad. Esto podría proporcionar una forma de regular la importación de proteínas a los cloroplastos. [43] [51]
Arabidopsis thaliana tiene dosproteínas homólogas , AtToc33 y AtToc34 ( At significa A rabidopsis t haliana ), [43] [51] que son aproximadamente un 60% idénticas en secuencia de aminoácidos a Toc34 en guisantes (llamado ps Toc34). [51] AtToc33 es el más común en Arabidopsis , [51] y es el análogo funcional de Toc34 porque puede desactivarse por fosforilación. AtToc34, por otro lado, no puede ser fosforilada. [43] [51]
Toc159 es otra subunidad de TOC que se une a GTP , como Toc34. Toc159 tiene tres dominios . En el extremo N-terminal está el dominio A, que es rico en aminoácidos ácidos y ocupa aproximadamente la mitad de la longitud de la proteína. [43] [53] El dominio A a menudo se escinde , dejando un fragmento de 86 kilodalton llamado Toc86. [53] En el medio está su dominio de unión a GTP , que es muy similar al dominio de unión a GTP homólogo en Toc34. [43] [53] En el extremo C-terminal está el dominio M hidrófilo , [43] que ancla la proteína a la membrana externa del cloroplasto. [53]
Probablemente, Toc159 funciona de forma muy similar a Toc34, ya que reconoce proteínas en el citosol mediante GTP . Puede regularse mediante fosforilación , pero mediante una proteína quinasa diferente a la que fosforila Toc34. [46] Su dominio M forma parte del túnel por el que viajan las preproteínas del cloroplasto y parece proporcionar la fuerza que las empuja a través de él, utilizando la energía del GTP . [43]
El Toc159 no siempre se encuentra como parte del complejo TOC; también se lo ha encontrado disuelto en el citosol . Esto sugiere que podría actuar como una lanzadera que encuentra preproteínas del cloroplasto en el citosol y las lleva de regreso al complejo TOC. Sin embargo, no hay mucha evidencia directa de este comportamiento. [43]
Se ha encontrado una familia de proteínas Toc159, Toc159, Toc132, Toc120 y Toc90 en Arabidopsis thaliana . Varían en la longitud de sus dominios A, que han desaparecido por completo en Toc90. Toc132, Toc120 y Toc90 parecen tener funciones especializadas en la importación de elementos como preproteínas no fotosintéticas y no pueden reemplazar a Toc159. [43]

Toc75 es la proteína más abundante en la envoltura externa del cloroplasto. Es un tubo transmembrana que forma la mayor parte del poro del TOC. Toc75 es un canal de barril β revestido por 16 láminas β-plegadas . [43] El orificio que forma tiene aproximadamente 2,5 nanómetros de ancho en los extremos y se encoge a aproximadamente 1,4–1,6 nanómetros de diámetro en su punto más estrecho, lo suficientemente ancho como para permitir que las preproteínas del cloroplasto parcialmente plegadas pasen a través de él. [43]
Toc75 también puede unirse a las preproteínas del cloroplasto, pero es mucho peor que Toc34 o Toc159. [43]
Arabidopsis thaliana tiene múltiples isoformas de Toc75 que reciben su nombre de las posiciones cromosómicas de los genes que las codifican. AtToc75 III es la más abundante de ellas. [43]
El translocón TIC , o translocón en la membrana interna del cloroplasto [ 43 ] es otro complejo proteico que importa proteínas a través de la envoltura interna del cloroplasto . Las cadenas polipeptídicas del cloroplasto probablemente viajan a menudo a través de los dos complejos al mismo tiempo, pero el complejo TIC también puede recuperar preproteínas perdidas en el espacio intermembrana . [43]
Al igual que el translocón TOC , el translocón TIC tiene un gran complejo central rodeado por algunas proteínas periféricas asociadas de forma vaga como Tic110, Tic40 y Tic21. [54] El complejo central pesa alrededor de un millón de daltons y contiene Tic214, Tic100, Tic56 y Tic20 I, posiblemente tres de cada uno. [54]
Tic20 es una proteína integral que se cree que tiene cuatro hélices α transmembrana . [43] Se encuentra en el complejo TIC de 1 millón de dalton . [54] Debido a que es similar a los transportadores de aminoácidos bacterianos y a la proteína de importación mitocondrial Tim17 [43] ( translocasa en la membrana mitocondrial interna ), [55] se ha propuesto que es parte del canal de importación de TIC. [43] Sin embargo , no hay evidencia in vitro de esto. [43] En Arabidopsis thaliana , se sabe que por aproximadamente cada cinco proteínas Toc75 en la membrana externa del cloroplasto, hay dos proteínas Tic20 I (la forma principal de Tic20 en Arabidopsis ) en la membrana interna del cloroplasto. [54]
A diferencia de Tic214, Tic100 o Tic56, Tic20 tiene parientes homólogos en las cianobacterias y en casi todos los linajes de cloroplastos, lo que sugiere que evolucionó antes de la primera endosimbiosis de cloroplastos. Tic214, Tic100 y Tic56 son exclusivos de los cloroplastos cloroplastídicos , lo que sugiere que evolucionaron más tarde. [54]
Tic214 es otra proteína del complejo central TIC, llamada así porque pesa poco menos de 214 kilodaltons . Tiene 1786 aminoácidos de longitud y se cree que tiene seis dominios transmembrana en su extremo N-terminal . Tic214 es notable por estar codificada por el ADN del cloroplasto, más específicamente el primer marco de lectura abierto ycf1 . Tic214 y Tic20 juntos probablemente forman la parte del complejo TIC de un millón de daltons que se extiende por toda la membrana . Tic20 está enterrado dentro del complejo mientras que Tic214 está expuesto en ambos lados de la membrana interna del cloroplasto . [54]
Tic100 es una proteína codificada nuclearmente que tiene 871 aminoácidos de longitud. Los 871 aminoácidos en conjunto pesan un poco menos de 100 mil daltons , y dado que la proteína madura probablemente no pierde ningún aminoácido cuando se importa al cloroplasto (no tiene péptido de tránsito escindible ), se la denominó Tic100. Tic100 se encuentra en los bordes del complejo de 1 millón de daltons en el lado que mira hacia el espacio intermembrana del cloroplasto . [54]
La Tic56 también es una proteína codificada nuclearmente . La preproteína que codifica su gen tiene 527 aminoácidos de longitud y pesa cerca de 62 mil daltons ; la forma madura probablemente sufre un procesamiento que la reduce a algo que pesa 56 mil daltons cuando se importa al cloroplasto. La Tic56 está en gran parte incrustada dentro del complejo de 1 millón de daltons. [54]
Tic56 y Tic100 están muy conservadas entre las plantas terrestres, pero no se parecen a ninguna proteína cuya función se conozca. Ninguna de ellas tiene dominios transmembrana . [54]
número de copias de ctDNA por cloroplasto.