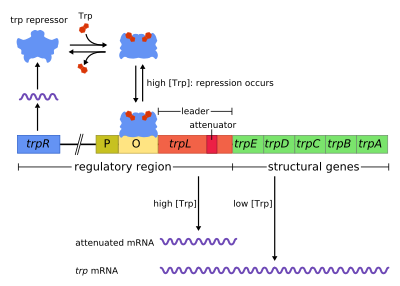
El operón trp es un grupo de genes que se transcriben juntos y codifican las enzimas que producen el aminoácido triptófano en las bacterias. El operón trp se caracterizó por primera vez en Escherichia coli y , desde entonces, se ha descubierto en muchas otras bacterias. [1] El operón está regulado de modo que, cuando el triptófano está presente en el medio ambiente, los genes para la síntesis de triptófano se reprimen.
El operón trp contiene cinco genes estructurales: trpE , trpD , trpC , trpB y trpA , que codifican las enzimas necesarias para sintetizar triptófano. También contiene un gen regulador represor llamado trpR . Cuando hay triptófano, la proteína trpR se une al operador , bloqueando la transcripción del operón trp por la ARN polimerasa.
Este operón es un ejemplo de regulación negativa reprimible de la expresión génica. La proteína represora se une al operador en presencia de triptófano (reprimiendo la transcripción ) y se libera del operón cuando el triptófano está ausente (permitiendo que la transcripción continúe). El operón trp utiliza además la atenuación para controlar la expresión del operón , un segundo mecanismo de control de retroalimentación negativa .
El operón trp ha sido bien estudiado y se utiliza comúnmente como ejemplo de regulación genética en bacterias junto con el operón lac .
El operón trp contiene cinco genes estructurales. Las funciones de sus productos son:

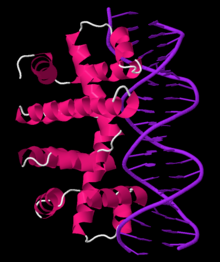
El operón funciona mediante un mecanismo de retroalimentación reprimible negativo. El represor para el operón trp es producido aguas arriba por el gen trpR, que se expresa constitutivamente a un nivel bajo. Los monómeros trpR sintetizados se asocian en dímeros. Cuando el triptófano está presente, estos dímeros represores de triptófano se unen al triptófano, lo que provoca un cambio en la conformación del represor, lo que permite que el represor se una al operador . Esto evita que la ARN polimerasa se una y transcriba el operón, por lo que el triptófano no se produce a partir de su precursor. Cuando el triptófano no está presente, el represor está en su conformación inactiva y no puede unirse a la región del operador, por lo que el represor no inhibe la transcripción.

La atenuación es un segundo mecanismo de retroalimentación negativa en el operón trp . El sistema de represión se dirige a la concentración intracelular de trp, mientras que la atenuación responde a la concentración de ARNt cargado trp . [2] Por lo tanto, el represor trpR disminuye la expresión génica alterando el inicio de la transcripción, mientras que la atenuación lo hace alterando el proceso de transcripción que ya está en curso. [2] Mientras que el represor TrpR disminuye la transcripción en un factor de 70, la atenuación puede disminuirla aún más en un factor de 10, lo que permite una represión acumulada de aproximadamente 700 veces. [3] La atenuación es posible gracias al hecho de que en los procariotas (que no tienen núcleo ), los ribosomas comienzan a traducir el ARNm mientras la ARN polimerasa todavía está transcribiendo la secuencia de ADN. Esto permite que el proceso de traducción afecte directamente a la transcripción del operón.
Al comienzo de los genes transcritos del operón trp hay una secuencia de al menos 130 nucleótidos denominada transcripción líder (trpL; P0AD92 ). [4] Lee y Yanofsky (1977) encontraron que la eficiencia de atenuación está correlacionada con la estabilidad de una estructura secundaria incrustada en trpL, [5] y las 2 horquillas constituyentes de la estructura del terminador fueron dilucidadas posteriormente por Oxender et al. (1979). [6] Esta transcripción incluye cuatro secuencias cortas designadas 1–4, cada una de las cuales es parcialmente complementaria a la siguiente. Por lo tanto, se pueden formar tres estructuras secundarias distintas ( horquillas ): 1–2, 2–3 o 3–4. La hibridación de las secuencias 1 y 2 para formar la estructura 1-2 es poco frecuente porque la ARN polimerasa espera a que se adhiera un ribosoma antes de continuar la transcripción más allá de la secuencia 1; sin embargo, si se formara la horquilla 1-2, evitaría la formación de la estructura 2-3 (pero no la 3-4). La formación de un bucle de horquilla entre las secuencias 2-3 evita la formación de bucles de horquilla entre 1-2 y 3-4. La estructura 3-4 es una secuencia de terminación de la transcripción (abundante en G/C e inmediatamente seguida por varios residuos de uracilo), una vez que se forma, la ARN polimerasa se disociará del ADN y no podrá ocurrir la transcripción de los genes estructurales del operón (consulte a continuación una explicación más detallada). La importancia funcional de la segunda horquilla para la terminación de la transcripción se ilustra por la frecuencia reducida de terminación de la transcripción observada en experimentos que desestabilizaron el apareamiento central G+C de esta horquilla. [5] [7] [8] [9]
Parte de la transcripción líder codifica un polipéptido corto de 14 aminoácidos, denominado péptido líder. Este péptido contiene dos residuos de triptófano adyacentes, lo cual es inusual, ya que el triptófano es un aminoácido bastante poco común (aproximadamente uno de cada cien residuos en una proteína típica de E. coli es triptófano). La hebra 1 en trpL abarca la región que codifica los residuos finales del péptido líder: Trp, Trp, Arg, Thr, Ser; [2] se observa conservación en estos 5 codones, mientras que la mutación de los codones anteriores no altera la expresión del operón. [2] [10] [11] [12] Si el ribosoma intenta traducir este péptido mientras los niveles de triptófano en la célula son bajos, se detendrá en cualquiera de los dos codones trp. Mientras está detenido, el ribosoma protege físicamente la secuencia 1 de la transcripción, evitando la formación de la estructura secundaria 1-2. La secuencia 2 queda entonces libre para hibridarse con la secuencia 3 para formar la estructura 2-3, que luego impide la formación de la horquilla de terminación 3-4, razón por la cual la estructura 2-3 se denomina horquilla anti-terminación. En presencia de la estructura 2-3, la ARN polimerasa queda libre para continuar transcribiendo el operón. El análisis mutacional y los estudios que involucran oligonucleótidos complementarios demuestran que la estabilidad de la estructura 2-3 corresponde al nivel de expresión del operón. [10] [13] [14] [15] Si los niveles de triptófano en la célula son altos, el ribosoma traducirá todo el péptido líder sin interrupción y solo se detendrá durante la terminación de la traducción en el codón de terminación . En este punto, el ribosoma protege físicamente ambas secuencias 1 y 2. Las secuencias 3 y 4 quedan así libres para formar la estructura 3-4 que termina la transcripción. Esta estructura de terminación se forma cuando ningún ribosoma se detiene en la proximidad del tándem Trp (es decir, codón Trp o Arg): o bien el péptido líder no se traduce o la traducción procede sin problemas a lo largo de la cadena 1 con abundante ARNt trp cargado. [2] [10] Además, se propone que el ribosoma solo bloquea unos 10 nts corriente abajo, por lo que el estancamiento del ribosoma en Gly corriente arriba o más abajo en Thr no parece afectar la formación de la horquilla de terminación. [2] [10] El resultado final es que el operón se transcribirá solo cuando el triptófano no esté disponible para el ribosoma, mientras que la transcripción trpL se expresa de forma constitutiva.
Este mecanismo de atenuación está respaldado experimentalmente. En primer lugar, se ha demostrado directamente que la traducción del péptido líder y el bloqueo ribosómico son necesarios para inhibir la terminación de la transcripción. [13] Además, el análisis mutacional que desestabiliza o altera el apareamiento de bases de la horquilla antiterminadora da como resultado una mayor terminación de varios pliegues; en consonancia con el modelo de atenuación, esta mutación no logra aliviar la atenuación incluso con Trp privado de proteínas. [10] [13] Por el contrario, los oligonucleótidos complementarios que se dirigen a la hebra 1 aumentan la expresión del operón al promover la formación del antiterminador. [10] [14] Además, en el operón histidina, la mutación compensatoria muestra que la capacidad de apareamiento de las hebras 2-3 es más importante que su secuencia primaria para inhibir la atenuación. [10] [15]
En la atenuación, el punto en el que el ribosoma traductor se detiene determina si se formará la horquilla de terminación. [10] Para que la polimerasa transcriptora capture simultáneamente la estructura alternativa, la escala de tiempo de la modulación estructural debe ser comparable a la de la transcripción. [2] Para garantizar que el ribosoma se una y comience la traducción de la transcripción líder inmediatamente después de su síntesis, existe un sitio de pausa en la secuencia trpL. Al llegar a este sitio, la ARN polimerasa detiene la transcripción y aparentemente espera a que comience la traducción. Este mecanismo permite la sincronización de la transcripción y la traducción, un elemento clave en la atenuación.
Un mecanismo de atenuación similar regula la síntesis de histidina , fenilalanina y treonina .
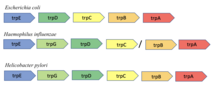
La disposición del operón trp en E. coli y Bacillus subtilis difiere. Hay 5 genes estructurales en E. coli que se encuentran bajo una sola unidad transcripcional. En Bacillus subtilis , hay 6 genes estructurales que están situados dentro de un supraoperón. Tres de estos genes se encuentran aguas arriba, mientras que los otros tres genes se encuentran aguas abajo del operón trp. [16] Hay un séptimo gen en el operón de Bacillus subtilis llamado trpG o pabA que es responsable de la síntesis de proteínas de triptófano y folato . [17] La regulación de los operones trp en ambos organismos depende de la cantidad de trp presente en la célula. Sin embargo, la regulación primaria de la biosíntesis de triptófano en B. subtilis es a través de la atenuación, en lugar de la represión, de la transcripción. [18] En B. subtilis , el triptófano se une a la proteína de atenuación de unión al ARN activado por triptófano de once subunidades (TRAP), que activa la capacidad de TRAP de unirse al ARN líder trp. [19] [20] La unión de TRAP activado por trp al ARN líder da como resultado la formación de una estructura terminadora que causa la terminación de la transcripción. [18] Además, el TRAP activado inhibe el inicio de la traducción de los genes trpP, trpE, trpG y ycbK. El gen trpP desempeña un papel en el transporte de trp, mientras que el gen trpG se utiliza en el operón de folato y el gen ycbK está involucrado en la síntesis de una proteína de eflujo. La proteína TRAP activada está regulada por una proteína anti-TRAP y la síntesis de AT. AT puede inactivar TRAP para reducir la transcripción de triptófano. [21]
{{cite journal}}: CS1 maint: DOI inactive as of November 2024 (link)