La enzima argininosuccinato liasa (EC 4.3.2.1, ASL , argininosuccinasa ; nombre sistemático 2-( N ω - L -arginino)succinato arginina-liasa (formadora de fumarato) ) cataliza la descomposición reversible del argininosuccinato :
Ubicada en el citosol del hígado, es la cuarta enzima del ciclo de la urea e involucrada en la biosíntesis de arginina en todas las especies y la producción de urea en especies ureotélicas . [2] Las mutaciones que resultan en una baja actividad de la enzima aumentan los niveles de urea en el cuerpo y dan lugar a diversos efectos secundarios.
El gen ASL está ubicado en el cromosoma 7 entre el centrómero (unión del brazo largo y corto) y el brazo largo (q) en la posición 11.2, desde el par de bases 64.984.963 hasta el par de bases 65.002.090.
El ASL está relacionado con la complementación intragénica . [3] [4] [5]
La ASL está compuesta por cuatro monómeros idénticos; cada monómero consiste en una sola cadena polipeptídica de entre 49 y 52 kDa, [6] entre 196 y 208 kDa para toda la enzima tetramérica. Cada monómero tiene tres regiones altamente conservadas alejadas una de la otra, pero estas regiones se agrupan en el tetrámero para formar cuatro sitios activos. Por lo tanto, cada homotetrámero de ASL tiene cuatro sitios activos para catalizar la descomposición del argininosuccinato.
Cada monómero del homotetrámero ASL está compuesto por tres dominios estructurales; los tres son principalmente helicoidales alfa. Los dominios 1 y 3 son similares en estructura, ya que ambos consisten en motivos de hélice-giro-hélice. El dominio 1 del monómero contiene el extremo amino. El dominio 2 contiene una pequeña lámina beta, nueve hélices alfa y el extremo carboxilo. Tres de las nueve hélices alfa de un monómero participan principalmente en interacciones hidrofóbicas con otro monómero para formar un dímero. Luego, dos dímeros se asocian mediante una hélice alfa, uno de cada monómero, para formar un núcleo central de 20 hélices. La asociación de los cuatro monómeros permite la actividad catalítica en cada posible sitio activo. [4]
Varias copias de un polipéptido codificado por un gen a menudo pueden formar un agregado denominado multímero. Cuando un multímero se forma a partir de polipéptidos producidos por dos alelos mutantes diferentes de un gen en particular, el multímero mixto puede exhibir una mayor actividad funcional que los multímeros no mezclados formados por cada uno de los mutantes solos. Cuando un multímero mixto muestra una mayor funcionalidad en relación con los multímeros no mezclados, el fenómeno se denomina complementación intragénica . En los seres humanos, la ASL es una proteína multímera (tetrámera). Un trastorno de la ASL en los seres humanos puede surgir de mutaciones en el gen ASL, en particular mutaciones que afectan el sitio activo de la proteína multímera mutante. El trastorno de la ASL se asocia con una considerable heterogeneidad clínica y genética que se considera que refleja la extensa complementación intragénica que se produce entre pacientes individuales. [3] [4] [5]
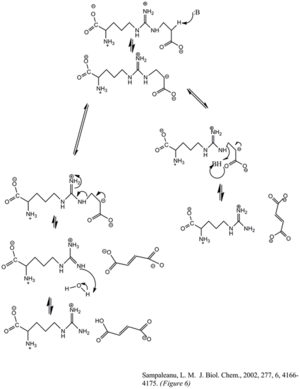

La escisión enzimática del argininosuccinato, para formar fumarato y arginina, ocurre a través de una reacción de eliminación de E1cb. La base inicia la reacción desprotonando el carbono adyacente a la arginina, o grupo saliente. Estudios mutagénicos recientes de homólogos de ASL han demostrado que la histidina 162 o la treonina 161 de ASL son responsables de la abstracción de protones del Cβ, ya sea directa o indirectamente a través de una molécula de agua. [6] Se cree que la lisina 289 estabiliza el intermediario carbanión cargado negativamente. Aunque no hay consenso sobre el ácido catalítico que dona el protón al grupo funcional imina del producto arginina, algunos estudios de mutagénesis muestran que la serina 283 puede estar involucrada. [6]
El amoniaco (NH3 ) es una sustancia tóxica para muchos organismos aeróbicos y debe ser excretado. Algunos organismos acuáticos liberan la toxina directamente en su entorno, mientras que otras especies ureotélicas deben convertir sus desechos nitrogenados tóxicos en componentes no tóxicos, como el ácido úrico o la urea, a través de una serie de pasos catalizados mejor conocidos como el ciclo de la urea. La ASL cataliza el cuarto paso del ciclo, siguiendo la acción de la argininosuccinato sintetasa (ASS) en el citosol del hígado. Mientras que la ASS cataliza la formación de argininosuccinato a partir de citrulina y aspartato, la ASL descompone el argininosuccinato recién formado en L-arginina y fumarato. La L-arginina continúa a través del ciclo de la urea para formar urea y ornitina, mientras que el fumarato puede ingresar al ciclo del ácido cítrico. [7]
La ASL, la δ- cristalina , la fumarasa de clase II, la aspartasa, la adenilosuccinasa liasa y la enzima lactonizante 3-carboxi-cis y cis-muconato son todas miembros de la misma superfamilia homotetramérica de enzimas, en la que la mayoría cataliza el mismo tipo de reacciones de eliminación donde se rompe un enlace CO o CN y se libera fumarato como producto. Las δ-cristalinas son las principales proteínas estructurales solubles en agua del cristalino del ojo de la mayoría de las aves, reptiles y algunos otros vertebrados. [4]
Dentro de la superfamilia, la ASL es la que está más estrechamente relacionada con la δ-cristalina en cuanto a la secuencia de aminoácidos y la estructura de plegamiento de la proteína. Existen dos isoformas de la δ-cristalina, δI y δII. Estas dos isoformas conservan el 69% y el 71% de la secuencia de aminoácidos de la ASL, respectivamente, pero solo la isoforma δII conserva la misma actividad enzimática que la ASL. Las similitudes han llevado a los investigadores a creer que estas cristalinas han evolucionado a partir del reclutamiento en el cristalino de enzimas metabólicas preexistentes, como la ASL, mediante un proceso denominado "intercambio de genes". El mismo producto génico funciona como cristalina del cristalino y como enzima en otros tejidos no oculares. Los estudios comparativos de las δ-cristalinas han sido beneficiosos para comprender el mecanismo enzimático de la reacción de la ASL. [8]
Las mutaciones en el gen humano ASL causan aciduria argininosuccínica, un trastorno autosómico recesivo poco frecuente, y dan lugar a deficiencias del ciclo de la urea. La argininosuccinato liasa es una enzima intermediaria en la vía de síntesis de la urea y su función es imperativa para la continuación del ciclo. Una enzima que no funciona da lugar a la acumulación de amoníaco, argininosuccinato y citrulina en la sangre de los pacientes, y el argininosuccinato se excreta en la orina. [9] Otros síntomas resultantes incluyen letargo, vómitos, hipotermia, hiperventilación, hepatomegalia y encefalopatía progresiva en pacientes infantiles, y crecimiento anormal del cabello, fibrosis hepática, vómitos episódicos, retraso del crecimiento y del desarrollo, [9] en pacientes que experimentan el trastorno más adelante en la infancia.
El ASL es una enzima clave en la conversión de amoníaco en urea a través del ciclo de la urea. El amoníaco se acumula hasta alcanzar niveles tóxicos, lo que produce hiperamonemia. [10] El amoníaco es tóxico en parte porque afecta al sistema nervioso. Hay evidencia bioquímica que muestra que los aumentos de amoníaco pueden inhibir la glutaminasa y, por lo tanto, limitar la tasa de síntesis de neurotransmisores como el glutamato, [11] lo que puede explicar el retraso del desarrollo en pacientes con aciduria argininosuccínica.
Una mutación en pacientes con aciduria argininosuccínica ocurre cuando la glutamina 286 se transforma en arginina. La enzima ahora tiene una arginina con carga positiva en lugar de una glutamina con carga neutra y los estudios sugieren que este cambio puede obstaculizar estéricamente y/o electrostáticamente un cambio conformacional necesario para la catálisis.