PubMed es una base de datos gratuita que incluye principalmente la base de datos MEDLINE de referencias y resúmenes sobre ciencias biológicas y temas biomédicos . La Biblioteca Nacional de Medicina de los Estados Unidos (NLM) en los Institutos Nacionales de Salud mantiene la base de datos como parte del sistema de recuperación de información Entrez . [1]
De 1971 a 1997, el acceso en línea a la base de datos MEDLINE se había realizado principalmente a través de instalaciones institucionales, como bibliotecas universitarias . [2] PubMed, lanzado por primera vez en enero de 1996, marcó el comienzo de la era de las búsquedas en MEDLINE privadas, gratuitas, basadas en el hogar y la oficina. [3] El sistema PubMed se ofreció gratuitamente al público a partir de junio de 1997. [2]
Además de MEDLINE, PubMed brinda acceso a:
Muchos registros de PubMed contienen enlaces a artículos de texto completo, algunos de los cuales están disponibles gratuitamente, a menudo en PubMed Central [5] y espejos locales, como Europe PubMed Central . [6]
La información sobre las revistas indexadas en MEDLINE y disponibles a través de PubMed se encuentra en el Catálogo NLM. [7]
Al 23 de mayo de 2023 [actualizar], PubMed tiene más de 35 millones de citas y resúmenes que se remontan a 1966, selectivamente al año 1865 y muy selectivamente a 1809. A la misma fecha [actualizar], 24,6 millones de registros de PubMed se enumeran con sus resúmenes, y 26,8 millones de registros tienen enlaces a versiones de texto completo (de los cuales 10,9 millones de artículos están disponibles en texto completo de forma gratuita). [8] Durante los últimos 10 años (que finalizaron el 31 de diciembre de 2019), se agregó un promedio de casi un millón de registros nuevos cada año.
En 2016, NLM cambió el sistema de indexación para que los editores puedan corregir directamente errores tipográficos y errores en los artículos indexados de PubMed. [9]
Se ha informado que PubMed incluye algunos artículos publicados en revistas depredadoras. Las políticas de MEDLINE y PubMed para la selección de revistas para su inclusión en bases de datos son ligeramente diferentes. Las debilidades en los criterios y procedimientos para indexar revistas en PubMed Central pueden permitir que publicaciones de revistas depredadoras se filtren en PubMed. [10] La Biblioteca Nacional de Medicina respondió que se pueden incluir artículos de revistas individuales en PMC para respaldar las políticas de acceso público de los financiadores de investigaciones y que las políticas rigurosas sobre revistas y editores garantizan la integridad de las bases de datos de literatura de la NLM. [11]
En octubre de 2009 se lanzó una nueva interfaz de PubMed que fomentó el uso de formulaciones de búsqueda rápidas similares a las de Google; también se han descrito como búsquedas de "telegramas". [12] De forma predeterminada, los resultados se ordenan por Más reciente, pero esto se puede cambiar a Mejor coincidencia, Fecha de publicación, Primer autor, Último autor, Revista o Título. [13]
El diseño y el dominio del sitio web PubMed se actualizaron en enero de 2020 y pasaron a ser predeterminados el 15 de mayo de 2020, con las funciones nuevas y actualizadas. [14] Hubo una reacción crítica por parte de muchos investigadores que utilizan con frecuencia el sitio. [15]
Se puede acceder a PubMed/MEDLINE a través de dispositivos portátiles, utilizando, por ejemplo, la opción "PICO" (para preguntas clínicas específicas) creada por la NLM. [16] También está disponible una opción "PubMed Mobile", que brinda acceso a una versión simplificada y compatible con dispositivos móviles de PubMed. [17]
Se pueden realizar búsquedas simples en PubMed ingresando aspectos clave de un tema en la ventana de búsqueda de PubMed.
PubMed traduce esta formulación de búsqueda inicial y agrega automáticamente nombres de campos, términos MeSH (encabezados de temas médicos) relevantes, sinónimos, operadores booleanos y 'anida' los términos resultantes de manera apropiada, mejorando significativamente la formulación de búsqueda, en particular combinando rutinariamente (usando el método OR operador) palabras de texto y términos MeSH. [ cita necesaria ]
Los ejemplos dados en un tutorial de PubMed [18] demuestran cómo funciona este proceso automático:
Causas El sonambulismo se traduce como ("etiología"[Subtítulo] O "etiología"[Todos los campos] O "causas"[Todos los campos] O "causalidad"[Términos MeSH] O "causalidad"[Todos los campos]) Y ( "sonambulismo "[Términos MeSH] O "sonambulismo"[Todos los campos] O ("dormir"[Todos los campos] Y "caminar"[Todos los campos]) O "sonambulismo"[Todos los campos])
Asimismo,
soft Attack Aspirin Prevention se traduce como ("infarto de miocardio"[Términos MeSH] O ("miocardio"[Todos los campos] Y "infarto"[Todos los campos]) O "infarto de miocardio"[Todos los campos] O ("corazón"[Todos Campos] Y "ataque"[Todos los campos]) O "ataque cardíaco"[Todos los campos]) Y ("aspirina"[Términos MeSH] O "aspirina"[Todos los campos]) Y ("prevención y control"[Subtítulo] O ("prevención"[Todos los campos] AND "control"[Todos los campos]) O "prevención y control"[Todos los campos] O "prevención"[Todos los campos])
Para realizar búsquedas óptimas en PubMed, es necesario comprender su componente principal, MEDLINE, y especialmente el vocabulario controlado MeSH (Medical Subject Headings) utilizado para indexar artículos de MEDLINE. También pueden requerir estrategias de búsqueda complejas, uso de nombres de campos (etiquetas), uso adecuado de límites y otras características; Los bibliotecarios de referencia y los especialistas en búsqueda ofrecen servicios de búsqueda. [19] [20]
La búsqueda en la ventana de búsqueda de PubMed sólo se recomienda para la búsqueda de temas inequívocos o nuevas intervenciones que aún no tengan un encabezado MeSH creado, así como para la búsqueda de marcas comerciales de medicamentos y nombres propios. También es útil cuando no existe un título adecuado o el descriptor representa un aspecto parcial. La búsqueda utilizando el diccionario de sinónimos MeSH es más precisa y arrojará menos resultados irrelevantes. Además, evita el inconveniente de la búsqueda de texto libre, en la que hay que tener en cuenta la ortografía, las diferencias singular/plural o abreviadas. Por otro lado, no se encontrarán los artículos incorporados más recientemente a la base de datos a los que aún no se les hayan asignado descriptores. Por lo tanto, para garantizar una búsqueda exhaustiva, se debe utilizar una combinación de encabezados de idiomas controlados y términos de texto libre. [21]
Cuando se indexa un artículo de revista, se extraen y almacenan numerosos parámetros del artículo como información estructurada. Dichos parámetros son: tipo de artículo (términos MeSH, por ejemplo, "ensayo clínico"), identificadores secundarios (términos MeSH), idioma, país de la revista o historial de publicación (fecha de publicación electrónica, fecha de publicación de la revista impresa).
El parámetro tipo de publicación permite realizar búsquedas por tipo de publicación , incluidos informes de diversos tipos de investigaciones clínicas. [22]
Desde julio de 2005, el proceso de indexación de artículos de MEDLINE extrae identificadores del resumen del artículo y los coloca en un campo llamado Identificador secundario (SI). El campo de identificador secundario sirve para almacenar números de acceso a varias bases de datos de datos de secuencias moleculares, expresión genética o compuestos químicos e identificaciones de ensayos clínicos. Para los ensayos clínicos, PubMed extrae los ID de los ensayos para los dos registros de ensayos más grandes: ClinicalTrials.gov (identificador NCT) y el Registro de números de ensayos controlados aleatorios estándar internacional (identificador IRCTN). [23]
Se puede marcar una referencia que se considere particularmente relevante y se pueden identificar "artículos relacionados". Si es relevante, se pueden seleccionar varios estudios y se pueden generar artículos relacionados con todos ellos (en PubMed o cualquiera de las otras bases de datos de NCBI Entrez) utilizando la opción "Buscar datos relacionados". Los artículos relacionados se enumeran en orden de "relación". Para crear estas listas de artículos relacionados, PubMed compara las palabras del título y el resumen de cada cita, así como los títulos MeSH asignados, utilizando un potente algoritmo ponderado por palabras. [24] Se ha considerado que la función 'artículos relacionados' es tan precisa que los autores de un artículo sugirieron que se puede utilizar en lugar de una búsqueda completa. [25]
PubMed vincula automáticamente a términos y subtítulos MeSH. Algunos ejemplos serían: "mal aliento" enlaza con (e incluye en la búsqueda) "halitosis", "ataque cardíaco" con "infarto de miocardio", "cáncer de mama" con "neoplasias de mama". Cuando corresponda, estos términos MeSH se "amplian" automáticamente, es decir, incluyen términos más específicos. Términos como "enfermería" se vinculan automáticamente a "Enfermería [MeSH]" o "Enfermería [Subtítulo]". Esta característica se llama Asignación automática de términos y se activa, de forma predeterminada, en la búsqueda de texto libre, pero no en la búsqueda de frases exactas (es decir, encerrando la consulta de búsqueda entre comillas dobles). [26] Esta característica hace que las búsquedas en PubMed sean más sensibles y evita resultados falsos negativos (omitidos) al compensar la diversidad de la terminología médica. [26]
PubMed no aplica el mapeo automático del término en las siguientes circunstancias: al escribir la frase citada (p. ej., "aloinjerto de riñón"), cuando está truncado en el asterisco (p. ej., aloinjerto de riñón*) y cuando se busca con etiquetas de campo (p. ej., Cáncer [ti]). [21]
La función opcional de PubMed "My NCBI" (con registro gratuito) proporciona herramientas para
y una amplia gama de otras opciones. [27] Se puede acceder al área "Mi NCBI" desde cualquier computadora con acceso web. Una versión anterior de "My NCBI" se llamaba "PubMed Cubby". [28]
LinkOut es una función de NLM para vincular y poner a disposición publicaciones de revistas locales en texto completo. [29] Unos 3.200 sitios (principalmente instituciones académicas) participan en esta instalación de NLM (a marzo de 2010 [actualizar]), desde la Universidad de Aalborg en Dinamarca hasta ZymoGenetics en Seattle. [30] Los usuarios de estas instituciones ven el logotipo de su institución dentro del resultado de búsqueda de PubMed (si la revista se lleva a cabo en esa institución) y pueden acceder al texto completo. El enlace se está consolidando con Outside Tool a partir de la actualización principal de la plataforma que se realizará en el verano de 2019. [31]
En 2016, PubMed permite a los autores de artículos comentar artículos indexados por PubMed. Esta función se probó inicialmente en modo piloto (desde 2013) y se hizo permanente en 2016. [32] En febrero de 2018, PubMed Commons se suspendió debido a que "el uso sigue siendo mínimo". [33] [34]
AskMEDLINE, una herramienta de consulta en lenguaje natural y texto libre para MEDLINE/PubMed, desarrollada por NLM, también adecuada para dispositivos portátiles. [35]
Un PMID (identificador de PubMed o identificador único de PubMed) [36] es un valor entero único , que comienza en 1, asignado a cada registro de PubMed. Un PMID no es lo mismo que un PMCID (identificador de PubMed Central), que es el identificador de todos los trabajos publicados en PubMed Central de acceso gratuito . [37]
La asignación de un PMID o PMCID a una publicación no le dice nada al lector sobre el tipo o la calidad del contenido. Los PMID se asignan a cartas al editor , opiniones editoriales, columnas de opinión y cualquier otro artículo que el editor decida incluir en la revista, así como a artículos revisados por pares. La existencia del número de identificación tampoco constituye prueba de que los papeles no hayan sido retractados por fraude, incompetencia o mala conducta. Al anuncio sobre cualquier corrección de los artículos originales se le podrá asignar un PMID.
Cada número que se ingresa en la ventana de búsqueda de PubMed se trata de manera predeterminada como si fuera un PMID. Por tanto, cualquier referencia en PubMed se puede localizar mediante el PMID.
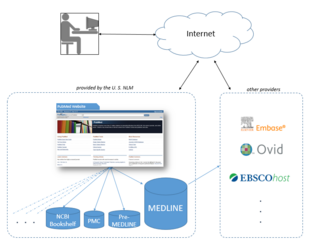
La Biblioteca Nacional de Medicina alquila la información de MEDLINE a varios proveedores privados como Embase , Ovid , Dialog , EBSCO , Knowledge Finder y muchos otros proveedores académicos, no comerciales y comerciales. [38] Hasta octubre de 2008 [actualizar], se habían concedido más de 500 licencias, más de 200 de ellas a proveedores fuera de los Estados Unidos. Como las licencias para utilizar los datos de MEDLINE están disponibles de forma gratuita, la NLM proporciona de hecho un campo de pruebas gratuito para una amplia gama [39] de interfaces alternativas y adiciones de terceros a PubMed, una de las pocas bases de datos grandes y seleccionadas profesionalmente que ofrece esta opción.
Lu identifica una muestra de 28 versiones actuales y gratuitas de PubMed basadas en la web, que no requieren instalación ni registro, que se agrupan en cuatro categorías: [39]
Como la mayoría de éstas y otras alternativas se basan esencialmente en datos de PubMed/MEDLINE alquilados bajo licencia de NLM/PubMed, se ha sugerido el término "derivados de PubMed". [39] Sin la necesidad de almacenar alrededor de 90 GB de conjuntos de datos PubMed originales, cualquiera puede escribir aplicaciones PubMed utilizando la interfaz del programa de aplicación eutils como se describe en "The E-utilities In-Depth: Parameters, Syntax and More", de Eric Sayers. , Doctor. [50] Varios generadores de formatos de citas, que toman números PMID como entrada, son ejemplos de aplicaciones web que utilizan la interfaz del programa de aplicación eutils. Las páginas web de muestra incluyen Citation Generator – Mick Schroeder, Pubmed Citation Generator – Ultrasound of the Week, PMID2cite y Cite this for me.
Los métodos alternativos para extraer datos en PubMed utilizan entornos de programación como Matlab , Python o R. En estos casos, las consultas de PubMed se escriben como líneas de código y se pasan a PubMed y luego la respuesta se procesa directamente en el entorno de programación. El código se puede automatizar para realizar consultas sistemáticas con diferentes palabras clave como enfermedad, año, órganos, etc.
Además de su función tradicional como base de datos biomédica, PubMed se ha convertido en un recurso común para la formación de modelos de lenguaje biomédico . [51] Los avances recientes en este campo incluyen el desarrollo de modelos como PubMedGPT, un modelo de 2.700 millones de parámetros entrenado con datos de PubMed por Stanford CRFM, y BiomedCLIP-PubMedBERT de Microsoft, que utiliza pares de figuras y leyendas de PubMed Central para el procesamiento de visión y lenguaje. Estos modelos demuestran el importante potencial de los datos de PubMed para mejorar las capacidades de la IA en la investigación médica y las aplicaciones sanitarias. Estos avances subrayan la creciente intersección entre la minería de datos a gran escala y el desarrollo de la IA en el campo biomédico.
Los datos a los que puede acceder PubMed se pueden reflejar localmente utilizando una herramienta no oficial como MEDOC. [52]
Millones de registros de PubMed aumentan varios conjuntos de datos abiertos sobre acceso abierto , como Unpaywall . Las bibliotecas utilizan herramientas de análisis de datos como Unpaywall Journals para ayudar con cancelaciones importantes : las bibliotecas pueden evitar suscripciones a materiales que ya se ofrecen mediante acceso abierto instantáneo a través de archivos abiertos como PubMed Central. [53]