
En biología molecular , la complementariedad describe una relación entre dos estructuras que siguen el principio de llave y cerradura. En la naturaleza, la complementariedad es el principio básico de la replicación y transcripción del ADN, ya que es una propiedad compartida entre dos secuencias de ADN o ARN , de modo que cuando están alineadas de forma antiparalela entre sí, las bases de nucleótidos en cada posición de las secuencias serán complementarias , como si nos miráramos en el espejo y viéramos el revés de las cosas. Este emparejamiento de bases complementarias permite a las células copiar información de una generación a otra e incluso encontrar y reparar daños en la información almacenada en las secuencias.
El grado de complementariedad entre dos cadenas de ácidos nucleicos puede variar, desde complementariedad completa (cada nucleótido está frente a su opuesto) hasta ninguna complementariedad (cada nucleótido no está frente a su opuesto) y determina la estabilidad de las secuencias para estar juntas. Además, varias funciones de reparación del ADN, así como funciones reguladoras, se basan en la complementariedad de pares de bases. En biotecnología, el principio de complementariedad de pares de bases permite la generación de híbridos de ADN entre ARN y ADN, y abre la puerta a herramientas modernas como las bibliotecas de ADNc . Si bien la mayor parte de la complementariedad se observa entre dos cadenas separadas de ADN o ARN, también es posible que una secuencia tenga complementariedad interna que resulte en la unión de la secuencia a sí misma en una configuración plegada.


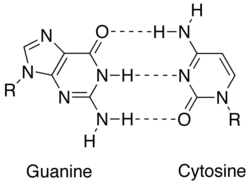
La complementariedad se logra mediante interacciones distintas entre nucleobases : adenina , timina ( uracilo en el ARN ), guanina y citosina . La adenina y la guanina son purinas , mientras que la timina, la citosina y el uracilo son pirimidinas . Las purinas son más grandes que las pirimidinas. Ambos tipos de moléculas se complementan entre sí y solo pueden aparearse con el tipo opuesto de nucleobase. En el ácido nucleico, las nucleobases se mantienen unidas por enlaces de hidrógeno , que solo funcionan de manera eficiente entre adenina y timina y entre guanina y citosina. El complemento de bases A = T comparte dos enlaces de hidrógeno, mientras que el par de bases G ≡ C tiene tres enlaces de hidrógeno. Todas las demás configuraciones entre nucleobases obstaculizarían la formación de doble hélice. Las cadenas de ADN están orientadas en direcciones opuestas, se dice que son antiparalelas . [1]
Se puede construir una cadena complementaria de ADN o ARN basándose en la complementariedad de las nucleobases. [2] Cada par de bases, A = T vs. G ≡ C, ocupa aproximadamente el mismo espacio, lo que permite la formación de una doble hélice de ADN retorcida sin distorsiones espaciales. Los enlaces de hidrógeno entre las nucleobases también estabilizan la doble hélice de ADN. [3]
La complementariedad de las cadenas de ADN en una doble hélice permite utilizar una cadena como plantilla para construir la otra. Este principio desempeña un papel importante en la replicación del ADN , sentando las bases de la herencia al explicar cómo se puede transmitir la información genética a la siguiente generación. La complementariedad también se utiliza en la transcripción del ADN , que genera una cadena de ARN a partir de una plantilla de ADN. [4] Además, el virus de la inmunodeficiencia humana , un virus de ARN monocatenario , codifica una ADN polimerasa dependiente de ARN ( transcriptasa inversa ) que utiliza la complementariedad para catalizar la replicación del genoma. La transcriptasa inversa puede cambiar entre dos genomas de ARN parentales mediante recombinación por elección de copia durante la replicación. [5]
Los mecanismos de reparación del ADN, como la corrección de pruebas , se basan en la complementariedad y permiten la corrección de errores durante la replicación del ADN mediante la eliminación de nucleobases desapareadas. [1] En general, los daños en una cadena de ADN se pueden reparar mediante la eliminación de la sección dañada y su reemplazo mediante el uso de la complementariedad para copiar información de la otra cadena, como ocurre en los procesos de reparación de desajustes , reparación por escisión de nucleótidos y reparación por escisión de bases . [6]
Las cadenas de ácidos nucleicos también pueden formar híbridos en los que el ADN monocatenario puede unirse fácilmente con el ADN o ARN complementario. Este principio es la base de técnicas de laboratorio que se utilizan habitualmente, como la reacción en cadena de la polimerasa (PCR). [1]
Dos cadenas de secuencia complementaria se denominan cadena con sentido y cadena antisentido . La cadena con sentido es, generalmente, la secuencia transcrita de ADN o ARN que se generó en la transcripción, mientras que la cadena antisentido es la cadena que es complementaria a la secuencia con sentido.

La autocomplementariedad se refiere al hecho de que una secuencia de ADN o ARN puede plegarse sobre sí misma, creando una estructura similar a una doble cadena. Dependiendo de qué tan cerca estén las partes de la secuencia que son autocomplementarias, la cadena puede formar bucles en horquilla, uniones, protuberancias o bucles internos. [1] Es más probable que el ARN forme este tipo de estructuras debido a la unión de pares de bases que no se observa en el ADN, como la unión de la guanina con el uracilo. [1]

Se puede encontrar complementariedad entre fragmentos cortos de ácido nucleico y una región codificante o un gen transcrito, y esto da como resultado un apareamiento de bases. Estas secuencias cortas de ácido nucleico se encuentran comúnmente en la naturaleza y tienen funciones reguladoras como el silenciamiento de genes. [1]
Las transcripciones antisentido son tramos de ARNm no codificante que son complementarios a la secuencia codificante. [7] Estudios de todo el genoma han demostrado que las transcripciones antisentido de ARN ocurren comúnmente en la naturaleza. En general, se cree que aumentan el potencial de codificación del código genético y agregan una capa general de complejidad a la regulación génica. Hasta ahora, se sabe que el 40% del genoma humano se transcribe en ambas direcciones, lo que subraya la importancia potencial de la transcripción inversa. [8] Se ha sugerido que las regiones complementarias entre las transcripciones sentido y antisentido permitirían la generación de híbridos de ARN bicatenario, que pueden desempeñar un papel importante en la regulación génica. Por ejemplo, el ARNm del factor 1α inducido por hipoxia y el ARNm de la β-secretasa se transcriben de manera bidireccional, y se ha demostrado que la transcripción antisentido actúa como un estabilizador de la secuencia de comandos sentido. [9]

Los miRNA , microRNA, son secuencias cortas de ARN que son complementarias a las regiones de un gen transcrito y tienen funciones reguladoras. La investigación actual indica que el miRNA circulante puede utilizarse como biomarcadores novedosos, por lo que muestra evidencia prometedora para ser utilizado en el diagnóstico de enfermedades. [10] Los miRNA se forman a partir de secuencias más largas de ARN que se cortan libres por una enzima Dicer de una secuencia de ARN que es de un gen regulador. Estas hebras cortas se unen a un complejo RISC . Se combinan con secuencias en la región aguas arriba de un gen transcrito debido a su complementariedad para actuar como un silenciador para el gen de tres maneras. Una es evitando que un ribosoma se una e inicie la traducción. Dos es degradando el ARNm al que se ha unido el complejo. Y tres es proporcionando una nueva secuencia de ARN bicatenario (dsRNA) sobre la que Dicer puede actuar para crear más miRNA para encontrar y degradar más copias del gen. Los ARN interferentes pequeños (siRNA) son similares en función a los miRNA; Provienen de otras fuentes de ARN, pero cumplen una función similar a la de los miRNA. [1] Dada su corta longitud, las reglas de complementariedad significan que aún pueden ser muy selectivos en sus objetivos de elección. Dado que hay cuatro opciones para cada base en la cadena y una longitud de 20 pb - 22 pb para un mi/siRNA, eso conduce a más de1 × 10 12 combinaciones posibles . Dado que el genoma humano tiene una longitud de aproximadamente 3.100 millones de bases, [11] esto significa que cada miRNA solo debería encontrar una coincidencia una vez en todo el genoma humano por accidente.
Las horquillas de beso se forman cuando una sola hebra de ácido nucleico se complementa consigo misma creando bucles de ARN en forma de horquilla. [12] Cuando dos horquillas entran en contacto entre sí in vivo , las bases complementarias de las dos hebras se forman y comienzan a desenrollar las horquillas hasta que se forma un complejo de ARN bicatenario (dsRNA) o el complejo se desenrolla de nuevo en dos hebras separadas debido a desajustes en las horquillas. La estructura secundaria de la horquilla antes del beso permite una estructura estable con un cambio de energía relativamente fijo. [13] El propósito de estas estructuras es equilibrar la estabilidad del bucle de la horquilla frente a la fuerza de unión con una hebra complementaria. Una unión inicial demasiado fuerte a una mala ubicación y las hebras no se desenrollarán lo suficientemente rápido; una unión inicial demasiado débil y las hebras nunca formarán completamente el complejo deseado. Estas estructuras de horquilla permiten la exposición de suficientes bases para proporcionar un control suficientemente fuerte en la unión inicial y una unión interna lo suficientemente débil para permitir el despliegue una vez que se ha encontrado una coincidencia favorable. [13]
---C G--- CG ---CG--- UACG Universidad de Georgia CGCC CGGA AAG (Asociación Estadounidense de Agentes Aduaneros) Cuaaa U CUU ---CCUGCAACUUAGGCAGG--- Una GAA ---GGACGUUGAAUCCGUCC--- GAUU UUUC UCGC GCCG CGAU GCAU GC ---G C------Sol C---Horquillas que se besan y se juntan en la parte superior de los bucles. La complementariedadde las dos cabezas anima a la horquilla a desplegarse y enderezarse paraconvertirse en una secuencia plana de dos hebras en lugar de dos horquillas.
La complementariedad permite que la información que se encuentra en el ADN o el ARN se almacene en una sola hebra. La hebra complementaria se puede determinar a partir de la plantilla y viceversa, como en las bibliotecas de ADNc. Esto también permite el análisis, como la comparación de las secuencias de dos especies diferentes. Se han desarrollado abreviaturas para escribir secuencias cuando hay desajustes (códigos de ambigüedad) o para acelerar la lectura de la secuencia opuesta en el complemento (ambigramas).
Una biblioteca de ADNc es una colección de genes de ADN expresados que se consideran una herramienta de referencia útil en los procesos de identificación y clonación de genes. Las bibliotecas de ADNc se construyen a partir de ARNm utilizando la transcriptasa inversa (RT) de la ADN polimerasa dependiente de ARN, que transcribe una plantilla de ARNm en ADN. Por lo tanto, una biblioteca de ADNc solo puede contener insertos que estén destinados a ser transcritos en ARNm. Este proceso se basa en el principio de complementariedad ADN/ARN. El producto final de las bibliotecas es ADN bicatenario, que puede insertarse en plásmidos. Por lo tanto, las bibliotecas de ADNc son una herramienta poderosa en la investigación moderna. [1] [14]
Al escribir secuencias para biología sistemática, puede ser necesario tener códigos IUPAC que signifiquen "cualquiera de los dos" o "cualquiera de los tres". El código IUPAC R (cualquier purina ) es complementario a Y (cualquier pirimidina ) y M (amino) a K (ceto). W (débil) y S (fuerte) no suelen intercambiarse [15], pero algunas herramientas lo han hecho en el pasado. [16] W y S denotan "débil" y "fuerte", respectivamente, e indican una cantidad de enlaces de hidrógeno que un nucleótido usa para emparejarse con su pareja complementaria. Una pareja usa la misma cantidad de enlaces para formar un par complementario. [17]
Un código IUPAC que excluye específicamente uno de los tres nucleótidos puede ser complementario a un código IUPAC que excluye el nucleótido complementario. Por ejemplo, V (A, C o G - "no T") puede ser complementario a B (C, G o T - "no A").
Se pueden utilizar caracteres específicos para crear una notación de ácido nucleico adecuada ( ambigráfica ) para bases complementarias (es decir, guanina = b , citosina = q , adenina = n y timina = u ), lo que hace posible complementar secuencias de ADN completas simplemente rotando el texto "al revés". [19] Por ejemplo, con el alfabeto anterior, buqn (GTCA) se leería como ubnq (TGAC, complemento inverso) si se girara al revés.
Las notaciones ambigráficas visualizan fácilmente tramos de ácidos nucleicos complementarios, como secuencias palindrómicas. [20] Esta característica se mejora cuando se utilizan fuentes o símbolos personalizados en lugar de caracteres ASCII o incluso Unicode comunes. [20]
{{cite book}}: CS1 maint: varios nombres: lista de autores ( enlace ){{cite journal}}: CS1 maint: nombres numéricos: lista de autores ( enlace )