Una coincidencia única máxima o MUM , para abreviar, es parte de un paso clave [1] en el alineamiento de secuencias múltiples de genomas en biología computacional . La identificación de MUM y otros anclajes potenciales es el primer paso en sistemas de alineación más grandes como MUMmer . Los anclajes son las áreas entre dos genomas donde son muy similares. Para entender qué es una MUM, cada palabra del acrónimo se puede desglosar individualmente. Coincidencia implica que la subcadena aparece en ambas secuencias a alinear. Único significa que la subcadena aparece sólo una vez en cada secuencia. Finalmente, maximal establece que la subcadena no es parte de otra cadena más grande que cumpla ambos requisitos anteriores. La idea detrás de esto es que las secuencias largas que coinciden exactamente y ocurren solo una vez en cada genoma son casi con certeza parte del alineamiento global.
"Dados dos genomas A y B, la subcadena Maximal Unique Match (MUM) es una subcadena común de A y B de longitud mayor que una longitud mínima especificada d (por defecto d = 20) tal que
- es máximo, es decir, no puede extenderse en ninguno de los extremos sin incurrir en un desajuste y
- es único en ambas secuencias" [2]
Identificar el conjunto de MUM en dos secuencias genómicas muy largas no es computacionalmente trivial. [1] Hay varias formas algorítmicas de abordar la identificación de MUM en alineación de secuencias múltiples. El método más simple y lento es usar la fuerza bruta donde para cada índice i en el genoma A y cada índice j en el genoma B , se calcula el prefijo común más largo (P) de A[i...n] y B[j... m] . A continuación, debe garantizar que la longitud de P sea al menos d, donde d es el tamaño MUM mínimo especificado. Finalmente debes asegurarte de que P sea único en ambos genomas. La complejidad del método de fuerza bruta es, por tanto, O(mn) . [2]
En realidad, las MUM se identifican mediante la construcción de un árbol de sufijos generalizado para A y B. Luego se crea una lista para todos los nodos internos con exactamente un hijo de cada secuencia del genoma. Para cada uno de estos nodos (identificaremos a los niños del genoma A como i y a los niños del genoma B como j ) verifique que A[i-1] ≠ B[j-1] y para aquellos donde se cumple esta condición, sabemos que esto es una MAMÁ. En este caso la complejidad se reduce a O(m+n) . [2]
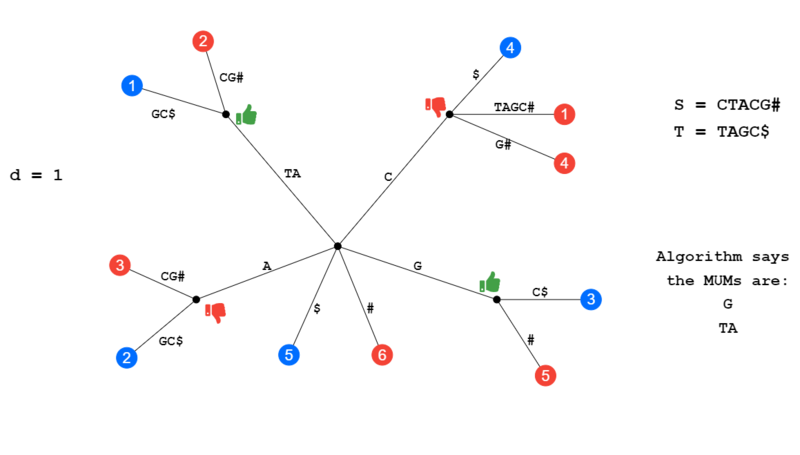
En la siguiente ilustración, dadas las cadenas iniciales S y T y una d de 1, las MUM deben ser G y TA. Una hoja roja indica que la hoja proviene de la cadena S y una hoja azul indica la cadena T. El nodo interno en A se descartó porque A[i-1] = B[j-1], lo que significa que el carácter que viene antes de ambas A es idéntico (T), esta es la condición en la que las secuencias pertenecen a una secuencia única más grande. El nodo interno en C se descarta porque tiene dos hijos de S. Esto nos deja con los MUM de G y TA.
Para ilustrar más a las MUM, tomamos el siguiente ejemplo. Digamos que d=3 y nuestros dos genomas son los siguientes:
S = CTACTGTCATGCTGAAGTCATGCGGCTGGCTTAT#
T = CTGGTGTGATGCTAAGTCATGCGGTCTGGCTTAT$
Trabajando en la secuencia, nuestra primera MUM que satisface las condiciones ocurre aquí:
S = CTAC TGT CATGCTGAAGTCATGCGGCTGGCTTAT#
T = CTGG TGT GATGCTAAGTCATGCGGTCTGGCTTAT$
TGT tiene al menos d de longitud, aparece solo una vez en ambas secuencias y los caracteres de la izquierda y la derecha difieren entre genomas. Para ilustrar un ejemplo en el que se necesita expansión para garantizar que nuestra MUM no sea parte de una secuencia más grande y única, tomemos lo siguiente:
S = CTAC TGT C ATG CTGAAGTCATGCGGCTGGCTTAT#
T = CTGG TGT G ATG CTAAGTCATGCGGTCTGGCTTAT$
Hay dos problemas en el caso de probar ATG como MUM porque ATG no es único y también es parte de una subsecuencia más grande como se ilustra aquí:
S = CTAC TGT C ATG CTGAAGTC ATGC GGCTGGCTTAT#
T = CTGG TGT G ATG CTAAGTC ATGC GGTTCTGGCTTAT$
Por lo tanto, se requiere la expansión de esta secuencia en un intento de satisfacer las condiciones para ser MUM como se muestra aquí.
S = CTAC TGT C ATGCT GAAGTC ATGCG GCTGGCTTAT#
T = CTGG TGT G ATGCT AAGTC ATGCG GTCTGGCTTAT$
El uso de este método produce de forma iterativa un producto final donde cada MUM se identifica para mayor claridad, cada MUM está codificada por colores:
S = CTAC TGT C ATGCT G AAGTCATGCGG CTGGCTTAT #
T = ACTT TGT G ATGCT AAGTCATGCGG T CTGGCTTAT $
Las MUM son un subconjunto de un conjunto más grande denominado coincidencias exactas máximas o MEMS. En un MEM se relaja la condición de unicidad de las MUM. Los MEM son como un alineamiento local , pero en este caso solo identifican secuencias donde no hay espacios.