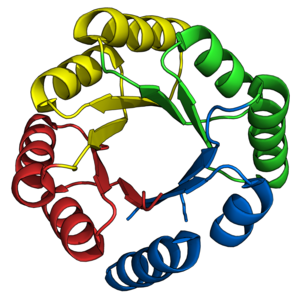
El barril TIM (triosa-fosfato isomerasa), también conocido como barril alfa/beta , [1] : 252 es un pliegue proteico conservado que consta de ocho hélices alfa (hélices α) y ocho hebras beta paralelas (hebras β) que alternan a lo largo del esqueleto peptídico . [2] La estructura lleva el nombre de la triosa-fosfato isomerasa , una enzima metabólica conservada . [3] Los barriles TIM son omnipresentes, y aproximadamente el 10% de todas las enzimas adoptan este pliegue. [4] Además, cinco de las siete clases de enzimas de la comisión de enzimas (EC) incluyen proteínas de barril TIM. [5] [6] El pliegue en barril de TIM es evolutivamente antiguo , y muchos de sus miembros poseen poca similitud en la actualidad, [7] en cambio caen dentro de la zona crepuscular de similitud de secuencia . [8] [9]
El barril beta interno (barril β) en muchos casos está estabilizado por intrincadas redes de puentes salinos . [10] Los bucles en los extremos C-terminales del barril β son responsables de la actividad catalítica [11] [12] mientras que los bucles del extremo N-terminal son importantes para la estabilidad de los barriles TIM. Se pueden insertar inserciones estructurales que van desde bucles extendidos hasta dominios proteicos independientes en lugar de estos bucles o en los terminales N/C. Los barriles TIM parecen haber evolucionado a través de eventos de duplicación genética y fusión de dominios de proteínas de medio barril, [13] y la mayoría de los barriles TIM se originan a partir de un ancestro común . Esto llevó a que muchos cilindros TIM poseyeran simetrías internas. [14] Otros eventos de duplicación de genes de este barril ancestral de TIM llevaron a enzimas divergentes que poseen la diversidad funcional que se observa hoy. Los barriles TIM también han sido un objetivo desde hace mucho tiempo para los diseñadores de proteínas . Los diseños exitosos de barriles TIM incluyen fusiones de dominios de proteínas existentes y diseños de novo . Los experimentos de fusión de dominios han dado como resultado muchos diseños exitosos, [15] [16] [17] [18] [19] [20] [21] , mientras que los diseños de novo solo arrojaron éxito después de 28 años de desarrollo incremental. [22]


El barril TIM recibe su nombre de la enzima triosa-fosfato isomerasa (TIM), que fue la primera proteína que poseyó el pliegue cristalizado . [3] Los barriles TIM contienen entre 200 y 250 residuos de aminoácidos, [2] plegados en 8 hélices alfa (hélices α) y 8 hebras beta (hebras β). Las hebras β están dispuestas en un barril beta paralelo (barril β) y están rodeadas por 8 hélices α. La propiedad definitoria de los barriles β de TIM es que siempre poseen un número de corte de 8. [2] El número de corte se determina seleccionando un residuo x en la cadena β-1 y moviéndolo a lo largo del barril β, en forma perpendicular. dirección a la dirección de las hebras, hasta alcanzar el residuo y en la cadena β-1 original. El número de residuos entre las posiciones inicial y final (|y−x|) es el número de corte. [24] Dado que el número de hebras es igual al número de corte, las cadenas laterales apuntan alternativamente hacia el poro y el núcleo, dando una simetría de 4 veces. Las hélices α rodean y encierran completamente el barril β interno. Los bucles cortos normalmente conectan las estructuras secundarias α y β, formando una topología de 8 repeticiones (βα). En algunos casos, se pueden insertar estructuras que van desde bucles extendidos hasta dominios independientes en lugar de estos bucles, o se pueden unir a los terminales N/C. Todas las enzimas del barril TIM poseen sitios catalíticos en el extremo C-terminal del barril β, [25] y los insertos estructurales presentes cerca de este extremo pueden ayudar en la actividad catalítica.
Los barriles TIM contienen dos regiones enterradas distintas , donde los residuos de aminoácidos están completamente envueltos por sus vecinos y carecen de acceso al solvente. El término "poro" es un nombre inapropiado, ya que no existen canales de disolvente dentro de esta región. La región central consta de todos los residuos que constituyen la interfaz α-β y se encuentra exterior al barril β central. La región de los poros consta de todos los residuos interiores del barril β, que están rodeados y encerrados por la columna vertebral del barril β.
Debido a la naturaleza plisada de las cadenas β, los residuos alternos a lo largo de una cadena se dividen casi uniformemente entre el poro (53%) y el núcleo (47%). En el caso de los barriles β, el 95% de los residuos del núcleo están enterrados. Sólo el 11% de sus residuos centrales son polares , poseen afinidad por el agua y poseen la capacidad de formar enlaces de hidrógeno o puentes salinos. [10] De manera similar, el 84% de los residuos de poros de la cadena β están enterrados. Sin embargo, el 42% de los residuos de sus poros son polares. Estos residuos forman intrincadas redes de puentes salinos para compensar su falta de accesibilidad a los disolventes.

Se cree que los puentes salinos dentro de los poros del barril TIM contribuyen a la estabilidad general del pliegue. Un ejemplo de una gran red de puentes salinos lo podemos encontrar en la 2-desoxirribosa-5-fosfato aldolasa . Se descubrió que esta red estaba conservada en toda la familia de aldolasas de Clase I.
La razón exacta de la sobrerrepresentación de residuos polares y puentes salinos dentro del poro aún no está clara. Un estudio propone que mejoran la capacidad de plegado en lugar de la estabilidad termodinámica de los barriles TIM. Durante el proceso de plegado , los residuos de los poros internos de las hebras β quedarían expuestos al agua. Los módulos βαβα parcialmente plegados, llamados pliegues, se estabilizarían energéticamente mediante residuos de poros polares durante esta etapa de plegado.
En otro estudio que involucró la proteína barril TIM indol-3-glicerol fosfato sintasa de S. solfataricus , se descubrió que un módulo βαβαβ conservado era una plantilla de plegado esencial, que guiaba el plegamiento de otras estructuras secundarias. El cierre del barril β sólo se produjo al final del proceso de plegado. Sin embargo, en este caso, los autores atribuyeron la estabilidad del plegado a los aminoácidos alifáticos ramificados (valina, leucina e isoleucina).
Otro elemento estabilizador en los cilindros TIM es la abrazadera de horquilla beta . Los donantes de enlaces H de cadena lateral en los extremos N de las cadenas β pares a menudo forman enlaces H con hidrógenos de amida de la cadena principal en las cadenas β impares precedentes. Estas abrazaderas (o análogos de puentes de cadena laterales hidrofóbicos) se conservan en 3 ortólogos de barril TIM de indol-3-glicerolfosfato sintasa de los reinos bacteriano y arqueal, lo que implica que surgieron en su último ancestro común y se han conservado durante más de mil millones de años.

"Las regiones N/C-terminal y de bucle de las proteínas de barril TIM son capaces de albergar inserciones estructurales que van desde motivos estructurales secundarios simples hasta dominios completos ". Estos dominios ayudan en el reconocimiento de sustratos y la actividad catalítica. A continuación se analizan cuatro ejemplos diversos de barriles TIM que contienen motivos y dominios adicionales.
Bacillus subtilis Orotidina 5'-fosfato descarboxilasa ( PDB : 1DBT ) es una proteína de barril TIM que muestra 4 hélices α en lugar de los bucles βα típicamente presentes en el terminal C del barril β (residuos 35-42, 89- 91, 126-133 y 215-219). Una de estas hélices (R215 → K219) contiene un residuo de arginina conservado (R215) necesario para interactuar con un resto fosfato en el 5'-monofosfato de orotidina. No se encontró que las otras hélices albergaran residuos críticos para la actividad catalítica y pueden desempeñar funciones estructurales.
Mycobacterium tuberculosis biosíntesis isomerasa bifuncional de histidina/triptófano (PriA) ( PDB : 2Y85 ) posee la capacidad de catalizar dos reacciones: (i) Reacción HisA: la conversión de N-[(5-fosforribosil)formimino]-5-aminoimidazol-4 -ribonucleótido carboxamida (ProFAR) a ribonucleótido N-[(5-fosforibulosil)formimino]-5-aminoimidazol-4-carboxamida (PRFAR), y (ii) reacción TrpF: antranilato de N-(5'-fosforribosil) (PRA) a 1-(O-carboxifenilamino)-1'-desoxiribulosa-5'-fosfato (CdRP). PriA es una enzima de barril TIM que se adapta a ambos sustratos utilizando bucles de sitio activo (bucles 1, 5 y 6, bucles βα extendidos en el extremo C-terminal del barril β) que cambian de conformación dependiendo del reactivo presente. El bucle 1 recorre el sitio activo solo en presencia de ProFAR. Loop5 envuelve el sitio activo, adoptando una conformación de hoja β en presencia de CdRP, o una conformación similar a un nudo en presencia de ProFAR. El bucle 6 envuelve el sitio activo para todos los reactivos.
Lactococcus lactis dihidroorotato deshidrogenasa A (DHODA) ( PDB : 2DOR ) es un ejemplo de un barril TIM que posee láminas β y bucles extendidos sobre el extremo C-terminal del barril β. DHODA cataliza la oxidación del dihidroorotato a orotato, que es parte de la vía de síntesis de novo de uridina 5'-monofosfato (UMP). Esta oxidación está mediada por el mononucleótido de flavina (FMN). Aquí, las láminas β y los bucles extendidos encierran el sitio activo formando una cavidad, al mismo tiempo que albergan varios residuos catalíticos.
El barril TIM de Mmethylophilus methylotrophus trimetilamina deshidrogenasa ( PDB : 2TMD ) es un ejemplo de inserción de dominio completa. Aquí, se inserta un dominio plegable de Rossmann en el extremo C-terminal del barril TIM. La trimetilamina deshidrogenasa cataliza la conversión de trimetilamina en formaldehído. Esta reacción requiere tanto un cofactor reducido de mononucleótido 6-S-cisteinilflavina (FMN) como un centro reducido de hierro-azufre ([4Fe-4S] + ). FMN está unido covalentemente dentro de la región C-terminal del barril β. El centro [4Fe-4S] + es demasiado grande para caber dentro del cilindro TIM y, en cambio, se coloca muy cerca, a 7 Å de distancia, en la interfaz entre el cilindro TIM y los dominios de pliegue de Rossmann.
La conservación del pliegue en barril de TIM se refleja en la conservación de su equilibrio y mecanismos de plegado cinético en parálogos bacterianos con linajes filogenéticamente distintos. La desnaturalización química de varias variantes de barril TIM naturales [27] [28] y 2 diseñadas [28] implica invariablemente un intermedio de equilibrio altamente poblado. Los intermedios cinéticos que aparecen después de la dilución de soluciones altamente desnaturalizantes implican una especie temprana mal plegada que debe desplegarse al menos parcialmente para acceder a la vía de plegamiento productivo. [27] [28] El paso limitante de la velocidad en el plegado es el cierre del barril β de 8 hebras, correspondiendo la forma de barril abierto anterior al intermedio de equilibrio. [29] Las simulaciones de dinámica molecular centradas en los nativos recapitulan los resultados experimentales y señalan el camino hacia modelos computacionales comprobables para mecanismos de plegado complejos. [30]
Las proteínas barril TIM poseen una plasticidad de secuencia inusualmente alta, formando grandes familias de enzimas ortólogas y parálogas en organismos muy divergentes. Esta plasticidad sugiere un paisaje de secuencias que permite la adaptación de las proteínas a una variedad de condiciones ambientales, en gran medida independiente de la historia filogenética, manteniendo al mismo tiempo su función. Se utilizó un enfoque de escaneo mutacional profundo [31] y un ensayo de competencia [32] para determinar la aptitud de todos los posibles mutantes de aminoácidos en todas las posiciones en 3 enzimas barril TIM hipertermófilas indol-3-glicerolfosfato sintasa (IGPS) para apoyar el crecimiento de un huésped de levadura que carece de IGPS. Aunque las enzimas IGPS de dos bacterias y una de arqueas eran sólo entre un 30% y un 40% idénticas en secuencia, sus paisajes de aptitud estaban fuertemente correlacionados: los mismos aminoácidos en las mismas posiciones en las tres proteínas diferentes tenían una aptitud muy similar. La correlación puede considerarse como la conservación del panorama de aptitud para una enzima barril TIM a lo largo del tiempo evolutivo.
De los aproximadamente 200 residuos necesarios para formar completamente un barril TIM, alrededor de 160 se consideran estructuralmente equivalentes entre diferentes proteínas que comparten este pliegue. Los residuos restantes se encuentran en las regiones de bucle que unen las hélices y las hebras; Los bucles en el extremo C-terminal de las hebras tienden a contener el sitio activo , que es una de las razones por las que este pliegue es tan común: los residuos necesarios para mantener la estructura y los residuos que efectúan la catálisis enzimática son en su mayor parte subconjuntos distintos: [33] De hecho, los bucles de enlace pueden ser tan largos que contengan otros dominios proteicos. Recientemente, se ha demostrado que se pueden intercambiar bucles catalíticos entre diferentes enzimas TIM barril como unidades semiautónomas de grupos funcionales. [34]


La teoría predominante sobre la evolución del barril TIM implica la duplicación y fusión de genes, comenzando con medio barril que eventualmente formó un barril TIM completo. Múltiples estudios respaldan la teoría de la evolución divergente a partir de un solo ancestro y se analizan a continuación.
A principios de la década de 1990, se observó que todas las estructuras de barriles de TIM resueltas en ese momento eran enzimas, lo que indicaba una divergencia con respecto a un ancestro común. [11] [12] Además, todos los barriles TIM poseían sitios activos en el extremo C-terminal de los barriles β. sugirió que un sitio de unión de fosfato común, formado por una pequeña hélice α y bucles de barril TIM-7/8, indicaba fuertemente una evolución divergente. [35] Estudios adicionales de estos grupos de fosfato, concluyeron que 12 de 23 familias de barriles SCOP TIM divergieron de un ancestro común. [36] De manera similar, hubo indicios de ascendencia común para 17 de las 21 familias de barriles de CATH TIM. [7] Con base en estos informes, se considera plausible que la mayoría de las proteínas barril TIM evolucionaron a partir de un ancestro común.

Muchas proteínas de barril TIM poseen simetría interna de 2, 4 u 8 veces, lo que sugiere que los barriles TIM evolucionaron a partir de motivos ancestrales (βα) 4 , (βα) 2 o βα a través de la duplicación de genes y la fusión de dominios . Un buen ejemplo de simetría interna doble se observa en las enzimas ProFAR isomerasa (HisA) e imidazol glicerol fosfato sintasa (HisF) de la vía de biosíntesis de histidina de Thermotoga maritima . [13] Catalizan 2 reacciones sucesivas en la vía, poseen una homología de secuencia del 25% y poseen desviaciones cuadráticas medias (RMSD) entre 1,5 y 2 Å, lo que sugiere una divergencia de un ancestro común. Más interesante aún, los bucles en los extremos C terminales tanto de HisA como de HisF mostraron un patrón repetido doble, lo que sugiere que su ancestro común también poseía simetría interna doble. Utilizando estas observaciones, se construyó un modelo para la evolución de los barriles TIM. [13] Un medio barril ancestral habría sufrido un evento de duplicación y fusión de genes, lo que resultó en una única proteína que contenía dos dominios de medio barril. Se habrían producido adaptaciones estructurales, lo que habría dado como resultado la fusión de estos dominios para formar un barril β cerrado y la formación de un barril TIM ancestral. También se habrían producido adaptaciones funcionales, lo que habría resultado en la evolución de una nueva actividad catalítica en el extremo C terminal del barril β. En este punto, el ancestro común de HisA e HisF habría sufrido un segundo evento de duplicación genética. La evolución divergente de los genes duplicados del ancestral barril TIM habría dado lugar a la formación de HisA e HisF.
Curiosamente, este modelo evolutivo ha sido validado experimentalmente mediante diseño racional de proteínas y evolución dirigida . Hocker et al. Primero fusionó dos mitades C-terminales de HisF, produciendo HisF-CC. Luego, esta construcción se estabilizó mediante la inserción de un puente salino interno , produciendo HisF-C*C. [17] Se logró una mayor estabilización y solubilización gradual de HisF-C*C optimizando la interfaz de medio barril, generando HisF-C**C y HisF-C***C, respectivamente. [15] [16] La estructura cristalina de HisF-C***C reveló un barril TIM simétrico doble, lo que valida la posibilidad de fusión de dominio natural. Además, Höcker creó los primeros cañones quiméricos HisAF e HisFA TIM utilizando medios cañones HisA e HisF. [17] Estos experimentos llevaron a la propuesta de un nuevo medio de diversificación y evolución de las enzimas de barril TIM mediante el intercambio de dominios de medio barril (βα)4 entre barriles TIM preexistentes. De acuerdo con esta idea, se estableció una alta actividad catalítica en la construcción HisAF. [18] De manera similar, también se han creado barriles TIM quiméricos de βα 5 -flavodoxina (CheY)/HisF, [19] [20] y un barril TIM basado en HisF perfectamente simétrico 2 veces [ 21] [28] .
La existencia de una simetría interna de 4/8 veces se sugirió basándose en un análisis computacional de secuencias de barriles TIM. [14] Por ejemplo, se sugirió que Escherichia coli KDPG aldolasa [37] ( PDB : 1FQ0 ) poseía una simetría distinta de 4 veces, con una simetría discernible de 8 veces. El diseño de un barril TIM con simetría cuádruple [22] confirmó la posibilidad de órdenes superiores de simetría interna en barriles TIM naturales, y se discutirá en detalle en la siguiente sección. Hasta la fecha no se ha informado de ninguna evidencia experimental de la existencia de barriles TIM simétricos 8 veces.
"El pliegue en barril TIM ha sido un objetivo desde hace mucho tiempo para los diseñadores de proteínas de novo ". Como se describió anteriormente, se han diseñado con éxito numerosos cañones TIM basándose en medios cañones naturales preexistentes. Por el contrario, el diseño de novo de los barriles TIM se produjo en pasos incrementales durante un período de 28 años. [38]
La serie Octarellin [39] [40] [41] [42] [43] de proteínas (Octarellin I → VI) fueron los primeros intentos de crear un barril TIM de novo . Como el campo del diseño de proteínas estaba todavía en su infancia, estos intentos de diseño sólo tuvieron un éxito limitado. Aunque mostraron espectros de dicroísmo circular consistentes con las proteínas αβ y algunas características de plegamiento cooperativo, todos los péptidos de la serie Octarellin eran insolubles y tuvieron que resolubilizarse de los cuerpos de inclusión para una mayor caracterización. Curiosamente, Octarellin V.1 [44] mostró un pliegue similar a Rossmann en condiciones de cocristal.
La serie de proteínas Symmetrin (Symmetrin-1→4) mostró características biofísicas más favorables. Symmetrin-1 era fácilmente soluble, mostraba espectros de dicroísmo circular consistentes con las proteínas αβ y mostraba excelentes características cooperativas de despliegue y replegamiento. A pesar de estos avances, todas las proteínas de esta familia mostraban características fundidas cuando se analizaban mediante RMN ( resonancia magnética nuclear ) y no se podían seguir trabajando para resolver sus estructuras.
Las proteínas de la serie sTIM [22] representaron el primer diseño exitoso de barril TIM de novo . [45] [38] sTIM-11 ( PDB : 5BVL ) fue diseñado con una simetría interna cuádruple, para reducir la complejidad del diseño computacional utilizando el paquete de software Rosetta. [46] Los primeros principios [47] derivados previamente se utilizaron para delinear topologías y longitudes de estructuras secundarias. sTIM-11 demostró ser un diseño altamente termoestable y plegable de forma cooperativa que adoptó su estructura prevista.
![]() Este artículo fue adaptado de la siguiente fuente bajo una licencia CC BY 4.0 (2020) (informes de los revisores): Deepesh Nagarajan; Neha Nanajkar (2020). "El pliegue del barril de TIM" (PDF) . WikiRevista científica . 3 (1): 4. doi :10.15347/WJS/2020.004. ISSN 2470-6345. Wikidata Q87400003.
Este artículo fue adaptado de la siguiente fuente bajo una licencia CC BY 4.0 (2020) (informes de los revisores): Deepesh Nagarajan; Neha Nanajkar (2020). "El pliegue del barril de TIM" (PDF) . WikiRevista científica . 3 (1): 4. doi :10.15347/WJS/2020.004. ISSN 2470-6345. Wikidata Q87400003.