El estudio de asociación de todo el transcriptoma (TWAS) es una metodología genética que se puede utilizar para comparar los componentes genéticos de la expresión génica y los componentes genéticos de un rasgo para determinar si existe una asociación entre los dos componentes. [1] [2] Los TWAS son útiles para la identificación y priorización de genes causales candidatos en el análisis de genes candidatos después de estudios de asociación de todo el genoma. [3] Los TWAS analizan los productos de ARN de un tejido específico y brindan a los investigadores la capacidad de observar los genes que se expresan, así como los niveles de expresión génica, que varían según el tipo de tejido. Los TWAS son herramientas bioinformáticas valiosas y flexibles que analizan las asociaciones entre las expresiones de genes y rasgos y enfermedades complejos. [4] Al observar la asociación entre la expresión génica y el rasgo expresado, se pueden investigar los mecanismos reguladores genéticos para determinar el papel que desempeñan en el desarrollo de rasgos y enfermedades específicos.
Un transcriptoma es la suma de todas las transcripciones de ARN que están presentes en una célula, tejido u órgano determinado dentro de un organismo. Los transcriptomas incluyen tanto el ARNm, que funciona como intermediario del dogma central, como los ARN no codificantes que pueden desempeñar otras funciones en la síntesis de proteínas. [5] En el dogma central, se describe cómo el ADN puede producir proteínas a través de la transcripción y la traducción. Los ARN están presentes en una célula en concentraciones variadas y desempeñan varias funciones fuera del dogma central y se pueden identificar en función de su longitud y función. Es a través de elementos funcionales que se pueden regular las actividades transcripcionales y traduccionales de los genes. [4] El análisis del transcriptoma es beneficioso para obtener información sobre todos los ARN presentes y puede proporcionar información valiosa sobre los mecanismos genéticos que son específicos de los tejidos. [6] El transcriptoma se investigó por primera vez en la década de 1990 en un experimento realizado para identificar un transcriptoma parcial del cerebro humano. Los investigadores pudieron identificar 609 secuencias de ARNm. [5] Desde entonces, se han logrado muchos avances en los métodos de secuenciación de próxima generación. [6] Ahora es posible desarrollar transcriptomas de forma rutinaria gracias a los avances en estos métodos y a las nuevas tecnologías, como los microarrays y el ARN-Seq. Ambos métodos requieren imágenes computarizadas, así como un alto nivel de lecturas y análisis estadístico. [5] Al obtener información sobre la expresión génica a través de los ARNm, se han descubierto muchas aplicaciones. El análisis de transcriptomas ha demostrado ser beneficioso para identificar procesos patológicos, así como elementos reguladores en las progresiones de enfermedades, ha ayudado al desarrollo de fármacos mediante la identificación de procesos patológicos, ofrece información sobre estrategias terapéuticas y ha mejorado la identificación de genes que pueden responder a factores ambientales tanto bióticos como abióticos, así como el papel que desempeñan las condiciones ambientales en la expresión génica. [5]
Un estudio de asociación de todo el genoma , o GWAS, es una herramienta genética que utiliza polimorfismos de un solo nucleótido, o SNP, para identificar si un rasgo o enfermedad está vinculado a una variante genética específica. Al observar si las frecuencias de una variante específica se asocian más comúnmente, o son más altas de lo esperado, con el rasgo dado, se desarrolla una asociación entre el rasgo y la variante. Sin embargo, muchas de estas asociaciones pueden desarrollarse en todo el individuo debido al desequilibrio de ligamiento y al gran tamaño del genoma. Aunque los GWAS brindan información valiosa para identificar marcadores en todo el genoma, una gran parte de los SNP están presentes en regiones de ARNm no codificantes y muchos tienen funciones desconocidas que son difíciles de determinar a través de métodos estándar, ya que estas regiones del genoma no fabrican ningún producto. [4]
Los estudios de asociación de transcripción amplia pueden tomar la información de los resultados de GWAS y pueden utilizar estos resultados como datos de referencia, y luego pueden ayudar a identificar y priorizar genes. [4] Para realizar este análisis, se debe obtener un panel de referencia para la expresión génica, como un loci de rasgos cuantitativos de expresión (eQTL), que ayuda a mostrar la regulación de la expresión génica. [4] Usando el panel de referencia, se puede generar un modelo predictivo para imputar la variación de expresión de genes. La imputación es el proceso por el cual se pueden predecir los niveles de expresión de genes en otros organismos a través de la variación que existe en su genoma con base en un panel de referencia. Al predecir los niveles de expresión génica dentro de un tejido, se eliminan otras variables como el medio ambiente y los efectos epigenéticos ya que esta predicción se basa únicamente en la variante presente y el nivel esperado de expresión génica. Sin embargo, esto puede conducir a imprecisiones con las predicciones de expresión génica ya que tanto el medio ambiente como las modificaciones epigenéticas pueden alterar el nivel de expresión génica. [7] Los componentes cis-genéticos son el foco principal de los eQTL, ya que son elementos que se encuentran dentro de 1 megabase de un gen. [2] El modelo predictivo se puede aplicar luego a las muestras de GWAS para predecir la expresión génica de los SNP significativos de ese estudio. Después de que se imputan los niveles de expresión, se realizan pruebas de asociación gen-rasgo asociando los niveles de expresión y genotipos previstos con los fenotipos del individuo. [8] Básicamente, un TWAS puede tomar los resultados de GWAS y predecir los efectos de cada variante en los niveles de expresión de los genes que están asociados con el rasgo, y puede hacer esto para cada loci que esté asociado con el rasgo en todo el genoma. [7]
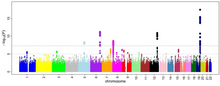
Los resultados se pueden interpretar en un gráfico de Manhattan, de manera similar a los resultados de GWAS. Cualquier loci que se considere que tiene resultados estadísticamente significativos tendrá un valor P más alto, y esto indica que es probable que el loci esté asociado con el rasgo o la enfermedad que se está investigando. Cualquier valor P estadísticamente significativo tiene un valor P logarítmico más alto y se muestra por encima de la línea de corrección de Bonferroni . El gráfico de Manhattan se llama así porque los genes estadísticamente significativos parecen aparecer como "rascacielos" en el gráfico, y cuando hay muchos genes que están asociados con el rasgo, el gráfico se asemeja al horizonte de Manhattan. Aunque la imagen del gráfico de Manhattan es para un estudio de GWAS, los resultados de TWAS se muestran de la misma manera. Los loci estadísticamente significativos son genes que tienen SNP significativamente asociados cuya expresión se correlaciona con el rasgo o la enfermedad de interés.
PrediXcan [1] y FUSION [2] son programas de análisis de doble cadena de caracteres (TWAS) que se han utilizado en estudios genéticos para investigar las asociaciones entre genes y rasgos. PrediXcan es un programa de análisis de doble cadena de caracteres bien desarrollado que tiene la capacidad de estimar la expresión regulada genéticamente y determinar las asociaciones con el fenotipo que se está investigando. Utiliza un modelo de regresión penalizado para dar peso a los niveles de expresión genética observada y a los SNP cis derivados del conjunto de datos de referencia. [8] Luego, el programa utiliza datos de genotipos individuales para realizar pruebas de asociación entre genes y rasgos. FUSION es otro programa de análisis de doble cadena de caracteres que utiliza un análisis estadístico diferente para crear las pruebas de asociación. En este modelo, los métodos de imputación del modelo predictivo se calculan en función de un modelo mixto lineal disperso bayesiano. [8] La ventaja de este programa es que puede realizar pruebas de asociación en datos de genotipos individuales, pero este programa también puede tomar información de conjuntos de datos a gran escala mediante imputación.
Las ventajas de esta metodología son la comprensión que proporciona a los investigadores sobre la función de los genes y la asociación entre las funciones de los genes y la expresión génica. El TWAS tiene el potencial de tomar los resultados del GWAS y extenderlos para ayudar a comprender los mecanismos de las enfermedades. [4] Además, como este método utiliza loci que se identificaron previamente mediante el análisis del GWAS, existe una menor carga de pruebas asociada con un TWAS ya que se analizan menos sitios. Al reducir el número de loci que se analizan, esto permite un análisis más profundo de los sitios analizados y puede brindar una mayor comprensión de las funciones y asociaciones de los loci significativos.
Los TWAS también tienen la ventaja de reducir los efectos de los factores de confusión. Al construir un modelo predictivo, solo se tiene en cuenta la expresión genética, no la expresión total. La expresión total incluye factores como el entorno y las modificaciones epigenéticas de los niveles de expresión, y no se tienen en cuenta en el modelo predictivo. Al no tener en cuenta estos factores, se puede reducir la precisión de los niveles previstos de expresión genética; sin embargo, también se reducen los efectos de las variables de confusión en los resultados. [2]
Otra ventaja de la TWAS es que los resultados son específicos de cada tejido. El nivel de expresión génica varía según el tejido en el que se encuentren los genes, ya que cada tejido tiene patrones de empalme y de regulación específicos. Al tener resultados específicos de cada tejido, se amplía la información que se puede obtener a través de estos estudios, ya que los resultados tienen la capacidad de mostrar cómo difiere la regulación génica según los tipos de tejido, así como también cómo se regulan las funciones y si existen mecanismos reguladores comunes entre los tejidos o si los mecanismos reguladores tienen diferentes funciones en diferentes tejidos. Los métodos de TWAS de tejido cruzado también tienen la posibilidad de identificar posibles genes causales de enfermedades y rasgos a mayor escala; sin embargo, los métodos de tejido único tienen la capacidad de determinar asociaciones en función de un caso específico. [8]
Muchas de las desventajas de TWAS son implicaciones de las capacidades de predicción del modelo utilizado para predecir los niveles de expresión genética en función de los genotipos. Una desventaja de TWAS es que se centra principalmente en los componentes cisgenéticos para la imputación y, en la mayoría de los estudios, no identifica ninguna variante de componente transgenético. Esto actúa como una desventaja para TWAS, ya que las variantes de componentes transgenéticos son cualquier mecanismo regulador que se encuentre fuera de un rango de 1 megabase del gen, y aunque se encuentren a una distancia significativa del gen de interés, muchos mecanismos reguladores tienen el potencial de actuar a largo plazo y aún pueden afectar la expresión. Al no tener en cuenta estos componentes, se reduce la precisión de los niveles de expresión genética previstos y puede causar una desviación entre los niveles de expresión esperados y observados. Como se mencionó anteriormente, otra desventaja de estos estudios es que los mecanismos ambientales y epigenéticos para la regulación de la expresión genética no se tienen en cuenta con el modelo predictivo de la expresión genética, lo que también tiene el potencial de generar imprecisiones con los niveles de expresión genética previstos y los niveles de expresión observados. Otro desafío para TWAS es que puede ser difícil predecir niveles precisos de expresión genética cuando los genes tienen niveles bajos de heredabilidad. Los eQTL dependen de un nivel de heredabilidad y, cuando se observa una heredabilidad baja, puede afectar la observación de falsos positivos y puede afectar negativamente las capacidades de predicción del modelo utilizado para TWAS. [4]
Además, otro desafío para los TWAS, muy similar a los resultados de los GWAS, es que estos estudios solo pueden demostrar asociaciones a partir de los resultados. Aunque se puede ver una asociación estadísticamente significativa entre el gen o loci de interés y el rasgo o la enfermedad, no se puede derivar ninguna relación causal. Para establecer una relación causal, se necesitarían realizar estudios adicionales que utilicen un enfoque de genética inversa para la eliminación de genes o mutagénesis dirigida al sitio para identificar relaciones causales.
Otro problema con los resultados de TWAS son las implicaciones del sesgo tisular y la corregulación. Debido a la especificidad de los mecanismos de regulación genética dentro de cada tejido, se necesitarían realizar muchos experimentos para determinar la naturaleza específica del tejido de cada asociación de loci y cómo estas asociaciones difieren entre los tipos de tejido. [3] La corregulación es el resultado de un mecanismo regulador que controla la expresión de más de un loci a la vez. Al controlar más de un loci, se pueden establecer asociaciones entre los loci de interés junto con otros genes o loci que están controlados únicamente por el mismo mecanismo y pueden no tener ninguna asociación con el rasgo o la enfermedad de interés, lo que conduce a resultados falsos positivos. [3]
Se realizó un estudio TWAS después de un GWAS que investigó los loci asociados con la esquizofrenia. A partir de los resultados del GWAS, se localizaron más de 100 loci de riesgo. Luego se utilizó un TWAS para identificar 157 loci significativos utilizando datos de expresión, y 35 de los loci identificados a partir del TWAS no se alinearon con los loci del GWAS. Luego se acotaron aún más los resultados utilizando investigaciones de objetivos reguladores. Se encontró que 42 de estos genes tenían una asociación estadísticamente significativa con los fenotipos de la cromatina, que es un mecanismo regulador que podría investigarse más a fondo. MAPK3 fue una asociación que se observó que tenía un gran impacto en los fenotipos del desarrollo neurológico, y se priorizó aún más como gen causal candidato. [9]
En 2018, se utilizó un TWAS para identificar genes causales candidatos para el cáncer de mama. Los datos se recopilaron del Atlas del Genoma del Cáncer para establecer modelos genéticos, así como de 229.000 mujeres de ascendencia europea. En este estudio, se evaluaron 8.597 genes. A través de estudios GWAS, alrededor de 170 loci se asociaron con al menos una variante del cáncer de mama. En este estudio, se encontró que 179 genes tenían una asociación con una variante del cáncer de mama. De los 179 genes con asociaciones, 48 se identificaron como estadísticamente significativos utilizando un umbral de corrección de Bonferroni (como se ve en el gráfico de Manhattan anterior). [10] Nunca antes se había informado que 14 de estos estuvieran asociados con un riesgo de cáncer de mama. Los otros 34 genes en loci de riesgo conocidos tenían 23 que no tenían ningún SNP de riesgo asociado. [10] Utilizando la eliminación de genes, se encontró que 13 genes con altos niveles previstos de expresión estaban asociados con un mayor riesgo. Cuando se inhibieron, los estudios mostraron que 11 de los genes investigados tuvieron un efecto en una línea celular de cáncer de mama, especialmente en células mamarias normales 184A1. [10] Estos genes incluyen los siguientes: PIDD1, NRBF2 y ABHD8 . [10] Todos los genes identificados en el estudio, tanto los regulados positivamente como negativamente, tenían una heredabilidad cis relativamente alta .
En 2021 se completó un estudio TWAS que utilizó el GWAS de la enfermedad de Parkinson (EP) más reciente que utilizó a 480.000 personas. A partir de esos resultados, se encontró que 18 genes tenían una asociación estadísticamente significativa con la EP. El más significativo de ellos fue LRRC37A2, que se encontró asociado en los 13 tejidos cerebrales. [11]
El Atlas TWAS es un sitio que ha sido diseñado para integrar los hallazgos de muchos estudios de TWAS. Este atlas existe virtualmente y es accesible al público. Los resultados y hallazgos que se publican en el Atlas TWAS se pueden integrar y combinar para ayudar a futuros estudios y a la comprensión de los mecanismos de regulación genética. Los resultados se presentan de manera visual para mejorar la integración de los resultados. Actualmente, se han publicado 401 226 asociaciones TWAS de 200 publicaciones, que abarcan 257 rasgos y 22 247 genes al 25 de abril de 2022. [12]