En filogenética y filogenética computacional , la parsimonia máxima es un criterio de optimización bajo el cual el árbol filogenético minimiza el número total de cambios de estado de carácter (o minimiza el costo de los cambios de estado de carácter ponderados diferencialmente). Bajo el criterio de máxima parsimonia, el árbol óptimo minimizará la cantidad de homoplasia (es decir, evolución convergente , evolución paralela y reversiones evolutivas ). En otras palabras, bajo este criterio, se considera mejor el árbol más corto posible que explique los datos. Algunas de las ideas básicas detrás de la máxima parsimonia fueron presentadas por James S. Farris [1] en 1970 y Walter M. Fitch en 1971. [2]
La máxima parsimonia es un criterio intuitivo y sencillo, y por este motivo es popular. Sin embargo, aunque es fácil calificar un árbol filogenético (contando el número de cambios de estado de carácter), no existe un algoritmo para generar rápidamente el árbol más parsimonioso. En cambio, se debe buscar el árbol más parsimonioso en el "espacio de árbol" (es decir, entre todos los árboles posibles). Para un número pequeño de taxones (es decir, menos de nueve) es posible hacer una búsqueda exhaustiva , en la que se puntúan todos los árboles posibles y se selecciona el mejor. Para nueve a veinte taxones, generalmente será preferible utilizar ramificar y unir , que también garantiza la obtención del mejor árbol. Para un mayor número de taxones se debe realizar una búsqueda heurística .
Debido a que el árbol más parsimonioso es siempre el árbol más corto posible, esto significa que, en comparación con un árbol hipotético "verdadero" que en realidad describe la historia evolutiva desconocida de los organismos bajo estudio, el "mejor" árbol según la parsimonia máxima Este criterio a menudo subestimará el cambio evolutivo real que podría haber ocurrido. Además, la parsimonia máxima no es estadísticamente consistente. Es decir, no se garantiza que se produzca el árbol verdadero con alta probabilidad, dados datos suficientes. Como lo demostró Joe Felsenstein en 1978 , [3] la parsimonia máxima puede ser inconsistente bajo ciertas condiciones, como la atracción de ramas largas . Por supuesto, cualquier algoritmo filogenético también podría ser estadísticamente inconsistente si el modelo que emplea para estimar el árbol preferido no coincide con precisión con la forma en que ocurrió la evolución en ese clado. Esto es incognoscible. Por lo tanto, si bien la consistencia estadística es una propiedad teórica interesante, queda fuera del ámbito de la comprobabilidad y es irrelevante para los estudios filogenéticos empíricos. [4]
En filogenética, la parsimonia se interpreta principalmente como favorecer a los árboles que minimizan la cantidad de cambio evolutivo requerido (ver, por ejemplo, [2] ). Alternativamente, la parsimonia filogenética se puede caracterizar por favorecer los árboles que maximizan el poder explicativo al minimizar el número de similitudes observadas que no pueden explicarse por la herencia y la descendencia común. [5] [6] La minimización del cambio evolutivo requerido, por un lado, y la maximización de las similitudes observadas que pueden explicarse como homología, por el otro, pueden dar como resultado diferentes árboles preferidos cuando algunas características observadas no son aplicables en algunos grupos que están incluidos en el grupo. árbol, y este último puede verse como el enfoque más general. [7] [8] [9]
Si bien la evolución no es un proceso inherentemente parsimonioso, siglos de experiencia científica respaldan el principio de parsimonia antes mencionado ( la navaja de Occam ). Es decir, la suposición de una cadena de acontecimientos más simple y parsimoniosa es preferible a la suposición de una cadena de acontecimientos más complicada y menos parsimoniosa. Por lo tanto, normalmente se busca la parsimonia ( sensu lato ) al inferir árboles filogenéticos y, en general, en la explicación científica. [10]
La parsimonia es parte de una clase de métodos de estimación de árboles basados en caracteres que utilizan una matriz de caracteres filogenéticos discretos y estados de caracteres para inferir uno o más árboles filogenéticos óptimos para un conjunto de taxones , comúnmente un conjunto de especies o poblaciones reproductivamente aisladas de un solo especies. Estos métodos operan evaluando árboles filogenéticos candidatos de acuerdo con un criterio explícito de optimización ; el árbol con la puntuación más favorable se toma como la mejor hipótesis de las relaciones filogenéticas de los taxones incluidos. Se utiliza la máxima parsimonia con la mayoría de los tipos de datos filogenéticos; Hasta hace poco, era el único método de estimación de árboles basado en caracteres ampliamente utilizado para datos morfológicos.
Inferir filogenias no es un problema trivial. Existe una gran cantidad de posibles árboles filogenéticos para cualquier conjunto de taxones de tamaño razonable; por ejemplo, tan sólo diez especies dan más de dos millones de posibles árboles sin raíces. Se deben buscar estas posibilidades para encontrar el árbol que mejor se ajuste a los datos según el criterio de optimización. Sin embargo, los datos en sí no conducen a una solución aritmética simple del problema. Idealmente, esperaríamos que la distribución de cualquier carácter evolutivo (como rasgos fenotípicos o alelos ) siguiera directamente el patrón de ramificación de la evolución. Así, podríamos decir que si dos organismos poseen un carácter compartido, deberían estar más estrechamente relacionados entre sí que con un tercer organismo que carezca de este carácter (siempre que ese carácter no estuviera presente en el último ancestro común de los tres, en cuyo caso sería una simplesiomorfía ). Predeciríamos que los murciélagos y los monos están más estrechamente relacionados entre sí que cualquiera de ellos con un elefante, porque los murciélagos y los monos macho poseen testículos externos , de los que carecen los elefantes. Sin embargo, no podemos decir que los murciélagos y los monos estén más estrechamente relacionados entre sí que con las ballenas, aunque ambos tienen testículos externos ausentes en las ballenas, porque creemos que los machos de la última especie ancestral común de los tres tenían testículos externos. .
Sin embargo, los fenómenos de evolución convergente , evolución paralela y reversiones evolutivas (denominados colectivamente homoplasia ) añaden un inconveniente desagradable al problema de inferir la filogenia. Por varias razones, dos organismos pueden poseer un rasgo que se infiere que no estuvo presente en su último ancestro común: si ingenuamente tomáramos la presencia de este rasgo como evidencia de una relación, inferiríamos un árbol incorrecto. Los datos filogenéticos empíricos pueden incluir una homoplasia sustancial, y diferentes partes de los datos sugieren a veces relaciones muy diferentes. Los métodos utilizados para estimar árboles filogenéticos tienen como objetivo explícito resolver el conflicto dentro de los datos eligiendo el árbol filogenético que mejor se ajuste a todos los datos en general, aceptando que algunos datos simplemente no encajarán. A menudo se cree erróneamente que la parsimonia supone que la convergencia es poco común; de hecho, incluso los caracteres derivados de manera convergente tienen algún valor en los análisis filogenéticos basados en la parsimonia máxima, y la prevalencia de la convergencia no afecta sistemáticamente el resultado de los métodos basados en la parsimonia. [11]
Los datos que no se ajustan perfectamente a un árbol no son simplemente "ruido", pueden contener señales filogenéticas relevantes en algunas partes de un árbol, incluso si entran en conflicto con el árbol en general. En el ejemplo de la ballena dado anteriormente, la falta de testículos externos en las ballenas es homoplástica: refleja un retorno a la condición que se infiere que estuvo presente en los antiguos ancestros de los mamíferos, cuyos testículos eran internos. Esta similitud inferida entre las ballenas y los antiguos ancestros de los mamíferos está en conflicto con el árbol que aceptamos basado en el peso de otros caracteres, ya que implica que los mamíferos con testículos externos deberían formar un grupo que excluya a las ballenas. Sin embargo, entre las ballenas, la inversión de los testículos internos en realidad asocia correctamente los distintos tipos de ballenas (incluidos los delfines y las marsopas) en el grupo Cetacea . Aún así, la determinación del árbol que mejor se ajusta (y, por tanto, qué datos no se ajustan al árbol) es un proceso complejo. La máxima parsimonia es un método desarrollado para hacer esto.
Los datos de entrada utilizados en un análisis de máxima parsimonia tienen la forma de "caracteres" para una variedad de taxones. No existe una definición generalmente aceptada de carácter filogenético, pero operativamente un carácter puede considerarse como un atributo, un eje a lo largo del cual se observa que varían los taxones. Estos atributos pueden ser físicos (morfológicos), moleculares, genéticos, fisiológicos o conductuales. El único acuerdo generalizado sobre los caracteres parece ser que la variación utilizada para el análisis de caracteres debe reflejar la variación hereditaria . No está del todo resuelto si debe ser heredable directamente o si la herencia indirecta (por ejemplo, conductas aprendidas) es aceptable.
Cada carácter se divide en estados de carácter discretos, en los que se clasifican las variaciones observadas. Los estados de carácter a menudo se formulan como descriptores, que describen la condición del sustrato del carácter. Por ejemplo, el carácter "color de ojos" puede tener los estados "azul" y "marrón". Los personajes pueden tener dos o más estados (sólo pueden tener uno, pero estos personajes no aportan nada a un análisis de máxima parsimonia y, a menudo, se excluyen).
Codificar caracteres para el análisis filogenético no es una ciencia exacta y existen numerosas cuestiones complicadas. Por lo general, los taxones se califican con el mismo estado si son más similares entre sí en ese atributo en particular que cada uno de los taxones calificados con un estado diferente. Esto no es sencillo cuando los estados de los personajes no están claramente delineados o cuando no logran capturar todas las variaciones posibles en un personaje. ¿Cómo se calificaría el carácter mencionado anteriormente para un taxón (o individuo) con ojos color avellana? ¿O verde? Como se señaló anteriormente, la codificación de caracteres generalmente se basa en la similitud: los ojos color avellana y verde pueden agruparse con los azules porque son más similares a ese color (ser claros), y el carácter podría luego recodificarse como "color de ojos: claro; oscuro". " Alternativamente, puede haber caracteres de varios estados, como "color de ojos: marrón; avellana, azul; verde".
Las ambigüedades en la delimitación y puntuación del estado de carácter pueden ser una fuente importante de confusión, disputa y error en el análisis filogenético que utiliza datos de carácter. Tenga en cuenta que, en el ejemplo anterior, "ojos: presentes; ausentes" también es un carácter posible, lo que crea problemas porque el "color de ojos" no es aplicable si los ojos no están presentes. Para tales situaciones, un "?" ("desconocido") se califica, aunque a veces se usa "X" o "-" (este último generalmente en datos de secuencia ) para distinguir los casos en los que un carácter no se puede calificar de un caso en el que el estado es simplemente desconocido. Las implementaciones actuales de máxima parsimonia generalmente tratan los valores desconocidos de la misma manera: las razones por las que los datos son desconocidos no tienen ningún efecto particular en el análisis. Efectivamente, el programa trata un ? como si mantuviera el estado que implicaría la menor cantidad de pasos adicionales en el árbol (ver más abajo), aunque este no es un paso explícito en el algoritmo.
Los datos genéticos son particularmente susceptibles a métodos filogenéticos basados en caracteres, como la máxima parsimonia, porque las secuencias de proteínas y nucleótidos son naturalmente discretas: una posición particular en una secuencia de nucleótidos puede ser adenina , citosina , guanina o timina / uracilo , o un espacio en la secuencia; una posición ( residuo ) en una secuencia de proteínas será uno de los aminoácidos básicos o un espacio en la secuencia. Por lo tanto, la puntuación de caracteres rara vez es ambigua, excepto en los casos en que los métodos de secuenciación no logran producir una asignación definitiva para una posición de secuencia particular. Los espacios en la secuencia a veces se tratan como caracteres, aunque no hay consenso sobre cómo deben codificarse.
Los personajes pueden ser tratados como desordenados u ordenados. Para un carácter binario (de dos estados), esto supone poca diferencia. Para un personaje de múltiples estados, se puede considerar que los personajes desordenados tienen el mismo "coste" (en términos de número de "eventos evolutivos") para cambiar de cualquier estado a cualquier otro; Complementariamente, no requieren pasar por estados intermedios. Los caracteres ordenados tienen una secuencia particular en la que los estados deben ocurrir a través de la evolución, de modo que pasar entre algunos estados requiere pasar por un intermedio. Se puede considerar que esto tiene diferentes costos para transferir entre diferentes pares de estados. En el ejemplo anterior del color de ojos, es posible dejarlo sin ordenar, lo que impone el mismo "coste" evolutivo para pasar de marrón-azul, verde-azul, verde-avellana, etc. Alternativamente, se podría ordenar marrón-avellana. -verde azul; Esto normalmente implicaría que costaría dos eventos evolutivos pasar del marrón-verde, tres del marrón-azul, pero sólo uno del marrón-avellana. También se puede considerar que esto requiere que los ojos evolucionen a través de una "etapa avellana" para pasar de marrón a verde, y una "etapa verde" para pasar de avellana a azul, etc. Para muchos personajes, no es obvio si y cómo. se deben ordenar. Por el contrario, para los caracteres que representan la discretización de una variable continua subyacente, como los caracteres de forma, tamaño y proporción, el orden es lógico [12] y las simulaciones han demostrado que esto mejora la capacidad de recuperar clados correctos, al tiempo que disminuye la recuperación de clados erróneos. clados. [13] [14] [15]
Existe un animado debate sobre la utilidad y adecuación del ordenamiento de los personajes, pero no hay consenso. Algunas autoridades ordenan los caracteres cuando hay una clara transición lógica, ontogenética o evolutiva entre los estados (por ejemplo, "piernas: cortas; medianas; largas"). Algunos aceptan sólo algunos de estos criterios. Algunos realizan un análisis desordenado y ordenan caracteres que muestran un orden claro de transición en el árbol resultante (práctica que podría acusarse de razonamiento circular ). Algunas autoridades se niegan a ordenar los caracteres en absoluto, sugiriendo que eso sesga un análisis al requerir transiciones evolutivas para seguir un camino particular.
También es posible aplicar ponderación diferencial a caracteres individuales. Esto generalmente se hace en relación con un "costo" de 1. Por lo tanto, es más probable que algunos caracteres reflejen las verdaderas relaciones evolutivas entre taxones y, por lo tanto, podrían ponderarse con un valor de 2 o más; Los cambios en estos caracteres contarían entonces como dos "pasos" evolutivos en lugar de uno al calcular las puntuaciones de los árboles (ver más abajo). Ha habido mucha discusión en el pasado sobre la ponderación de los personajes. La mayoría de las autoridades ahora ponderan todos los caracteres por igual, aunque las excepciones son comunes. Por ejemplo, los datos de frecuencia de alelos a veces se agrupan en contenedores y se califican como un carácter ordenado. En estos casos, el carácter en sí suele perder peso, de modo que los pequeños cambios en las frecuencias alélicas cuentan menos que los cambios importantes en otros caracteres. Además, la posición del tercer codón en una secuencia de nucleótidos codificante es particularmente lábil y, a veces, se le reduce el peso o se le asigna un peso de 0, suponiendo que es más probable que presente homoplasia. En algunos casos, se realizan análisis repetidos, con personajes reponderados en proporción inversa al grado de homoplasia descubierto en el análisis anterior (lo que se denomina ponderación sucesiva); ésta es otra técnica que podría considerarse razonamiento circular .
Los cambios de estado de los personajes también se pueden ponderar individualmente. Esto se hace a menudo con datos de secuencias de nucleótidos ; se ha determinado empíricamente que ciertos cambios de bases (AC, AT, GC, GT y sus cambios inversos) ocurren con mucha menos frecuencia que otros (AG, CT y sus cambios inversos). Por lo tanto, estos cambios suelen tener más peso. Como se muestra arriba en la discusión sobre el orden de los caracteres, los caracteres ordenados pueden considerarse como una forma de ponderación del estado de los caracteres.
Algunos sistemáticos prefieren excluir los caracteres que se sabe o se sospecha que son altamente homoplásticos o que tienen una gran cantidad de entradas desconocidas ("?"). Como se indica más adelante, el trabajo teórico y de simulación ha demostrado que es probable que esto sacrifique la precisión en lugar de mejorarla. Este también es el caso de los caracteres que son variables en los taxones terminales: los estudios teóricos, de congruencia y de simulación han demostrado que dichos caracteres polimórficos contienen información filogenética significativa. [ cita necesaria ]
El tiempo requerido para un análisis de parsimonia (o cualquier análisis filogenético) es proporcional al número de taxones (y caracteres) incluidos en el análisis. Además, debido a que más taxones requieren que se estimen más ramas, se puede esperar más incertidumbre en análisis grandes. Debido a que los costos de recolección de datos en tiempo y dinero a menudo escalan directamente con el número de taxones incluidos, la mayoría de los análisis incluyen solo una fracción de los taxones que podrían haberse muestreado. De hecho, algunos autores han sostenido que cuatro taxones (el mínimo requerido para producir un árbol sin raíces significativo) es todo lo que se necesita para un análisis filogenético preciso, y que más caracteres son más valiosos que más taxones en filogenética. Esto ha dado lugar a una gran controversia sobre el muestreo de taxones.
Los estudios empíricos, teóricos y de simulación han dado lugar a una serie de demostraciones espectaculares de la importancia de un muestreo adecuado de taxones. La mayoría de estos se pueden resumir en una simple observación: una matriz de datos filogenéticos tiene dimensiones de caracteres multiplicados por taxones. Duplicar el número de taxones duplica la cantidad de información en una matriz con la misma seguridad que duplicar el número de caracteres. Cada taxón representa una nueva muestra para cada carácter, pero, lo que es más importante, (normalmente) representa una nueva combinación de estados de carácter. Estos estados de carácter no sólo pueden determinar dónde se ubica ese taxón en el árbol, sino que también pueden informar todo el análisis, posiblemente provocando que se favorezcan diferentes relaciones entre los taxones restantes al cambiar las estimaciones del patrón de cambios de carácter.
La debilidad más preocupante del análisis de parsimonia, la de la atracción de ramas largas (ver más abajo), es particularmente pronunciada cuando el muestreo de taxones es deficiente, especialmente en el caso de cuatro taxones. Éste es un caso bien comprendido en el que un muestreo de caracteres adicional puede no mejorar la calidad de la estimación. A medida que se añaden taxones, a menudo rompen ramas largas (especialmente en el caso de los fósiles), mejorando efectivamente la estimación de los cambios de estado de carácter a lo largo de ellas. Debido a la riqueza de información agregada por el muestreo de taxones, es incluso posible producir estimaciones muy precisas de filogenias con cientos de taxones utilizando sólo unos pocos miles de caracteres. [ cita necesaria ]
Aunque se han realizado muchos estudios, todavía queda mucho trabajo por hacer sobre las estrategias de muestreo de taxones. Debido a los avances en el rendimiento de las computadoras y al costo reducido y la mayor automatización de la secuenciación molecular, los tamaños de las muestras en general están aumentando y los estudios que abordan las relaciones de cientos de taxones (u otras entidades terminales, como los genes) se están volviendo comunes. Por supuesto, esto no quiere decir que añadir caracteres no sea también útil; El número de personajes también está aumentando.
Algunos sistemáticos prefieren excluir taxones basándose en el número de entradas de caracteres desconocidos ("?") que exhiben, o porque tienden a "saltar" el árbol en los análisis (es decir, son "comodines"). Como se indica más adelante, el trabajo teórico y de simulación ha demostrado que es probable que esto sacrifique la precisión en lugar de mejorarla. Aunque estos taxones pueden generar árboles más parsimoniosos (ver más abajo), métodos como los subárboles de acuerdo y el consenso reducido aún pueden extraer información sobre las relaciones de interés.
Se ha observado que la inclusión de más taxones tiende a reducir los valores de soporte generales ( porcentajes de arranque o índices de descomposición, ver más abajo). La causa de esto es clara: a medida que se añaden taxones adicionales a un árbol, subdividen las ramas a las que se unen y, por tanto, diluyen la información que sustenta esa rama. Si bien se reduce el apoyo a las ramas individuales, en realidad aumenta el apoyo a las relaciones generales. Considere el análisis que produce el siguiente árbol: (pez, (lagarto, (ballena, (gato, mono)))). Agregar una rata y una morsa probablemente reducirá el apoyo al clado (ballena, (gato, mono)), porque la rata y la morsa pueden caer dentro de este clado, o fuera del clado, y dado que estos cinco animales son todos relativamente estrechamente relacionados, debería haber más incertidumbre sobre sus relaciones. Dentro del error, puede ser imposible determinar las relaciones de estos animales entre sí. Sin embargo, la rata y la morsa probablemente agregarán datos de carácter que consoliden el agrupamiento de dos de estos mamíferos, excluyendo al pez o al lagarto; donde el análisis inicial podría haber sido engañado, digamos, por la presencia de aletas en el pez y la ballena, la presencia de la morsa, con grasa y aletas como una ballena pero bigotes como un gato y una rata, une firmemente a la ballena con los mamíferos.
Para hacer frente a este problema, los subárboles de concordancia, el consenso reducido y el análisis de doble desintegración buscan identificar relaciones respaldadas (en forma de "declaraciones de n-taxones", como la declaración de cuatro taxones "(pez, (lagarto, (gato , ballena)))") en lugar de árboles enteros. Si el objetivo de un análisis es un árbol resuelto, como es el caso de la filogenética comparada , estos métodos no pueden resolver el problema. Sin embargo, si la estimación del árbol tiene tan poco respaldo, los resultados de cualquier análisis derivado del árbol probablemente serán demasiado sospechosos para utilizarlos de todos modos.
Un análisis de máxima parsimonia se realiza de una manera muy sencilla. Los árboles se puntúan según el grado en que implican una distribución parsimoniosa de los datos de los caracteres. El árbol más parsimonioso para el conjunto de datos representa la hipótesis preferida de relaciones entre los taxones en el análisis.
Los árboles se puntúan (evalúan) utilizando un algoritmo simple para determinar cuántos "pasos" (transiciones evolutivas) se requieren para explicar la distribución de cada carácter. Un paso es, en esencia, un cambio de un estado de personaje a otro, aunque con personajes ordenados algunas transiciones requieren más de un paso. Contrariamente a la creencia popular, el algoritmo no asigna explícitamente estados de carácter particulares a los nodos (uniones de ramas) de un árbol: la menor cantidad de pasos puede implicar asignaciones y distribuciones de transiciones evolutivas múltiples e igualmente costosas. Lo que se optimiza es el número total de cambios.
Hay muchos más árboles filogenéticos posibles de los que se pueden buscar exhaustivamente para más de ocho taxones aproximadamente. Por tanto, se utilizan varios algoritmos para buscar entre los posibles árboles. Muchos de estos implican tomar un árbol inicial (normalmente el árbol favorito de la última iteración del algoritmo) y perturbarlo para ver si el cambio produce una puntuación más alta.
Los árboles resultantes de la búsqueda de parsimonia no tienen raíces: muestran todas las relaciones posibles de los taxones incluidos, pero carecen de información sobre los tiempos relativos de divergencia. El usuario elige una rama particular para enraizar el árbol. Luego se considera que esta rama está fuera de todas las demás ramas del árbol, que juntas forman un grupo monofilético . Esto imparte una sensación de tiempo relativo al árbol. La elección incorrecta de una raíz puede dar como resultado relaciones incorrectas en el árbol, incluso si el árbol es correcto en su forma no enraizada.
El análisis de parsimonia a menudo arroja una cantidad de árboles igualmente más parsimoniosos (MPT). Una gran cantidad de MPT a menudo se considera una falla analítica y se cree ampliamente que está relacionada con la cantidad de entradas faltantes ("?") en el conjunto de datos, caracteres que muestran demasiada homoplasia o la presencia de "comodines" topológicamente lábiles. taxones (a los que pueden faltar muchas entradas). Se han propuesto numerosos métodos para reducir el número de MPT, incluida la eliminación de caracteres o taxones con grandes cantidades de datos faltantes antes del análisis, la eliminación o reducción de peso de caracteres altamente homoplásticos (ponderación sucesiva) o la eliminación de taxones comodín (el método del tronco filogenético) a posteriori y luego volver a analizar los datos.
Numerosos estudios teóricos y de simulación han demostrado que los caracteres altamente homoplásticos, los caracteres y taxones con abundantes datos faltantes y los taxones "comodines" contribuyen al análisis. Aunque puede parecer que excluir caracteres o taxones mejora la resolución, el árbol resultante se basa en menos datos y, por lo tanto, es una estimación menos confiable de la filogenia (a menos que los caracteres o taxones no sean informativos, consulte reducción taxonómica segura). El consenso general actual es que tener múltiples MPT es un resultado analítico válido; simplemente indica que no hay datos suficientes para resolver el árbol por completo. En muchos casos, existe una estructura común sustancial en los MPT y las diferencias son leves e implican incertidumbre en la ubicación de algunos taxones. Hay varios métodos para resumir las relaciones dentro de este conjunto, incluidos árboles de consenso, que muestran relaciones comunes entre todos los taxones, y subárboles de acuerdo podados, que muestran una estructura común al podar temporalmente los taxones "comodines" de cada árbol hasta que todos estén de acuerdo. . El consenso reducido lleva esto un paso más allá, al mostrar todos los subárboles (y por lo tanto todas las relaciones) respaldados por los árboles de entrada.
Incluso si se devuelven múltiples MPT, el análisis de parsimonia sigue produciendo básicamente una estimación puntual, sin intervalos de confianza de ningún tipo. Esto a menudo se ha planteado como una crítica, ya que ciertamente hay un error al estimar el árbol más parsimonioso, y el método no incluye inherentemente ningún medio para establecer qué tan sensibles son sus conclusiones a este error. Se han utilizado varios métodos para evaluar el apoyo.
Con el análisis de parsimonia se han empleado jackknifing y bootstrapping , conocidos procedimientos de remuestreo estadístico . El jackknife, que implica un nuevo muestreo sin reemplazo ("dejar uno fuera"), se puede emplear en caracteres o taxones; la interpretación puede volverse complicada en el último caso, porque la variable de interés es el árbol y la comparación de árboles con diferentes taxones no es sencilla. El bootstrap, remuestreo con reemplazo (muestreo de x elementos aleatoriamente de una muestra de tamaño x, pero los elementos se pueden seleccionar varias veces), solo se usa en caracteres, porque agregar taxones duplicados no cambia el resultado de un análisis de parsimonia. El bootstrap se emplea mucho más comúnmente en filogenética (como en otros lugares); Ambos métodos implican un número arbitrario pero grande de iteraciones repetidas que implican la perturbación de los datos originales seguida del análisis. Los MPT resultantes de cada análisis se agrupan y los resultados generalmente se presentan en un árbol de consenso de regla de mayoría del 50%, con ramas (o nodos) individuales etiquetadas con el porcentaje de MPT de arranque en los que aparecen. Este "porcentaje de arranque" (que no es un valor P , como a veces se afirma) se utiliza como medida de apoyo. Técnicamente, se supone que es una medida de repetibilidad, la probabilidad de que esa rama (nodo, clado) se recupere si se muestrean nuevamente los taxones. Las pruebas experimentales con filogenias virales sugieren que el porcentaje de arranque no es un buen estimador de repetibilidad para la filogenética, pero es un estimador razonable de precisión. [ cita necesaria ] De hecho, se ha demostrado que el porcentaje de arranque, como estimador de precisión, está sesgado, y que este sesgo da como resultado en promedio una subestimación de la confianza (de modo que tan solo un 70% de apoyo realmente podría indicar un aumento al 95% de confianza). Sin embargo, la dirección del sesgo no se puede determinar en casos individuales, por lo que no se justifica asumir que los valores altos de soporte de arranque indican una confianza aún mayor.
Otro medio de evaluar el soporte es el soporte de Bremer, [16] [17] o el índice de decaimiento, que es un parámetro de un conjunto de datos determinado, en lugar de una estimación basada en submuestras pseudorreplicadas, como lo son los procedimientos bootstrap y jackknife descritos anteriormente. El soporte de Bremer (también conocido como soporte de rama) es simplemente la diferencia en el número de pasos entre la puntuación de los MPT y la puntuación del árbol más parsimonioso que no contiene un clado particular (nodo, rama). Puede considerarse como la cantidad de pasos que debes agregar para perder ese clado; implícitamente, pretende sugerir cuán grande debe ser el error en la estimación de la puntuación del MPT para que el clado ya no sea respaldado por el análisis, aunque esto no es necesariamente lo que hace. Los valores de soporte de sucursales suelen ser bastante bajos para conjuntos de datos de tamaño modesto (lo típico es uno o dos pasos), pero a menudo parecen ser proporcionales a los porcentajes de arranque. A medida que las matrices de datos se hacen más grandes, los valores de soporte de sucursales a menudo continúan aumentando a medida que los valores de arranque se estabilizan en 100%. Por lo tanto, para matrices de datos grandes, los valores de soporte de sucursales pueden proporcionar un medio más informativo para comparar el soporte de ramas fuertemente respaldadas. [18] Sin embargo, la interpretación de los valores de desintegración no es sencilla y parecen ser los preferidos por los autores con objeciones filosóficas al bootstrap (aunque muchos sistemáticos morfológicos, especialmente paleontólogos, informan sobre ambos). El análisis de doble desintegración es una contraparte de desintegración del consenso reducido que evalúa el índice de desintegración para todas las posibles relaciones de subárbol (declaraciones de n-taxones) dentro de un árbol.
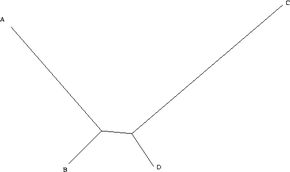
La máxima parsimonia es un enfoque epistemológicamente sencillo que hace pocos supuestos mecanicistas y es popular por esta razón. Sin embargo, puede no ser estadísticamente consistente bajo ciertas circunstancias. La coherencia, aquí significa la convergencia monótona hacia la respuesta correcta con la adición de más datos, es una propiedad deseable de los métodos estadísticos . Como lo demostró Joe Felsenstein en 1978 , [3] la parsimonia máxima puede ser inconsistente bajo ciertas condiciones. La categoría de situaciones en las que se sabe que esto ocurre se llama atracción de rama larga , y ocurre, por ejemplo, cuando hay ramas largas (un alto nivel de sustituciones) para dos personajes (A y C), pero ramas cortas para otros dos. (B y D). A y B divergieron de un ancestro común, al igual que C y D. Por supuesto, para saber que un método está dando una respuesta incorrecta, necesitaría saber cuál es la respuesta correcta. En general, este no es el caso en la ciencia. Por esta razón, algunos consideran que la coherencia estadística es irrelevante para las cuestiones filogenéticas empíricas. [19]
Por simplicidad, supongamos que estamos considerando un solo carácter binario (puede ser + o -). Debido a que la distancia de B a D es pequeña, en la gran mayoría de los casos, B y D serán la misma. Aquí, asumiremos que ambos son + (+ y - se asignan arbitrariamente e intercambiarlos es sólo una cuestión de definición). Si este es el caso, quedan cuatro posibilidades. A y C pueden ser ambos +, en cuyo caso todos los taxones son iguales y todos los árboles tienen la misma longitud. A puede ser + y C puede ser -, en cuyo caso sólo un carácter es diferente y no podemos aprender nada, ya que todos los árboles tienen la misma longitud. De manera similar, A puede ser - y C puede ser +. La única posibilidad que queda es que A y C sean ambos -. En este caso, sin embargo, la evidencia sugiere que A y C se agrupan juntos, y B y D juntos. Como consecuencia, si el "árbol verdadero" es un árbol de este tipo, cuantos más datos recopilemos (es decir, cuantos más caracteres estudiemos), más evidencia respaldará el árbol equivocado. Por supuesto, excepto en simulaciones matemáticas, nunca sabemos cuál es el "árbol verdadero". Por lo tanto, a menos que seamos capaces de diseñar un modelo que garantice la recuperación precisa del "árbol verdadero", cualquier otro criterio de optimización o esquema de ponderación también podría, en principio, ser estadísticamente inconsistente. La conclusión es que, si bien la inconsistencia estadística es una cuestión teórica interesante, empíricamente es una preocupación puramente metafísica, fuera del ámbito de las pruebas empíricas. Cualquier método puede ser inconsistente y no hay forma de saber con certeza si lo es o no. Es por esta razón que muchos sistemáticos caracterizan sus resultados filogenéticos como hipótesis de relación.
Otra complicación de la máxima parsimonia y otros métodos filogenéticos basados en criterios de optimización es que encontrar el árbol más corto es un problema NP difícil . [20] La única forma eficiente y disponible actualmente de obtener una solución, dado un conjunto arbitrariamente grande de taxones, es mediante el uso de métodos heurísticos que no garantizan que se recuperará el árbol más corto. Estos métodos emplean algoritmos de escalada para acercarse progresivamente al mejor árbol. Sin embargo, se ha demostrado que puede haber "islas de árboles" de soluciones subóptimas, y el análisis puede quedar atrapado en estos óptimos locales . Por lo tanto, se requieren heurísticas complejas y flexibles para garantizar que el espacio del árbol se haya explorado adecuadamente. Hay varias heurísticas disponibles, incluido el intercambio de vecinos más cercanos (NNI), la reconexión de bisección de árboles (TBR) y el trinquete de parsimonia.
Se ha afirmado que un problema importante, especialmente para la paleontología , es que la máxima parsimonia supone que la única forma en que dos especies pueden compartir el mismo nucleótido en la misma posición es si están relacionadas genéticamente. [ cita necesaria ] Esto afirma que las aplicaciones filogenéticas de la parsimonia suponen que toda similitud es homóloga (otras interpretaciones, como la afirmación de que dos organismos podrían no estar relacionados en absoluto, no tienen sentido). Este no es en absoluto el caso: como ocurre con cualquier forma de estimación de la filogenia basada en caracteres, la parsimonia se utiliza para probar la naturaleza homóloga de las similitudes encontrando el árbol filogenético que mejor explica todas las similitudes.
A menudo se afirma que la parsimonia no es relevante para la inferencia filogenética porque "la evolución no es parsimoniosa". [ cita necesaria ] En la mayoría de los casos, no se propone ninguna alternativa explícita; si no hay alternativa disponible, cualquier método estadístico es preferible a ninguno. Además, no está claro qué significaría si la afirmación "la evolución es parsimoniosa" fuera cierta. Esto podría interpretarse en el sentido de que históricamente pueden haber ocurrido más cambios de carácter de los que se predicen utilizando el criterio de parsimonia. Debido a que la estimación de la filogenia parsimonia reconstruye el número mínimo de cambios necesarios para explicar un árbol, esto es bastante posible. Sin embargo, se ha demostrado mediante estudios de simulación, pruebas con filogenias virales in vitro conocidas y congruencia con otros métodos, que la precisión de la parsimonia en la mayoría de los casos no se ve comprometida por esto. El análisis de parsimonia utiliza el número de cambios de caracteres en los árboles para elegir el mejor árbol, pero no requiere que exactamente esa cantidad de cambios, y no más, hayan producido el árbol. Siempre que los cambios que no se hayan tenido en cuenta se distribuyan aleatoriamente en el árbol (una expectativa nula razonable), el resultado no debe estar sesgado. En la práctica, la técnica es sólida: la parsimonia máxima exhibe un sesgo mínimo como resultado de elegir el árbol con la menor cantidad de cambios.
Se puede establecer una analogía con la elección entre contratistas basándose en su estimación inicial (no vinculante) del costo de un trabajo. Es muy probable que el costo final real sea mayor que el estimado. A pesar de esto, elegir al contratista que proporcionó la estimación más baja debería, en teoría, dar como resultado el costo final más bajo del proyecto. Esto se debe a que, en ausencia de otros datos, asumiríamos que todos los contratistas relevantes tienen el mismo riesgo de sobrecostos. En la práctica, por supuesto, las prácticas comerciales sin escrúpulos pueden sesgar este resultado; También en filogenética, algunos problemas filogenéticos particulares (por ejemplo, la atracción de ramas largas , descrita anteriormente) pueden sesgar potencialmente los resultados. Sin embargo, en ambos casos no hay forma de saber si el resultado va a estar sesgado o en qué grado estará sesgado, basándose en la estimación misma. También con parsimonia no hay forma de decir que los datos son claramente engañosos, sin compararlos con otras pruebas.
La parsimonia a menudo se caracteriza por adoptar implícitamente la posición de que el cambio evolutivo es raro o que la homoplasia (convergencia y reversión) es mínima en la evolución. Esto no es del todo cierto: la parsimonia minimiza el número de convergencias y reversiones que asume el árbol preferido, pero esto puede resultar en un número relativamente grande de tales eventos homoplásticos. Sería más apropiado decir que la parsimonia supone sólo la cantidad mínima de cambio implicada por los datos. Como se indicó anteriormente, esto no requiere que estos hayan sido los únicos cambios que ocurrieron; simplemente no infiere cambios de los que no hay evidencia. La abreviatura para describir esto, parafraseando a Farris [5] , es que "la parsimonia minimiza las homoplasias supuestas, no supone que la homoplasia sea mínima".
Estudios de simulación recientes sugieren que la parsimonia puede ser menos precisa que los árboles construidos utilizando enfoques bayesianos para los datos morfológicos, [21] potencialmente debido a una sobreprecisión, [22] aunque esto ha sido cuestionado. [23] Los estudios que utilizan nuevos métodos de simulación han demostrado que las diferencias entre los métodos de inferencia resultan de la estrategia de búsqueda y el método de consenso empleados, más que de la optimización utilizada. [24] Además, los análisis de 38 conjuntos de datos empíricos moleculares y 86 morfológicos han demostrado que el mecanismo común asumido por los modelos evolutivos utilizados en la filogenética basada en modelos se aplica a la mayoría de los conjuntos de datos moleculares, pero a pocos morfológicos. [25] Este hallazgo valida el uso de filogenética basada en modelos para datos moleculares, pero sugiere que para datos morfológicos, la parsimonia sigue siendo ventajosa, al menos hasta que estén disponibles modelos más sofisticados para datos fenotípicos.
Existen varios otros métodos para inferir filogenias basadas en datos de caracteres discretos, incluida la máxima verosimilitud y la inferencia bayesiana . Cada uno ofrece posibles ventajas y desventajas. En la práctica, estos métodos tienden a favorecer árboles que son muy similares a los árboles más parsimoniosos para el mismo conjunto de datos; [26] sin embargo, permiten el modelado complejo de procesos evolutivos y, como clases de métodos, son estadísticamente consistentes y no son susceptibles a la atracción de ramas largas . Tenga en cuenta, sin embargo, que el rendimiento de los métodos bayesianos y de probabilidad depende de la calidad del modelo particular de evolución empleado; un modelo incorrecto puede producir un resultado sesgado, al igual que la parsimonia. Además, todavía son bastante lentos desde el punto de vista computacional en comparación con los métodos de parsimonia, y a veces requieren semanas para ejecutar grandes conjuntos de datos. La mayoría de estos métodos tienen defensores y detractores particularmente ávidos; La parsimonia especialmente ha sido defendida como filosóficamente superior (sobre todo por ardientes cladistas ). [ cita necesaria ] Un área donde la parsimonia todavía tiene mucha influencia es en el análisis de datos morfológicos, porque, hasta hace poco, los modelos estocásticos de cambio de carácter no estaban disponibles para datos no moleculares y todavía no se implementan ampliamente. También se ha demostrado recientemente que la parsimonia tiene más probabilidades de recuperar el árbol verdadero frente a cambios profundos en los parámetros evolutivos ("modelo") (por ejemplo, la tasa de cambio evolutivo) dentro de un árbol. [27]
Las matrices de distancia también se pueden utilizar para generar árboles filogenéticos. Los métodos de distancia no paramétricos se aplicaron originalmente a datos fenéticos utilizando una matriz de distancias por pares y se reconciliaron para producir un árbol . La matriz de distancias puede provenir de varias fuentes diferentes, incluida la distancia inmunológica, el análisis morfométrico y las distancias genéticas . Para los datos de caracteres filogenéticos, los valores de distancia brutos se pueden calcular simplemente contando el número de diferencias por pares en los estados de los caracteres ( distancia de Manhattan ) o aplicando un modelo de evolución. En particular, los métodos a distancia también permiten el uso de datos que pueden no convertirse fácilmente en datos de caracteres, como los ensayos de hibridación ADN-ADN . Hoy en día, los métodos basados en distancias suelen estar mal vistos porque se pueden perder datos filogenéticamente informativos al convertir caracteres a distancias. Hay varios métodos de matriz de distancia y criterios de optimización, de los cuales el criterio de evolución mínima está más estrechamente relacionado con la parsimonia máxima.
Entre los métodos de distancia , existe un criterio de estimación filogenética, conocido como Evolución Mínima (ME), que comparte con la máxima parsimonia el aspecto de buscar la filogenia que tenga la suma total más corta de longitudes de ramas. [28] [29]
Una sutil diferencia distingue el criterio de máxima parsimonia del criterio ME: mientras que la máxima parsimonia se basa en una heurística abductiva, es decir, la plausibilidad de la hipótesis evolutiva más simple de los taxones con respecto a los más complejos, el criterio ME se basa en Las conjeturas de Kidd y Sgaramella-Zonta (que demostraron ser ciertas 22 años después por Rzhetsky y Nei [30] ) afirmaban que si las distancias evolutivas de los taxones fueran estimaciones imparciales de las verdaderas distancias evolutivas, entonces la verdadera filogenia de los taxones tendría una longitud más corta que cualquier otra. filogenia alternativa compatible con esas distancias. Los resultados de Rzhetsky y Nei liberan el criterio ME del principio de la navaja de Occam y le confieren una sólida base teórica y cuantitativa. [31]