En biología molecular , un marco de lectura es una forma de dividir la secuencia de nucleótidos de una molécula de ácido nucleico ( ADN o ARN ) en un conjunto de tripletes consecutivos que no se superponen. Cuando estos tripletes equivalen a aminoácidos o señales de parada durante la traducción , se denominan codones .
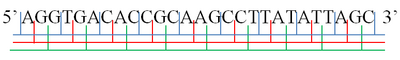
Una sola hebra de una molécula de ácido nucleico tiene un extremo fosforilo , llamado extremo 5' , y un extremo hidroxilo o 3' . Estos definen la dirección 5′→3′ . Hay tres marcos de lectura que se pueden leer en esta dirección 5′→3′, cada uno comenzando desde un nucleótido diferente en un triplete. En un ácido nucleico bicatenario, se pueden leer tres marcos de lectura adicionales de la otra hebra complementaria en la dirección 5'→3' a lo largo de esta hebra. Como las dos hebras de una molécula de ácido nucleico bicatenario son antiparalelas, la dirección 5′→3′ en la segunda hebra corresponde a la dirección 3′→5′ a lo largo de la primera hebra. [1] [2]
En general, como máximo, un marco de lectura en una sección determinada de un ácido nucleico es biológicamente relevante ( marco de lectura abierto ). Algunas transcripciones virales se pueden traducir utilizando múltiples marcos de lectura superpuestos. Hay un ejemplo conocido de marcos de lectura superpuestos en el ADN mitocondrial de mamíferos : porciones codificantes de genes para 2 subunidades de ATPasa se superponen.

El ADN codifica la secuencia de proteínas mediante una serie de codones de tres nucleótidos . Por lo tanto, cualquier secuencia de ADN puede leerse de seis maneras diferentes: tres marcos de lectura en una dirección (comenzando en diferentes nucleótidos) y tres en la dirección opuesta. Durante la transcripción , la ARN polimerasa lee la cadena de ADN molde en la dirección 3′→5′, pero el ARNm se forma en la dirección 5′ a 3′. [3] El ARNm es monocatenario y por lo tanto sólo contiene tres posibles marcos de lectura, de los cuales sólo uno está traducido . Los codones del marco de lectura del ARNm se traducen en la dirección 5′→3′ en aminoácidos mediante un ribosoma para producir una cadena polipeptídica .
Un marco de lectura abierto (ORF) es un marco de lectura que tiene el potencial de transcribirse en ARN y traducirse en proteínas. Requiere una secuencia continua de ADN que puede incluir un codón de inicio , a través de una región posterior que tiene una longitud múltiplo de 3 nucleótidos, hasta un codón de parada en el mismo marco de lectura. [4]
Cuando una supuesta secuencia de aminoácidos resultante de la traducción de un ORF seguía siendo desconocida en los genomas mitocondriales y de cloroplastos, el marco de lectura abierto correspondiente se denominaba marco de lectura no identificado (URF). Por ejemplo, el gen MT-ATP8 se describió por primera vez como URF A6L cuando se secuenció el genoma mitocondrial humano completo. [5]

El uso de múltiples marcos de lectura conduce a la posibilidad de que se superpongan genes ; Puede haber muchos de estos en genomas virales, procariotas y mitocondriales . [6] Algunos virus, por ejemplo el virus de la hepatitis B y el BYDV , utilizan varios genes superpuestos en diferentes marcos de lectura.
En casos raros, un ribosoma puede pasar de un marco a otro durante la traducción de un ARNm ( cambio de marco traslacional ). Esto hace que la primera parte del ARNm se traduzca en un marco de lectura y la última parte se traduzca en un marco de lectura diferente. Esto se diferencia de una mutación por cambio de marco , ya que la secuencia de nucleótidos (ADN o ARN) no se altera, solo el marco en el que se lee.