Un giro es un elemento de estructura secundaria en las proteínas donde la cadena polipeptídica invierte su dirección general.
Según una definición, un giro es un motivo estructural en el que los átomos de C α de dos residuos separados por unos pocos (normalmente de 1 a 5) enlaces peptídicos están cerca (menos de 7 Å [0,70 nm ]). [1] La proximidad de los átomos C α terminales a menudo se correlaciona con la formación de un enlace de hidrógeno entre las cadenas principales entre los residuos correspondientes. Estos enlaces de hidrógeno son la base de la definición de giro original, quizás más conocida. En muchos casos, pero no en todos, las definiciones de enlace de hidrógeno y distancia C α son equivalentes.
Las vueltas se clasifican [2] según la separación entre los dos residuos finales:
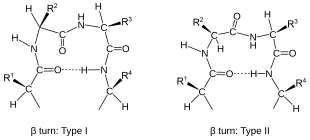
Los giros se clasifican según los ángulos de su columna vertebral (ver diagrama de Ramachandran ). Un giro se puede convertir en su giro inverso (en el que los átomos de la cadena principal tienen quiralidad opuesta ) cambiando el signo de sus ángulos diédricos. (El giro inverso no es un enantiómero verdadero ya que se mantiene la quiralidad del átomo de C α ). Por lo tanto, el giro γ tiene dos formas, una forma clásica con ángulos diédricos ( φ , ψ ) de aproximadamente (75°, −65°) y una forma inversa con ángulos diédricos (−75°, 65°). Se producen al menos ocho formas del giro beta , que varían según si está involucrado un isómero cis de un enlace peptídico y según los ángulos diédricos de los dos residuos centrales. Los giros β clásicos e inversos se distinguen con un primo, por ejemplo , giros beta tipo I y tipo I′ . Si se toma como criterio para los giros un enlace de hidrógeno i → i + 3, las cuatro categorías de Venkatachalam [4] (I, II, II′, I′) son suficientes [5] para describir todos los giros beta posibles . Los cuatro ocurren con frecuencia en las proteínas, pero I es el más común, seguido de II, I′ y II′ en ese orden.
Un bucle ω es un término general para un bucle más largo, extendido o irregular sin enlaces de hidrógeno internos fijos.
En muchos casos, uno o más residuos están involucrados en dos vueltas parcialmente superpuestas. Por ejemplo, en una secuencia de 5 residuos, tanto los residuos 1 a 4 como los residuos 2 a 5 forman una vuelta; en tal caso, se habla de un doble giro ( i , i + 1) . En las proteínas se producen comúnmente múltiples giros (hasta siete veces). [6] Las cintas de curvatura beta son un tipo diferente de giro múltiple.
Una horquilla es un caso especial de giro, en el que la dirección de la columna vertebral de la proteína se invierte y los elementos de la estructura secundaria que la flanquean interactúan. Por ejemplo, una horquilla beta conecta dos cadenas β antiparalelas unidas por enlaces de hidrógeno (un nombre bastante confuso, ya que una horquilla β puede contener muchos tipos de giros: α, β, γ, etc.).
Las horquillas beta se pueden clasificar según el número de residuos que forman la vuelta, es decir, que no forman parte de las hebras β flanqueantes. [7] Si este número es X o Y (según dos definiciones diferentes de hojas β), la horquilla β se define como X:Y.
Los giros beta en los extremos de las horquillas beta tienen una distribución de tipos diferente a los demás; el tipo I′ es el más común, seguido de los tipos II′, I y II.
A veces se encuentran giros dentro de enlazadores o bucles flexibles que conectan dominios de proteínas . Las secuencias enlazadoras varían en longitud y típicamente son ricas en aminoácidos polares no cargados . Los enlazadores flexibles permiten que los dominios conectados se giren y giren libremente para reclutar a sus socios de unión mediante la dinámica de dominios proteicos . También permiten que sus socios vinculantes induzcan cambios conformacionales a mayor escala mediante alostería de largo alcance . [8] [9] [10]
Se han propuesto dos hipótesis sobre el papel de los giros en el plegamiento de proteínas . Desde un punto de vista, los giros desempeñan un papel fundamental en el plegado al reunir y permitir interacciones entre elementos regulares de la estructura secundaria. Esta opinión está respaldada por estudios de mutagénesis que indican un papel crítico para residuos particulares en las espiras de algunas proteínas. Además, los isómeros no nativos de los enlaces peptídicos X- Pro , a su vez, pueden bloquear completamente el plegamiento conformacional de algunas proteínas. Desde el punto de vista opuesto, los giros desempeñan un papel pasivo en el plegado. Esta opinión está respaldada por la mala conservación de los aminoácidos observada en la mayoría de los turnos. Los isómeros no nativos de muchos enlaces peptídicos X-Pro , a su vez, también tienen poco o ningún efecto sobre el plegamiento.
A lo largo de los años, se han desarrollado muchos métodos de predicción de giros beta. Recientemente, el grupo del Dr. Raghava desarrolló el método BetaTPred3 que predice un giro beta completo en lugar de residuos individuales que caen en un giro beta. El método también logra una buena precisión y es el primer método que predice los 9 tipos de giros beta. Además de la predicción, este método también se puede utilizar para encontrar el número mínimo de mutaciones necesarias para iniciar o romper un giro beta en una proteína en una ubicación deseada.
{{cite book}}: |journal=ignorado ( ayuda )Estas referencias están ordenadas por fecha.
{{cite book}}: |journal=ignorado ( ayuda ){{cite book}}: |journal=ignorado ( ayuda )