
La espectrometría de masas de proteínas se refiere a la aplicación de la espectrometría de masas al estudio de proteínas . La espectrometría de masas es un método importante para la determinación de masas y la caracterización precisas de proteínas, y se han desarrollado una variedad de métodos e instrumentaciones para sus múltiples usos. Sus aplicaciones incluyen la identificación de proteínas y sus modificaciones postraduccionales , la elucidación de complejos proteicos, sus subunidades e interacciones funcionales, así como la medición global de proteínas en proteómica . También se puede utilizar para localizar proteínas en diversos orgánulos y determinar las interacciones entre diferentes proteínas, así como con los lípidos de membrana. [1] [2]
Los dos métodos principales utilizados para la ionización de proteínas en espectrometría de masas son la ionización por electropulverización (ESI) y la desorción/ionización por láser asistida por matriz (MALDI). Estas técnicas de ionización se utilizan junto con analizadores de masas como la espectrometría de masas en tándem . En general, las proteínas se analizan con un enfoque " de arriba hacia abajo ", en el que las proteínas se analizan intactas, o con un enfoque " de abajo hacia arriba ", en el que las proteínas se digieren primero en fragmentos. A veces también se puede utilizar un enfoque intermedio en el que se analizan fragmentos peptídicos más grandes.
La aplicación de la espectrometría de masas para estudiar proteínas se popularizó en la década de 1980 tras el desarrollo de MALDI y ESI . Estas técnicas de ionización han jugado un papel importante en la caracterización de proteínas. (MALDI) La ionización por desorción láser asistida por matriz fue acuñada a finales de la década de 1980 por Franz Hillenkamp y Michael Karas . [3] Hillenkamp, Karas y sus compañeros investigadores pudieron ionizar el aminoácido alanina mezclándolo con el aminoácido triptófano e irradiando con un láser de pulso de 266 nm. [3] Aunque importante, el avance no se produjo hasta 1987. En 1987, Koichi Tanaka utilizó el "método de matriz líquida y metal ultrafino" e ionizó biomoléculas del tamaño de 34.472 Da de proteína carboxipeptidasa-A. [4]
En 1968, Malcolm Dole informó sobre el primer uso de ionización por electropulverización con espectrometría de masas. Casi al mismo tiempo que se popularizó MALDI, se citó a John Bennett Fenn por el desarrollo de la ionización por electropulverización. [5] [6] Koichi Tanaka recibió el Premio Nobel de Química en 2002 junto con John Fenn y Kurt Wüthrich "por el desarrollo de métodos para la identificación y análisis de estructuras de macromoléculas biológicas". [7] Estos métodos de ionización han facilitado enormemente el estudio de proteínas mediante espectrometría de masas. En consecuencia, la espectrometría de masas de proteínas desempeña ahora un papel destacado en la caracterización de proteínas.
La espectrometría de masas de proteínas requiere que las proteínas en solución o en estado sólido se conviertan en una forma ionizada en fase gaseosa antes de inyectarlas y acelerarlas en un campo eléctrico o magnético para su análisis. Los dos métodos principales para la ionización de proteínas son la ionización por electropulverización (ESI) y la desorción/ionización por láser asistida por matriz (MALDI). En la electropulverización, los iones se crean a partir de proteínas en solución y permite ionizar intactas las moléculas frágiles, preservando a veces las interacciones no covalentes. En MALDI, las proteínas están incrustadas dentro de una matriz normalmente en forma sólida, y los iones se crean mediante pulsos de luz láser. El electrospray produce más iones con carga múltiple que MALDI, lo que permite medir proteínas de alta masa y una mejor fragmentación para su identificación, mientras que MALDI es rápido y es menos probable que se vea afectado por contaminantes, tampones y aditivos. [8]
El análisis de masa de proteínas completas se realiza principalmente mediante EM de tiempo de vuelo (TOF) o resonancia ciclotrón de iones por transformada de Fourier (FT-ICR). Estos dos tipos de instrumentos son preferibles aquí debido a su amplio rango de masas y, en el caso de FT-ICR, su alta precisión de masas. La ionización por electropulverización de una proteína a menudo da como resultado la generación de múltiples especies cargadas de 800 < m/z <2000 y el espectro resultante se puede desconvolucionar para determinar la masa promedio de la proteína dentro de 50 ppm o mejor usando TOF o instrumentos de trampa de iones .
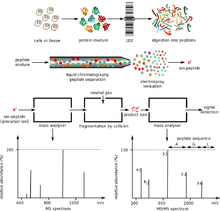
El análisis masivo de péptidos proteolíticos es un método popular de caracterización de proteínas, ya que para la caracterización se pueden utilizar diseños de instrumentos más económicos. Además, la preparación de muestras es más fácil una vez que las proteínas enteras se han digerido en fragmentos peptídicos más pequeños. El instrumento más utilizado para el análisis de masas de péptidos son los instrumentos MALDI-TOF, ya que permiten la adquisición de huellas dactilares de masas de péptidos (PMF) a gran velocidad (se puede analizar 1 PMF en aproximadamente 10 segundos). El tiempo de vuelo de cuadrupolo de múltiples etapas y la trampa de iones de cuadrupolo también encuentran uso en esta aplicación.

La espectrometría de masas en tándem (MS/MS) se utiliza para medir espectros de fragmentación e identificar proteínas a alta velocidad y precisión. La disociación inducida por colisión se utiliza en aplicaciones convencionales para generar un conjunto de fragmentos de un ion peptídico específico. El proceso de fragmentación da lugar principalmente a productos de escisión que rompen los enlaces peptídicos. Debido a esta simplicidad en la fragmentación, es posible utilizar las masas de los fragmentos observados para compararlas con una base de datos de masas predichas para una de las muchas secuencias peptídicas dadas. La EM en tándem de iones de proteínas completas se ha investigado recientemente mediante disociación por captura de electrones y, en principio, ha demostrado una amplia información de secuencia, pero no es una práctica común.
De acuerdo con el rendimiento y el rango de masas de los espectrómetros de masas disponibles, se utilizan dos enfoques para caracterizar proteínas. En el primero, las proteínas intactas se ionizan mediante cualquiera de las dos técnicas descritas anteriormente y luego se introducen en un analizador de masas. Este enfoque se conoce como estrategia " de arriba hacia abajo " de análisis de proteínas, ya que implica comenzar con toda la masa y luego separarla. Sin embargo, el enfoque de arriba hacia abajo se limita principalmente a estudios de una sola proteína de bajo rendimiento debido a los problemas relacionados con el manejo de proteínas completas, su heterogeneidad y la complejidad de sus análisis. [8]
En el segundo enfoque, denominado EM " de abajo hacia arriba ", las proteínas se digieren enzimáticamente en péptidos más pequeños utilizando una proteasa como la tripsina . Posteriormente, estos péptidos se introducen en el espectrómetro de masas y se identifican mediante huella peptídica de masas o espectrometría de masas en tándem . Por lo tanto, este enfoque utiliza la identificación a nivel de péptidos para inferir la existencia de proteínas reconstruidas con detección repetida de novo . [9] Los fragmentos más pequeños y uniformes son más fáciles de analizar que las proteínas intactas y también pueden determinarse con alta precisión; por lo tanto, este enfoque "de abajo hacia arriba" es el método preferido de estudios en proteómica. Un enfoque adicional que está empezando a ser útil es el enfoque intermedio "en el medio" en el que se analizan péptidos proteolíticos más grandes que los péptidos trípticos típicos. [8]
Las proteínas de interés suelen formar parte de una mezcla compleja de múltiples proteínas y moléculas que coexisten en el medio biológico. Esto presenta dos problemas importantes. En primer lugar, las dos técnicas de ionización utilizadas para moléculas grandes sólo funcionan bien cuando la mezcla contiene cantidades aproximadamente iguales de material, mientras que en muestras biológicas, diferentes proteínas tienden a estar presentes en cantidades muy diferentes. Si dicha mezcla se ioniza mediante electropulverización o MALDI , las especies más abundantes tienden a "ahogarse" o suprimir las señales de las menos abundantes. En segundo lugar, el espectro de masas de una mezcla compleja es muy difícil de interpretar debido a la abrumadora cantidad de componentes de la mezcla. Esto se ve agravado por el hecho de que la digestión enzimática de una proteína da lugar a una gran cantidad de productos peptídicos.
A la luz de estos problemas, los métodos de electroforesis en gel unidimensional y bidimensional y la cromatografía líquida de alta resolución se utilizan ampliamente para la separación de proteínas. El primer método fracciona proteínas enteras mediante electroforesis en gel bidimensional . La primera dimensión del gel 2D es el enfoque isoeléctrico (IEF). En esta dimensión, la proteína está separada por su punto isoeléctrico (pI) y la segunda dimensión es la electroforesis en gel de poliacrilamida-SDS (SDS-PAGE). Esta dimensión separa la proteína según su peso molecular. [10] Una vez que se completa este paso, se produce la digestión en gel. En algunas situaciones, puede ser necesario combinar ambas técnicas. Las manchas de gel identificadas en un gel 2D suelen ser atribuibles a una proteína. Si se desea conocer la identidad de la proteína, normalmente se aplica el método de digestión en gel , en el que la mancha de proteína de interés se escinde y se digiere proteolíticamente. Las masas peptídicas resultantes de la digestión se pueden determinar mediante espectrometría de masas utilizando la huella dactilar de masa peptídica . Si esta información no permite una identificación inequívoca de la proteína, sus péptidos pueden someterse a espectrometría de masas en tándem para una secuenciación de novo . Se pueden detectar pequeños cambios de masa y carga con 2D-PAGE. Las desventajas de esta técnica son su pequeño rango dinámico en comparación con otros métodos, algunas proteínas aún son difíciles de separar debido a su acidez, basicidad, hidrofobicidad y tamaño (demasiado grande o demasiado pequeño). [11]
El segundo método, la cromatografía líquida de alta resolución, se utiliza para fraccionar péptidos después de la digestión enzimática. La caracterización de mezclas de proteínas mediante HPLC/MS también se denomina proteómica de escopeta y MuDPIT (tecnología de identificación de proteínas multidimensional). Una mezcla de péptidos que resulta de la digestión de una mezcla de proteínas se fracciona mediante uno o dos pasos de cromatografía líquida. El eluyente de la etapa de cromatografía puede introducirse directamente en el espectrómetro de masas mediante ionización por electropulverización o depositarse en una serie de pequeños puntos para su posterior análisis de masas utilizando MALDI.
Hay dos formas principales en que se utiliza la EM para identificar proteínas. La toma de huellas dactilares de masas de péptidos utiliza masas de péptidos proteolíticos como entrada para una búsqueda en una base de datos de masas predichas que surgirían de la digestión de una lista de proteínas conocidas. Si una secuencia de proteínas en la lista de referencia da lugar a un número significativo de masas predichas que coinciden con los valores experimentales, existe alguna evidencia de que esta proteína estaba presente en la muestra original. Por lo tanto, los pasos de purificación limitan el rendimiento del enfoque de identificación de masas peptídicas. Alternativamente, los péptidos se pueden fragmentar con MS/MS para identificarlos más definitivamente. [9]
La EM también es el método preferido para la identificación de modificaciones postraduccionales en proteínas frente a otros enfoques, como los métodos basados en anticuerpos. [1]
La secuenciación de péptidos de novo para espectrometría de masas normalmente se realiza sin conocimiento previo de la secuencia de aminoácidos. Es el proceso de asignación de aminoácidos a partir de masas de fragmentos peptídicos de una proteína . La secuenciación de novo ha demostrado ser exitosa para confirmar y ampliar los resultados de las búsquedas en bases de datos.
Como la secuenciación de novo se basa en la masa y algunos aminoácidos tienen masas idénticas (por ejemplo, leucina e isoleucina ), la secuenciación manual precisa puede resultar difícil. Por lo tanto, puede ser necesario utilizar una aplicación de búsqueda de homología de secuencia para trabajar en conjunto entre una búsqueda en una base de datos y una secuenciación de novo para abordar esta limitación inherente.
La búsqueda en bases de datos tiene la ventaja de identificar rápidamente secuencias, siempre que ya hayan sido documentadas en una base de datos. Otras limitaciones inherentes a la búsqueda en bases de datos incluyen modificaciones/mutaciones de secuencias (algunas búsquedas en bases de datos no tienen en cuenta adecuadamente las alteraciones de la secuencia "documentada", por lo que pueden perderse información valiosa), lo desconocido (si una secuencia no está documentada, no se encontrará ), falsos positivos y datos incompletos y corruptos. [12]
También se puede utilizar una biblioteca espectral de péptidos anotada como referencia para la identificación de proteínas/péptidos. Ofrece la ventaja única de un espacio de búsqueda reducido y una mayor especificidad. Las limitaciones incluyen que los espectros no incluidos en la biblioteca no se identificarán, los espectros recopilados de diferentes tipos de espectrómetros de masas pueden tener características bastante distintas y los espectros de referencia en la biblioteca pueden contener picos de ruido, lo que puede dar lugar a identificaciones falsas positivas. [13] Se han descrito varios enfoques algorítmicos diferentes para identificar péptidos y proteínas mediante espectrometría de masas en tándem (MS/MS), secuenciación de novo de péptidos y búsqueda basada en etiquetas de secuencia. [14]
La presentación de antígenos es el primer paso para educar al sistema inmunológico para que reconozca nuevos patógenos. Para ello, las células presentadoras de antígenos exponen fragmentos de proteínas al sistema inmunológico a través de moléculas MHC . Sin embargo, no todos los fragmentos de proteínas se unen a las moléculas MHC de un determinado individuo. Mediante espectrometría de masas, se puede determinar el verdadero espectro de moléculas presentadas al sistema inmunológico. [15]

Múltiples métodos permiten la cuantificación de proteínas mediante espectrometría de masas [16] y avances recientes han permitido cuantificar miles de proteínas en células individuales. [17] [18] Normalmente, se incorporan a una muestra isótopos estables (por ejemplo, no radiactivos) más pesados de carbono ( 13 C) o nitrógeno ( 15 N), mientras que la otra se marca con los isótopos ligeros correspondientes (por ejemplo, 12 C y 14 NORTE). [19] Las dos muestras se mezclan antes del análisis. Los péptidos derivados de las diferentes muestras se pueden distinguir por su diferencia de masa. La proporción de sus intensidades máximas corresponde a la proporción de abundancia relativa de los péptidos (y proteínas). Los métodos más populares para el marcaje de isótopos son SILAC (marcaje de isótopos estables mediante aminoácidos en cultivos celulares), marcaje con 18 O catalizado por tripsina, ICAT (marcaje de afinidad codificado por isótopos), iTRAQ (etiquetas isobáricas para cuantificación relativa y absoluta). [20] La espectrometría de masas "semicuantitativa" se puede realizar sin etiquetar las muestras. [21] Normalmente, esto se hace con análisis MALDI (en modo lineal). La intensidad del pico, o el área del pico, de moléculas individuales (normalmente proteínas) se correlaciona aquí con la cantidad de proteína en la muestra. Sin embargo, la señal individual depende de la estructura primaria de la proteína, de la complejidad de la muestra y de la configuración del instrumento. Otros tipos de espectrometría de masas cuantitativa "sin etiquetas" utilizan los recuentos espectrales (o recuentos de péptidos) de proteínas digeridas como medio para determinar las cantidades relativas de proteínas. [12]
Las características indicativas de la estructura tridimensional de las proteínas pueden investigarse mediante espectrometría de masas de diversas formas. [22] La comparación de distribuciones de estados de carga puede proporcionar información sobre la estructura de una proteína. Una amplia variedad de estados de carga alta indica desorden de la proteína, mientras que las proteínas más compactas y plegadas dan como resultado estados de carga más bajos. [23] Mediante el uso de enlaces cruzados químicos para acoplar partes de la proteína que están cerca en el espacio, pero muy separadas en la secuencia, se puede inferir información sobre la estructura general. Siguiendo el intercambio de protones de amida con deuterio del disolvente, es posible investigar la accesibilidad al disolvente de varias partes de la proteína. [24] La espectrometría de masas de intercambio de hidrógeno-deuterio se ha utilizado para estudiar proteínas y sus conformaciones durante más de 20 años. Este tipo de análisis estructural de proteínas puede ser adecuado para proteínas que presentan desafíos para otros métodos estructurales. [25] Otra vía interesante en los estudios estructurales de proteínas es el marcaje covalente inducido por láser. En esta técnica, los sitios de la proteína expuestos al disolvente son modificados por radicales hidroxilo. Su combinación con mezcla rápida se ha utilizado en estudios de plegamiento de proteínas. [26]
En lo que ahora se conoce comúnmente como proteogenómica , los péptidos identificados con espectrometría de masas se utilizan para mejorar las anotaciones de genes (por ejemplo, sitios de inicio de genes) y las anotaciones de proteínas. El análisis paralelo del genoma y el proteoma facilita el descubrimiento de modificaciones postraduccionales y eventos proteolíticos, [27] especialmente cuando se comparan múltiples especies. [28]