Un ensayo de tetrámeros (también conocido como tinción de tetrámeros ) es un procedimiento que utiliza proteínas tetraméricas para detectar y cuantificar células T que son específicas para un antígeno dado dentro de una muestra de sangre. [1] Los tetrámeros utilizados en el ensayo están formados por cuatro moléculas del complejo mayor de histocompatibilidad (MHC), que se encuentran en la superficie de la mayoría de las células del cuerpo. [2] Las moléculas del MHC presentan péptidos a las células T como una forma de comunicar la presencia de virus, bacterias, mutaciones cancerosas u otros antígenos en una célula. Si el receptor de una célula T coincide con el péptido que presenta una molécula del MHC, se desencadena una respuesta inmunitaria. [3] Por lo tanto, los tetrámeros del MHC que se bioingenierizan para presentar un péptido específico se pueden utilizar para encontrar células T con receptores que coincidan con ese péptido. Los tetrámeros se marcan con un fluoróforo , lo que permite analizar las células T unidas al tetrámero con citometría de flujo . [4] La cuantificación y clasificación de las células T mediante citometría de flujo permite a los investigadores investigar la respuesta inmunitaria a la infección viral y la administración de vacunas, así como la funcionalidad de las células T específicas de antígeno. [5] En general, si el sistema inmunitario de una persona se ha encontrado con un patógeno , el individuo poseerá células T con especificidad hacia algún péptido de ese patógeno. Por lo tanto, si una tinción de tetrámero específica para un péptido patógeno da como resultado una señal positiva, esto puede indicar que el sistema inmunitario de la persona se ha encontrado con ese patógeno y ha creado una respuesta contra él. [ cita requerida ]
Esta metodología fue publicada por primera vez en 1996 por un laboratorio de la Universidad de Stanford. [6] Los intentos previos de cuantificar las células T específicas de antígenos implicaron el ensayo de dilución limitante menos preciso, que estima el número de células T entre 50 y 500 veces por debajo de sus niveles reales. [7] [8] Las tinciones que utilizan monómeros solubles de MHC tampoco tuvieron éxito debido a la baja afinidad de unión de los receptores de células T y los monómeros de péptidos de MHC. Los tetrámeros de MHC pueden unirse a más de un receptor en la célula T objetivo, lo que resulta en una mayor fuerza de unión total y tasas de disociación más bajas. [5]
Las tinciones de tetrámeros suelen analizar las poblaciones de linfocitos T citotóxicos (CTL). [9] Los CTL también se denominan células T CD8+, porque tienen correceptores CD8 que se unen a las moléculas MHC de clase I. La mayoría de las células del cuerpo expresan moléculas MHC de clase I, que son responsables de procesar antígenos intracelulares y presentarlos en la superficie de la célula. Si los péptidos que presentan las moléculas MHC de clase I son extraños (por ejemplo, derivados de proteínas virales en lugar de las propias proteínas de la célula), el CTL con un receptor que coincida con el péptido destruirá la célula. [2] [3] Las tinciones de tetrámeros permiten la visualización, cuantificación y clasificación de estas células mediante citometría de flujo, lo que es extremadamente útil en inmunología . Las poblaciones de células T se pueden rastrear durante la duración de un virus o después de la aplicación de una vacuna. Las tinciones de tetrámeros también se pueden combinar con ensayos funcionales como ELIspot , que detecta la cantidad de células secretoras de citocinas en una muestra. [9]
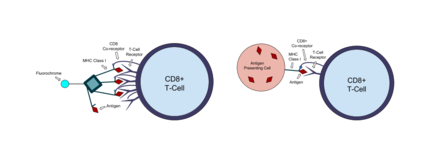
Las moléculas de tetrámero de MHC desarrolladas en un laboratorio pueden imitar el complejo presentador de antígenos en las células y unirse a las células T que reconocen el antígeno. Las moléculas de MHC de clase I están formadas por una cadena pesada α polimórfica asociada con una microglobulina beta-2 de cadena ligera invariante (β2m). Se utilizan Escherichia coli para sintetizar la cadena ligera y una versión acortada de la cadena pesada que incluye la etiqueta de reconocimiento de 15 aminoácidos de biotina . Estas cadenas de MHC se biotinilan con la enzima BirA y se repliegan con el péptido antigénico de interés. La biotina es una molécula pequeña que forma un enlace fuerte con otra proteína llamada estreptavidina. La estreptavidina marcada con fluoróforo se agrega a los monómeros de MHC biodiseñados, y la interacción biotina-estreptavidina hace que cuatro monómeros de MHC se unan a la estreptavidina y creen un tetrámero. Cuando los tetrámeros se mezclan con una muestra de sangre, se unirán a las células T que expresan el receptor específico de antígeno apropiado. Cualquier tetrámero de MHC que no esté unido se elimina de la muestra antes de analizarla con citometría de flujo. [9]
Los recientes avances en las moléculas de MHC recombinantes han democratizado la formulación de complejos de péptidos y MHC y su posterior multimerización. Las formulaciones altamente activas de una amplia gama de moléculas de MHC de clase I [10] ahora permiten a los usuarios no expertos elaborar sus propios complejos de péptidos y MHC personalizados día a día en cualquier laboratorio sin necesidad de equipo especial. [ cita requerida ]
También se han desarrollado tetrámeros que se unen a las células T colaboradoras. [9] Las células T colaboradoras o células T CD4+ expresan correceptores CD4 . Se unen a las moléculas MHC de clase II , que solo se expresan en células presentadoras de antígenos profesionales como las células dendríticas o los macrófagos . Las moléculas MHC de clase II presentan antígenos extracelulares, lo que permite a las células T colaboradoras detectar bacterias, [11] hongos y parásitos. [2] El uso de tetrámeros MHC de clase II se está volviendo más común, pero los tetrámeros son más difíciles de crear que los tetrámeros de clase I y el vínculo entre las células T colaboradoras y las moléculas MHC es aún más débil. [9] [12]
Las células T asesinas naturales (células NKT) también se pueden visualizar con tecnología de tetrámeros. Las células NKT se unen a proteínas que presentan antígenos lipídicos o glucolipídicos . [13] El complejo presentador de antígenos al que se unen las células NKT involucra proteínas CD1 , por lo que se pueden usar tetrámeros hechos de CD1 para teñir células NKT. [9]
Una de las primeras aplicaciones de la tecnología de los tetrámeros se centró en la respuesta inmunitaria mediada por células a la infección por VIH . Los tetrámeros MHC se desarrollaron para presentar antígenos del VIH y se utilizaron para encontrar el porcentaje de CTL específicos de esos antígenos del VIH en muestras de sangre de pacientes infectados. Esto se comparó con los resultados de los ensayos citotóxicos y la carga viral del ARN plasmático para caracterizar la función de los CTL en la infección por VIH. Los CTL que se unieron a los tetrámeros se clasificaron en pocillos ELIspot para el análisis de la secreción de citocinas. [14]
Otro estudio utilizó complejos de tetrámeros del MHC para investigar la eficacia de un método de administración de vacunas contra la gripe . Se administró a ratones vacunas contra la gripe por vía subcutánea e intranasal, y se utilizaron tinciones de tetrámeros acopladas a citometría de flujo para cuantificar los CTL específicos del antígeno utilizado en la vacuna. Esto permitió comparar la respuesta inmunitaria (la cantidad de células T que atacan a un virus) en dos métodos diferentes de administración de vacunas. [15]