En genética humana , el ancestro común más reciente del cromosoma Y ( Y-MRCA , informalmente conocido como Adán del cromosoma Y ) es el ancestro común más reciente patrilineal (MRCA) de quien descienden todos los humanos vivos en la actualidad . Es el varón más reciente de quien descienden todos los humanos vivos a través de una línea ininterrumpida de sus ancestros masculinos. El término Y-MRCA refleja el hecho de que los cromosomas Y de todos los varones humanos vivos en la actualidad se derivan directamente del cromosoma Y de este ancestro remoto. El concepto análogo del ancestro común más reciente matrilineal se conoce como " Eva mitocondrial " (mt-MRCA, llamada así por la transmisión matrilineal del ADNmt ), la mujer más reciente de quien descienden matrilinealmente todos los humanos vivos . Al igual que con "Eva mitocondrial", el título de "Adán del cromosoma Y" no está fijado permanentemente a un solo individuo, sino que puede avanzar a lo largo de la historia humana a medida que los linajes paternos se extinguen.
Las estimaciones de la época en que vivió el Y-MRCA también han cambiado a medida que cambia el conocimiento moderno sobre la ascendencia humana. Por ejemplo, en 2013, se anunció el descubrimiento de un haplogrupo del cromosoma Y previamente desconocido [4] , lo que dio lugar a un ligero ajuste de la edad estimada del Y-MRCA humano. [5]
Por definición, no es necesario que el Y-MRCA y el mt-MRCA hayan vivido al mismo tiempo. [6] Si bien las estimaciones de 2014 sugerían la posibilidad de que los dos individuos pudieran haber sido aproximadamente contemporáneos, [7] el descubrimiento del haplogrupo Y arcaico ha retrasado la edad estimada del Y-MRCA más allá de la edad más probable del mt-MRCA. A partir de 2015, las estimaciones de la edad del Y-MRCA oscilan entre 200.000 y 300.000 años atrás, aproximadamente en consonancia con el surgimiento de los humanos anatómicamente modernos . [2]
Los datos del cromosoma Y tomados de un neandertal de El Sidrón , España , produjeron un YT-MRCA (tiempo hasta Y-MRCA) de hace 588.000 años para los linajes paternos de neandertal y Homo sapiens , denominados ante Adán, y de hace 275.000 años para el Y-MRCA. [8]
El ancestro común más reciente del cromosoma Y es el ancestro común más reciente de los cromosomas Y que se encuentran en los varones humanos actualmente vivos.
Debido a la definición a través de la población "actualmente viva", la identidad de un MRCA, y por extensión del Y-MRCA humano, depende del tiempo (depende del momento en el tiempo al que se refiere el término "actualmente"). El MRCA de una población puede avanzar en el tiempo a medida que se extinguen linajes arcaicos dentro de la población: una vez que un linaje se ha extinguido, se pierde irremediablemente. Este mecanismo solo puede desplazar el título de Y-MRCA hacia adelante en el tiempo. Tal evento podría deberse a la extinción total de varios haplogrupos basales. [6] Lo mismo se aplica a los conceptos de MRCA matrilineales y patrilineales: de la definición de Y-MRCA se desprende que tuvo al menos dos hijos que tienen linajes ininterrumpidos que han sobrevivido hasta el día de hoy. Si los linajes de todos esos hijos, menos uno, se extinguen, el título de Y-MRCA pasa del hijo restante a sus descendientes patrilineales, hasta llegar al primer descendiente que tuvo al menos dos hijos que tengan descendientes patrilineales vivos. El título de Y-MRCA no está fijado permanentemente para un solo individuo, y el Y-MRCA de cualquier población dada habría sido parte de una población que tenía su propio Y-MRCA, más remoto.
Aunque el nombre informal "Adán del cromosoma Y" es una referencia al Adán bíblico , esto no debe malinterpretarse como una implicación de que el portador del cromosoma era el único hombre humano vivo durante su tiempo. [9] Sus otros contemporáneos masculinos también pueden tener descendientes vivos hoy, pero no, por definición, a través de descendencia únicamente patrilineal; en otras palabras, ninguno de ellos tiene una línea masculina ininterrumpida de descendientes ( hijo del hijo del hijo del hijo... hijo ) que los conecte con personas vivas actualmente.
Por la naturaleza del concepto de ancestros comunes más recientes, estas estimaciones sólo pueden representar un terminus ante quem ("límite antes del cual"), hasta que se haya examinado el genoma de toda la población (en este caso, el genoma de todos los humanos vivos).
Las estimaciones de la edad del Y-MRCA dependen fundamentalmente del haplogrupo más arcaico conocido que existe en las poblaciones contemporáneas. En 2018 [actualizar], este es el haplogrupo A00 (descubierto en 2013). Las estimaciones de edad basadas en este haplogrupo publicadas durante 2014-2015 oscilan entre 160.000 y 300.000 años, compatibles con el momento de la aparición y la dispersión temprana del Homo sapiens . [1] [2]
Además de la tendencia del título de Y-MRCA a desplazarse hacia adelante en el tiempo, la estimación de la secuencia de ADN de Y-MRCA, su posición en el árbol genealógico, el momento en que vivió y su lugar de origen están sujetos a futuras revisiones.
Los siguientes eventos cambiarían la estimación de quién era el individuo designado como Y-MRCA:
El tiempo en que el cromosoma Y-MRCA vivió se determina aplicando un reloj molecular a los cromosomas Y humanos. A diferencia del ADN mitocondrial (ADNmt), que tiene una secuencia corta de 16.000 pares de bases y muta con frecuencia, el cromosoma Y es significativamente más largo (60 millones de pares de bases) y tiene una tasa de mutación menor. Estas características del cromosoma Y han ralentizado la identificación de sus polimorfismos; como consecuencia, han reducido la precisión de las estimaciones de la tasa de mutación del cromosoma Y. [10]
Los métodos para estimar la edad del Y-MRCA para una población de varones humanos cuyos cromosomas Y han sido secuenciados se basan en la aplicación de las teorías de la evolución molecular al cromosoma Y. A diferencia de los autosomas , el cromosoma Y humano no se recombina a menudo con el cromosoma X durante la meiosis , sino que generalmente se transfiere intacto de padre a hijo; sin embargo, puede recombinarse con el cromosoma X en las regiones pseudoautosómicas en los extremos del cromosoma Y. [ cita requerida ] Las mutaciones ocurren periódicamente dentro del cromosoma Y, y estas mutaciones se transmiten a los varones en las generaciones posteriores.
Estas mutaciones se pueden utilizar como marcadores para identificar relaciones patrilineales compartidas. Los cromosomas Y que comparten una mutación específica se denominan haplogrupos . Se supone que los cromosomas Y dentro de un haplogrupo específico comparten un ancestro patrilineal común que fue el primero en portar la mutación definitoria. (Esta suposición podría ser errónea, ya que es posible que la misma mutación ocurra más de una vez). [ cita requerida ] Se puede construir un árbol genealógico de cromosomas Y, con las mutaciones que sirven como puntos de ramificación a lo largo de los linajes. El Y-MRCA se coloca en la raíz del árbol genealógico, ya que los cromosomas Y de todos los varones vivos descienden de su cromosoma Y.
Los investigadores pueden reconstruir secuencias de ADN del cromosoma Y ancestral revirtiendo los segmentos de ADN mutados a su estado original. El estado original o ancestral más probable de una secuencia de ADN se determina comparando las secuencias de ADN humano con las de una especie estrechamente relacionada, generalmente primates no humanos como los chimpancés y los gorilas. Al revertir las mutaciones conocidas en un linaje del cromosoma Y, se puede inferir una secuencia ancestral hipotética para el MRCA, el Adán del cromosoma Y.
Para determinar la secuencia de ADN del Y-MRCA y la época en la que vivió, es necesario identificar los linajes del cromosoma Y humano que son más divergentes entre sí (es decir, los linajes que comparten menos mutaciones entre sí en comparación con una secuencia de primates no humanos en un árbol filogenético ). Por lo tanto, el ancestro común de los linajes más divergentes es el ancestro común de todos los linajes.
Las primeras estimaciones de la edad del Y-MRCA publicadas durante la década de 1990 oscilaban entre aproximadamente 200 y 300 mil años atrás (kya). [11] Estas estimaciones fueron revisadas sustancialmente a la baja más tarde, como en Thomson et al. 2000, [10] que propuso una edad de aproximadamente 59.000. Esta fecha sugería que el Y-MRCA vivió unos 84.000 años después de su contraparte femenina mt-MRCA (el ancestro común matrilineal más reciente), que vivió hace 150.000-200.000 años. [12] Esta fecha también significaba que el Adán del cromosoma Y vivió en una época muy cercana a, y posiblemente después de, la migración desde África que se cree que tuvo lugar hace 50.000-80.000 años. Una explicación dada para esta discrepancia en las profundidades temporales de los linajes patrilineales vs. matrilineales fue que las mujeres tienen una mejor probabilidad de reproducirse que los hombres debido a la práctica de la poligamia . Cuando un individuo masculino tiene varias esposas, ha impedido efectivamente que otros hombres en la comunidad se reproduzcan y transmitan sus cromosomas Y a las generaciones posteriores. Por otro lado, la poligamia no impide que la mayoría de las mujeres en una comunidad transmitan su ADN mitocondrial a las generaciones posteriores. Este éxito reproductivo diferencial de hombres y mujeres puede llevar a que menos linajes masculinos en relación con los linajes femeninos persistan en el futuro. Estos menos linajes masculinos son más sensibles a la deriva y lo más probable es que se fusionen en un ancestro común más reciente. Esto potencialmente explicaría las fechas más recientes asociadas con el Y-MRCA. [13] [14]
La estimación "hiperreciente" de significativamente menos de 100 kya fue nuevamente corregida al alza en estudios de principios de la década de 2010, que oscilaron entre aproximadamente 120 kya y 160 kya. Esta revisión se debió a la reorganización de la estructura principal de la filogenia del cromosoma Y después de la resecuenciación de los linajes del haplogrupo A. [15] En 2013, Francalacci et al. informaron la secuenciación de polimorfismos de un solo nucleótido del cromosoma Y (MSY- SNP ) específicos de machos de 1204 machos sardos , lo que indicó una estimación de 180.000 a 200.000 años para el origen común de todos los humanos a través del linaje paterno. [16] [17] También en 2013, Poznik et al. Se informó que el Y-MRCA vivió entre 120.000 y 156.000 años atrás, basándose en la secuenciación del genoma de 69 hombres de 9 poblaciones diferentes. Además, el mismo estudio estimó la edad de Eva mitocondrial en alrededor de 99.000 y 148.000 años. [18] Como estos rangos se superponen durante un período de 28.000 años (148 a 120 kya), los resultados de este estudio se han presentado en términos de la posibilidad de que "Adán y Eva genéticos pudieron haber caminado sobre la Tierra al mismo tiempo" en la prensa popular. [7] [19]
El anuncio por Méndez et al. [4] del descubrimiento de un linaje previamente desconocido, el haplogrupo A00 , en 2013, resultó en otro cambio en la estimación de la edad del Adán del cromosoma Y. Los autores estimaron la división de los otros haplogrupos en hace 338.000 años (intervalo de confianza del 95% 237–581 kya), pero más tarde Elhaik et al. (2014) la dataron entre 163.900 y 260.200 años atrás ( IC del 95% ), [1] y Karmin et al. (2015) la dataron entre 192.000 y 307.000 años atrás ( IC del 95% ). [2] El mismo estudio informa que las poblaciones no africanas convergen en un grupo de Y-MRCA en una ventana cercana a los 50.000 años atrás ( migración fuera de África ), y un cuello de botella adicional para las poblaciones no africanas alrededor de los 10.000 años atrás, lo que se interpreta como un reflejo de los cambios culturales que aumentan la varianza en el éxito reproductivo masculino (es decir, una mayor estratificación social ) en el Neolítico . [2]
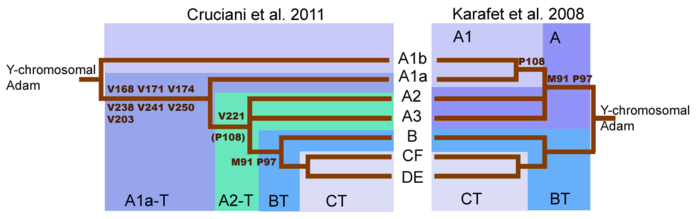
La secuenciación inicial (Karafet et al., 2008) del cromosoma Y humano sugirió que los dos linajes más basales del cromosoma Y eran el haplogrupo A y el haplogrupo BT . El haplogrupo A se encuentra en frecuencias bajas en partes de África, pero es común entre ciertos grupos de cazadores-recolectores. Los linajes del haplogrupo BT representan la mayoría de los linajes africanos del cromosoma Y y virtualmente todos los linajes no africanos. [20] El Adán del cromosoma Y fue representado como la raíz de estos dos linajes. El haplogrupo A y el haplogrupo BT representaron los linajes del propio Adán del cromosoma Y y de uno de sus hijos, que tenía un nuevo SNP .
Cruciani et al. 2011, determinaron que la división más profunda en el árbol genealógico del cromosoma Y se encontró entre dos subclados informados previamente del haplogrupo A, en lugar de entre el haplogrupo A y el haplogrupo BT. Más tarde, se encontró el grupo A00, fuera del árbol conocido previamente. La reorganización del árbol genealógico del cromosoma Y implica que los linajes clasificados como haplogrupo A no necesariamente forman un clado monofilético . [21] Por lo tanto, el haplogrupo A se refiere a una colección de linajes que no poseen los marcadores que definen al haplogrupo BT, aunque el haplogrupo A incluye los cromosomas Y más distantemente relacionados.
Las mutaciones M91 y P97 distinguen al haplogrupo A del haplogrupo BT. Dentro de los cromosomas del haplogrupo A, el marcador M91 consiste en un tramo de 8 unidades de nucleobases T. En el haplogrupo BT y en los cromosomas de chimpancé , este marcador consiste en 9 unidades de nucleobases T. Este patrón sugirió que el tramo de 9 T del haplogrupo BT era la versión ancestral y que el haplogrupo A se formó por la eliminación de una nucleobase . Los haplogrupos A1b y A1a se consideraron subclados del haplogrupo A ya que ambos poseían el M91 con 8 T. [20] [21]
Pero según Cruciani et al. 2011, la región que rodea al marcador M91 es un punto crítico mutacional propenso a mutaciones recurrentes. Por lo tanto, es posible que el tramo 8T del haplogrupo A pueda ser el estado ancestral de M91 y el 9T del haplogrupo BT pueda ser el estado derivado que surgió por una inserción de 1T. Esto explicaría por qué los subclados A1b y A1a-T, las ramas más profundas del haplogrupo A, poseen ambos la misma versión de M91 con 8T. Además, Cruciani et al. 2011 determinaron que el marcador P97, que también se utiliza para identificar el haplogrupo A, poseía el estado ancestral en el haplogrupo A pero el estado derivado en el haplogrupo BT. [21]
Como las estimaciones actuales sobre TMRCA convergen con las estimaciones de la edad de los humanos anatómicamente modernos y son muy anteriores a la migración fuera de África , las hipótesis de origen geográfico continúan limitadas al continente africano . [ cita requerida ]
Según Cruciani et al. 2011, la mayoría de los linajes basales se han detectado en África occidental , noroccidental y central , lo que sugiere la plausibilidad de que el Y-MRCA viva en la región general de "África central y noroccidental". [22]
Scozzari et al. (2012) coincidieron en que el haplogrupo A1b podría haber surgido en el "cuadrante noroccidental del continente africano". [23] El informe de 2013 sobre el haplogrupo A00 encontrado entre el pueblo Mbo del oeste del actual Camerún también es compatible con esta imagen. [4]
La revisión de la filogenia del cromosoma Y desde 2011 ha afectado las estimaciones del probable origen geográfico del Y-MRCA, así como las estimaciones sobre la profundidad temporal. Por el mismo razonamiento, el descubrimiento futuro de haplogrupos arcaicos actualmente desconocidos en personas vivas conduciría nuevamente a tales revisiones. En particular, la posible presencia de entre el 1% y el 4% de ADN derivado de Neandertal en genomas euroasiáticos implica que el (improbable) evento de un descubrimiento de un solo varón euroasiático vivo que exhiba una línea patrilineal neandertal retrasaría inmediatamente el T-MRCA ("tiempo hasta MRCA") al menos al doble de su estimación actual. Sin embargo, el descubrimiento de un cromosoma Y neandertal por Méndez et al . [8] sugiere la extinción de los linajes patrilineales neandertales, ya que el linaje inferido a partir de la secuencia neandertal está fuera del rango de variación genética humana contemporánea . Las cuestiones de origen geográfico se convertirían en parte del debate sobre la evolución neandertal a partir del Homo erectus .