Caulimoviridae es una familia de virus que infectan plantas. [1] Hay 94 especies en esta familia, asignadas a 11 géneros. [2] [3] Los virus que pertenecen a la familia Caulimoviridae se denominan virus de transcripción inversa de ADN bicatenario (dsDNA) (o pararetrovirus), es decir, virus que contienen una etapa de transcripción inversa en su ciclo de replicación. Esta familia contiene todos los virus de plantas con un genoma de dsDNA que tienen una fase de transcripción inversa en su ciclo de vida.
Se reconocen los siguientes géneros: [3]
Todos los virus de esta familia no tienen envoltura. Las partículas virales son baciliformes o isométricas. El tipo de nucleocápside incorporada a la estructura del virus determina el tamaño de las partículas virales. Las partículas baciliformes tienen un diámetro de aproximadamente 35 a 50 nm y una longitud de hasta 900 nm. Las partículas isométricas tienen un diámetro promedio de 45 a 50 nm y presentan simetría icosaédrica.
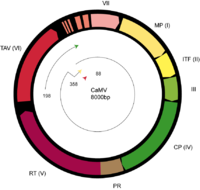
Los genomas de los virus de esta familia contienen dsADN circular monopartito, cerrado de forma no covalente, de 7,2 a 9,3 kbp con discontinuidades en ambas cadenas del genoma en lugares específicos. Estos genomas contienen desde un marco de lectura abierto (ORF), como se observa en los petuvirus , hasta ocho ORF, como en los soymovirus . Las proteínas codificadas por los genomas virales incluyen la transcriptasa inversa -ribonucleasa H, las proteasas aspárticas , las nucleocápsides y los transactivadores; hay otras proteínas esenciales para la replicación a las que aún no se les ha asignado una función específica.
La replicación tiene lugar tanto en el citoplasma como en el núcleo de las células huésped. En primer lugar, el genoma viral entra en el citoplasma . El ADN viral forma estructuras superenrolladas de minicromosomas al entrar en el núcleo del huésped, donde se transcribe en ARN poliadenilado que es terminalmente redundante (debido a que la transcripción ocurre dos veces para algunas partes del ADN). El ARN recién transcrito entra en el citoplasma donde se traduce en proteínas virales o se retrotranscribe en nuevas copias del genoma viral de dsADN por la transcriptasa inversa viral . Los nuevos genomas de dsADN se encapsidan en el citoplasma y se liberan.
El proceso de replicación implica un paso de retrotranscripción y un ARN intermediario, por lo que los virus de la familia Caulimoviridae no se consideran verdaderos virus dsADN , sino que se denominan virus de transcripción inversa de ADN. Comparten esta característica con los retrovirus . Sin embargo, a diferencia de estos, los virus de la familia Caulimoviridae no requieren la integración del genoma viral en el genoma de sus hospedadores para replicarse y, por esta razón, su genoma no codifica la proteína enzimática integrasa .
La presencia de elementos virales endógenos (EVE) en los genomas de las plantas está muy extendida. [4] [5] [6] y la mayoría de los EVE de plantas conocidos se originan a partir de virus con genomas de ADN de la familia Caulimoviridae . Se cree que la integración se produce a través de la unión de extremos no homólogos (recombinación ilegítima) durante los mecanismos de reparación del ADN. La mayoría de los EVE de plantas no son infecciosos. Sin embargo, se han descrito EVE Caulimoviridae infecciosos en el genoma de la petunia [7] ( Virus de aclaramiento de las venas de la petunia ), el plátano [8] ( Virus de la raya del plátano OL , Virus de la raya del plátano GF , Virus de la raya del plátano IM ) y Nicotiana edwardsonii [9] ( Virus de aclaramiento de las venas del tabaco ).
{{cite book}}: Mantenimiento de CS1: otros ( enlace )