El análisis C 0 t , una técnica basada en los principios de la cinética de reasociación del ADN , es una técnica bioquímica que mide la cantidad de ADN repetitivo en una muestra de ADN como un genoma . [1] Se utiliza para estudiar la estructura y la organización del genoma y también se ha utilizado para simplificar la secuenciación de genomas que contienen grandes cantidades de secuencias repetitivas. [2]
El procedimiento implica calentar una muestra de ADN genómico hasta que se desnaturalice y adopte la forma monocatenaria, y luego enfriarla lentamente para que las hebras puedan volver a aparearse. Mientras la muestra se enfría, se toman medidas de la cantidad de ADN que tiene bases apareadas a cada temperatura.
La cantidad de ADN monocatenario y bicatenario se mide diluyendo rápidamente la muestra, lo que ralentiza la reasociación, y luego uniendo el ADN a una columna de hidroxiapatita . La columna se lava primero con una concentración baja de tampón de fosfato de sodio , que eluye el ADN monocatenario, y luego con concentraciones altas de fosfato, que eluye el ADN bicatenario. Luego se mide la cantidad de ADN en estas dos soluciones utilizando un espectrofotómetro .
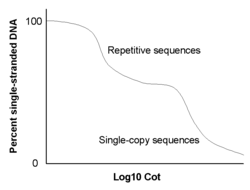
Como una secuencia de ADN monocatenario necesita encontrar su cadena complementaria para volver a formar una doble hélice, las secuencias comunes se renaturalizan más rápidamente que las secuencias raras. De hecho, la velocidad a la que una secuencia se reasocia es proporcional al número de copias de esa secuencia en la muestra de ADN. Una muestra con una secuencia altamente repetitiva se renaturalizará rápidamente, mientras que las secuencias complejas lo harán lentamente.
Sin embargo, en lugar de medir simplemente el porcentaje de ADN de doble cadena en función del tiempo, se mide la cantidad de renaturalización en relación con un valor C 0 t. El valor C 0 t es el producto de C 0 (la concentración inicial de ADN), t (tiempo en segundos) y una constante que depende de la concentración de cationes en el tampón. El ADN repetitivo se renaturalizará a valores bajos de C 0 t, mientras que las secuencias de ADN complejas y únicas se renaturalizarán a valores altos de C 0 t. La rápida renaturalización del ADN repetitivo se debe a la disponibilidad de numerosas secuencias complementarias.
La filtración C 0 t es una técnica que utiliza los principios de la cinética de renaturalización del ADN para separar las secuencias repetitivas de ADN que dominan muchos genomas eucariotas de las secuencias únicas o de bajo número de copias "ricas en genes". [2] Esto permite que la secuenciación del ADN se concentre en las partes del genoma que son más informativas e interesantes, lo que acelerará el descubrimiento de nuevos genes y hará que el proceso sea más eficiente. [3] [4]
Fue desarrollado y utilizado por primera vez por Roy Britten y sus colegas en la Institución Carnegie de Washington en la década de 1960. [5] [6] Cabe destacar que fue a través del análisis C 0 t que se descubrió por primera vez la naturaleza redundante (repetitiva) de los genomas eucariotas . [7] Sin embargo, no fue hasta los innovadores experimentos de cinética de reasociación de ADN de Britten y sus colegas que se demostró que no todo el ADN codifica genes. De hecho, sus experimentos demostraron que la mayoría del ADN genómico eucariota está compuesto de elementos repetitivos y no codificantes. [1]