La ADN polimerasa II (también conocida como ADN Pol II o Pol II ) es una ADN polimerasa dependiente de ADN procariota codificada por el gen PolB. [1]
La ADN polimerasa II es una proteína de 89,9 kDa y es miembro de la familia B de las ADN polimerasas. Fue aislada originalmente por Thomas Kornberg en 1970 y caracterizada en los siguientes años. [2] [3] [4] La funcionalidad in vivo de la Pol II está en debate, pero el consenso muestra que la Pol II está involucrada principalmente como una enzima de respaldo en la replicación del ADN procariota . La enzima tiene capacidad de síntesis de ADN 5′→3′ así como actividad de corrección de pruebas de exonucleasa 3′→5′ . La ADN Pol II interactúa con múltiples socios de unión comunes con la ADN Pol III para mejorar su fidelidad y procesividad . [1]
La ADN polimerasa I fue la primera ADN polimerasa dirigida por ADN que se aisló de E. coli . [5] Varios estudios que involucraron a esta enzima aislada indicaron que la ADN polimerasa I probablemente estaba involucrada en la replicación de reparación y no era la principal polimerasa replicativa. [6] Para comprender mejor el papel in vivo de la ADN polimerasa I, De Lucia y Cairns generaron en 1969 mutantes de E. coli deficientes en esta enzima (denominadas Pol A1 − ). [7] Como se caracterizó, esta nueva cepa mutante era más sensible a la luz ultravioleta , lo que corrobora la hipótesis de que la ADN polimerasa I estaba involucrada en la replicación de reparación. El mutante creció al mismo ritmo que el tipo salvaje , lo que indica la presencia de otra enzima responsable de la replicación del ADN . El aislamiento y la caracterización de esta nueva polimerasa involucrada en la replicación de ADN semiconservativa siguieron, en estudios paralelos realizados por varios laboratorios. [2] [3] [4] La nueva polimerasa se denominó ADN polimerasa II y se creyó que era la principal enzima replicativa de E. coli durante un tiempo. [8] La ADN pol II fue cristalizada por primera vez por Anderson et al. en 1994. [9]
En 2023 se informó que la transcripción acelerada relacionada con el envejecimiento hace que la Pol II cometa más errores, lo que genera copias defectuosas que pueden causar numerosas enfermedades. [10]
La DNA Pol II es una proteína de 89,9 kD, compuesta por 783 aminoácidos, codificada por el gen polB (dinA). La DNA Pol II es una proteína globular que funciona como un monómero, mientras que muchas otras polimerasas forman complejos. Hay tres secciones principales de este monómero, conocidas coloquialmente como palma, dedos y pulgar. Esta “mano” se cierra alrededor de una hebra de ADN. La palma del complejo contiene tres residuos catalíticos que se coordinarán con dos iones metálicos divalentes para funcionar. La DNA Pol II tiene una gran cantidad de copias en la célula, alrededor de 30-50, mientras que el nivel de DNA Pol III en una célula es cinco veces menor.
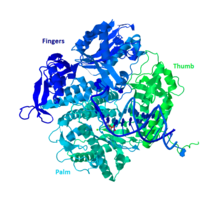
La mayoría de las polimerasas se han agrupado en familias en función de su estructura y función similares. La DNA Pol II pertenece al grupo B junto con las DNA Pol α, δ, ϵ y ζ humanas. Todas ellas son homólogas de RB69, 9°N-7 y Tgo. Los demás miembros del grupo B tienen al menos otra subunidad que hace que la DNA Pol II sea única. [11]
Todas las polimerasas participan de alguna manera en la replicación del ADN , sintetizando cadenas de ácidos nucleicos. La replicación del ADN es un aspecto vital de la proliferación celular. Sin replicar su ADN, una célula no puede dividirse y compartir su información genética con su progenie. En los procariotas, como E. coli , la ADN Pol III es la principal polimerasa involucrada en la replicación del ADN. Si bien la ADN Pol II no es un factor importante en la replicación cromosómica, tiene otras funciones que cumplir.
La DNA Pol II sí participa en la replicación del ADN. Si bien puede que no sea tan rápida como la DNA Pol III, tiene algunas capacidades que la convierten en una enzima eficaz. Esta enzima tiene una actividad exonucleasa 3′→5′ asociada junto con la actividad primasa . La DNA Pol II es una enzima de alta fidelidad con una tasa de error de sustitución de ≤ 2×10 −6 y una tasa de error de desplazamiento del marco de lectura de ≤ 1×10 −6 . La DNA Pol II puede corregir y procesar los desajustes causados por la Pol III. Banach-Orlowska et al. demostraron que la DNA Pol II está involucrada en la replicación, pero depende de la cadena y replica preferentemente la cadena rezagada . Un mecanismo propuesto sugiere que cuando la DNA Pol III se detiene o deja de funcionar, la DNA Pol II puede ser reclutada específicamente al punto de replicación y continuar la replicación. [1]
Existen muchas formas diferentes de dañar el ADN, desde daño por rayos UV hasta oxidación, por lo que es lógico que existan diferentes tipos de polimerasas para reparar estos daños. Un papel importante que cumple la Pol II del ADN es que es la principal polimerasa para la reparación de los enlaces cruzados entre cadenas. Los enlaces cruzados entre cadenas son causados por sustancias químicas como la mostaza nitrogenada y el psoraleno, que crean lesiones citotóxicas. Reparar estas lesiones es difícil porque ambas cadenas de ADN han sido dañadas por el agente químico y, por lo tanto, la información genética en ambas cadenas es incorrecta. El mecanismo exacto de cómo se reparan estos enlaces cruzados entre cadenas aún se está investigando, pero se sabe que la Pol II está muy involucrada. [11]
La ADN Pol II no es la polimerasa más estudiada, por lo que hay muchas funciones propuestas para esta enzima, que son todas funciones probables pero que en última instancia no están confirmadas: [1]

Durante la replicación del ADN, los pares de bases están sujetos a daños en la secuencia. Una secuencia de ADN dañada puede hacer que la replicación se detenga. [12] Para corregir un error en la secuencia, la ADN Pol II cataliza la reparación de los pares de bases de nucleótidos. Los estudios in vitro han demostrado que la Pol II ocasionalmente interactúa con las proteínas accesorias de la Pol III (β-clamp y complejo de carga de pinza) dando a la Pol II acceso a la hebra naciente en crecimiento. [1] [13] [14] [15] Con respecto a la función de la ADN Pol II durante la replicación del ADN, esto tiene sentido ya que cualquier error que produzca la Pol III estará en la hebra en crecimiento y no en la hebra conservativa. El dominio N-terminal de la ADN Pol II es responsable de la asociación y disociación de la hebra de ADN a la subunidad catalítica. Lo más probable es que haya dos sitios en el dominio N-terminal de la ADN Pol II que reconozcan el ADN monocatenario. Un sitio(s) es responsable de reclutar ADN monocatenario a la ADN Pol II y otro sitio(s) es responsable de la disociación del ADN monocatenario de la ADN Pol II. [16]

Tras la unión del sustrato, la DNA Pol II se une a los trifosfatos de nucleósido para mantener la estructura del ADN unida por enlaces de hidrógeno. A continuación, se une el dNTP correcto y el complejo enzimático sufre cambios conformacionales de subdominios y residuos de aminoácidos. Estos cambios conformacionales permiten que la velocidad de la síntesis de reparación sea rápida. [17] El sitio activo contiene dos iones Mg2 + que se estabilizan mediante los ácidos aspárticos catalíticos D419 y D547. [18] Los iones de magnesio se unen al ADN junto con el dNTP en el estado abierto y coordinan los cambios conformacionales de los residuos de aminoácidos del sitio activo para que se produzca la catálisis (estado cerrado). Una vez que se liberan los iones de magnesio, la enzima vuelve a su estado abierto. [19]
La ADN polimerasa II es un miembro de la familia de las polimerasas B y ayuda a la polimerasa III en la replicación del ADN, moviéndose desde el extremo 3′ hasta el extremo 5′. [20] En el caso en que la polimerasa III se detenga durante un error de replicación, la polimerasa II puede interrumpir y extirpar las bases no coincidentes. La polimerasa II tiene un factor de fidelidad mucho mayor que la polimerasa III, lo que significa que es mucho menos probable que cree emparejamientos incorrectos. Sin el paso de corrección de la polimerasa II, la polimerasa III extendería los emparejamientos incorrectos y, por lo tanto, crearía una mutación. [1]
Además de proteger contra las mutaciones que podría causar la polimerasa III, la polimerasa II funciona para proteger contra las mutaciones causadas por la polimerasa IV. La polimerasa IV es mucho más propensa a errores que la polimerasa II, pero también funciona para reparar los pares de bases desapareados comenzando desde el extremo 3'. La polimerasa II protege el extremo 3' de la polimerasa IV y evita que actúe. Esta protección evitará la formación de mutaciones mientras la polimerasa II esté funcionando normalmente. Si la polimerasa II es eliminada por una mutación o deshabilitada por otros factores, la polimerasa IV ocupará su lugar para reparar las bases desapareadas. [1]
Si bien la polimerasa II no funciona de manera natural junto con los miembros eucariotas de la familia B, sí comparte motivos estructurales y funcionales similares. Los miembros de la familia B incluyen las polimerasas α, ε, ζ y δ. Todas estas polimerasas funcionan para corregir el ADN recién sintetizado en la dirección 3′→5′. Estas polimerasas son capaces de sintetizar ADN tanto en la cadena principal como en la cadena rezagada. Esta clase de polimerasa tiende a ser muy precisa, lo que les permite corregir cualquier apareamiento incorrecto que ocurra durante la síntesis de ADN. [20]
La ADN polimerasa II es naturalmente abundante en la célula, lo que suele ser cinco veces mayor que la cantidad de polimerasa III. Esta mayor abundancia permite que la polimerasa II supere a la polimerasa III en caso de apareamientos incorrectos. Esta cantidad puede aumentarse mediante la inducción de la respuesta SOS, que regula positivamente el gen polB, de modo que la cantidad de polimerasa II aumenta hasta aproximadamente siete veces más. Aunque la polimerasa II puede actuar en ambas hebras, se ha demostrado que prefiere la hebra rezagada frente a la hebra líder. [1]