El índice de Jaccard es una estadística que se utiliza para medir la similitud y diversidad de conjuntos de muestras . Se define en general tomando la relación de dos tamaños (áreas o volúmenes), el tamaño de la intersección dividido por el tamaño de la unión, también llamado intersección sobre unión ( IoU ).
Fue desarrollado por Grove Karl Gilbert en 1884 como su índice de verificación (v) [1] y ahora a menudo se le llama índice de éxito crítico en meteorología. [2] Posteriormente fue desarrollado independientemente por Paul Jaccard , dando originalmente el nombre francés de coeficiente de communauté (coeficiente comunitario), [3] [4] y formulado independientemente nuevamente por T. Tanimoto. [5] Por lo tanto, también se le llama índice de Tanimoto o coeficiente de Tanimoto en algunos campos.
El coeficiente de Jaccard mide la similitud entre conjuntos de muestras finitas y se define como el tamaño de la intersección dividido por el tamaño de la unión de los conjuntos de muestras:
Tenga en cuenta que, por diseño, si la intersección de A y B está vacía, entonces J ( A , B ) = 0. El coeficiente de Jaccard se utiliza ampliamente en informática, ecología, genómica y otras ciencias, donde se utilizan datos binarios o binarizados . Tanto los métodos de solución exacta como los de aproximación están disponibles para probar hipótesis con el coeficiente de Jaccard. [6]
La similitud de Jaccard también se aplica a las bolsas, es decir, a los multiconjuntos . Esta tiene una fórmula similar, [7] pero los símbolos utilizados representan la intersección de bolsas y la suma de bolsas (no la unión). El valor máximo es 1/2.
La distancia de Jaccard , que mide la disimilitud entre conjuntos de muestras, es complementaria al coeficiente de Jaccard y se obtiene restando el coeficiente de Jaccard de 1 o, equivalentemente, dividiendo la diferencia de los tamaños de la unión y la intersección de dos conjuntos por el tamaño de la unión:
Una interpretación alternativa de la distancia de Jaccard es la relación entre el tamaño de la diferencia simétrica y la unión. La distancia de Jaccard se utiliza habitualmente para calcular una matriz n × n para la agrupación y el escalamiento multidimensional de conjuntos de muestras n .
Esta distancia es una métrica de la colección de todos los conjuntos finitos. [8] [9] [10]
También existe una versión de la distancia de Jaccard para medidas , incluidas las medidas de probabilidad . Si es una medida en un espacio medible , entonces definimos el coeficiente de Jaccard por
y la distancia de Jaccard por
Se debe tener cuidado si o , ya que estas fórmulas no están bien definidas en estos casos.
El esquema de hash sensible a la localidad de permutaciones independientes min-wise MinHash se puede utilizar para calcular de manera eficiente una estimación precisa del coeficiente de similitud de Jaccard de pares de conjuntos, donde cada conjunto está representado por una firma de tamaño constante derivada de los valores mínimos de una función hash .
Dados dos objetos, A y B , cada uno con n atributos binarios , el coeficiente de Jaccard es una medida útil de la superposición que A y B comparten con sus atributos. Cada atributo de A y B puede ser 0 o 1. El número total de cada combinación de atributos para A y B se especifica de la siguiente manera:
Cada atributo debe caer en una de estas cuatro categorías, lo que significa que
El coeficiente de similitud de Jaccard, J , se da como
La distancia de Jaccard, d J , se da como
Se puede hacer una inferencia estadística basada en los coeficientes de similitud de Jaccard y, en consecuencia, en las métricas relacionadas. [6] Dados dos conjuntos de muestras A y B con n atributos, se puede realizar una prueba estadística para ver si una superposición es estadísticamente significativa . La solución exacta está disponible, aunque el cálculo puede ser costoso a medida que n aumenta. [6] Los métodos de estimación están disponibles ya sea mediante la aproximación de una distribución multinomial o mediante el método bootstrap. [6]
Cuando se utiliza para atributos binarios, el índice de Jaccard es muy similar al coeficiente de coincidencia simple . La principal diferencia es que el SMC tiene el término en su numerador y denominador, mientras que el índice de Jaccard no. Por lo tanto, el SMC cuenta tanto las presencias mutuas (cuando un atributo está presente en ambos conjuntos) como las ausencias mutuas (cuando un atributo está ausente en ambos conjuntos) como coincidencias y las compara con el número total de atributos en el universo, mientras que el índice de Jaccard solo cuenta la presencia mutua como coincidencias y la compara con el número de atributos que han sido elegidos por al menos uno de los dos conjuntos.
En el análisis de la cesta de compra , por ejemplo, la cesta de dos consumidores que deseamos comparar puede contener solo una pequeña fracción de todos los productos disponibles en la tienda, por lo que el SMC generalmente devolverá valores muy altos de similitudes incluso cuando las cestas tengan muy poca semejanza, lo que hace que el índice de Jaccard sea una medida de similitud más apropiada en ese contexto. Por ejemplo, considere un supermercado con 1000 productos y dos clientes. La cesta del primer cliente contiene sal y pimienta y la cesta del segundo contiene sal y azúcar. En este escenario, la similitud entre las dos cestas medida por el índice de Jaccard sería 1/3, pero la similitud se convierte en 0,998 utilizando el SMC.
En otros contextos, donde 0 y 1 llevan información equivalente (simetría), el SMC es una mejor medida de similitud. Por ejemplo, los vectores de variables demográficas almacenadas en variables ficticias , como el género, se compararían mejor con el SMC que con el índice de Jaccard, ya que el impacto del género en la similitud debería ser igual, independientemente de si masculino se define como un 0 y femenino como un 1 o al revés. Sin embargo, cuando tenemos variables ficticias simétricas, se podría replicar el comportamiento del SMC dividiendo las variables ficticias en dos atributos binarios (en este caso, masculino y femenino), transformándolos así en atributos asimétricos, lo que permite el uso del índice de Jaccard sin introducir ningún sesgo. Sin embargo, el SMC sigue siendo más eficiente computacionalmente en el caso de variables ficticias simétricas, ya que no requiere agregar dimensiones adicionales.
Si y son dos vectores con todos los valores reales , entonces su coeficiente de similitud de Jaccard (también conocido como similitud de Ruzicka) se define como
y la distancia de Jaccard (también conocida entonces como distancia de Soergel)
Con aún más generalidad, si y son dos funciones medibles no negativas en un espacio medible con medida , entonces podemos definir
donde y son operadores puntuales. Entonces la distancia de Jaccard es
Entonces, por ejemplo, para dos conjuntos medibles , tenemos donde y son las funciones características del conjunto correspondiente.
La similitud ponderada de Jaccard descrita anteriormente generaliza el índice de Jaccard a vectores positivos, donde un conjunto corresponde a un vector binario dado por la función indicadora , es decir . Sin embargo, no generaliza el índice de Jaccard a distribuciones de probabilidad, donde un conjunto corresponde a una distribución de probabilidad uniforme, es decir
Siempre es menor si los conjuntos difieren en tamaño. Si , y entonces
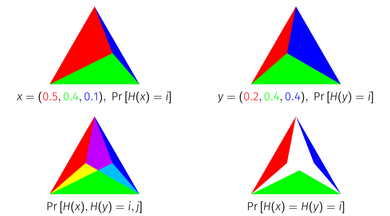
En cambio, una generalización que es continua entre distribuciones de probabilidad y sus conjuntos de soporte correspondientes es
que se llama Jaccard de "Probabilidad". [11] Tiene los siguientes límites contra el Jaccard ponderado en vectores de probabilidad.
Aquí, el límite superior es el coeficiente de Sørensen–Dice (ponderado) . La distancia correspondiente, , es una métrica sobre distribuciones de probabilidad y una pseudométrica sobre vectores no negativos.
El índice de probabilidad de Jaccard tiene una interpretación geométrica como el área de una intersección de símplices . Cada punto de un símplice unitario corresponde a una distribución de probabilidad sobre elementos, porque el símplice unitario es el conjunto de puntos en dimensiones que suman 1. Para derivar el índice de probabilidad de Jaccard geométricamente, represente una distribución de probabilidad como el símplice unitario dividido en subsímplices según la masa de cada elemento. Si superpone dos distribuciones representadas de esta manera una sobre otra e interseca los símplices correspondientes a cada elemento, el área que queda es igual al índice de probabilidad de Jaccard de las distribuciones.

Consideremos el problema de construir variables aleatorias de manera que colisionen entre sí tanto como sea posible. Es decir, si y , nos gustaría construir y para maximizar . Si observamos solo dos distribuciones de forma aislada, lo máximo que podemos lograr está dado por donde es la distancia de variación total . Sin embargo, supongamos que no solo nos preocupa maximizar ese par en particular, supongamos que nos gustaría maximizar la probabilidad de colisión de cualquier par arbitrario. Uno podría construir un número infinito de variables aleatorias, una para cada distribución , y buscar maximizar para todos los pares . En un sentido bastante fuerte descrito a continuación, el índice de probabilidad de Jaccard es una forma óptima de alinear estas variables aleatorias.
Para cualquier método de muestreo y distribuciones discretas , si entonces para algún lugar donde y , o bien . [11]
Es decir, ningún método de muestreo puede lograr más colisiones que en un par sin lograr menos colisiones que en otro par, donde el par reducido es más similar que el par aumentado. Este teorema es cierto para el índice de Jaccard de conjuntos (si se interpreta como distribuciones uniformes) y el Jaccard de probabilidad, pero no para el Jaccard ponderado. (El teorema utiliza la palabra "método de muestreo" para describir una distribución conjunta sobre todas las distribuciones en un espacio, porque se deriva del uso de algoritmos de minhashing ponderados que logran esto como su probabilidad de colisión).
Este teorema tiene una prueba visual en distribuciones de tres elementos utilizando la representación simplex.
En la literatura y en Internet se encuentran diversas formas de funciones descritas como similitud de Tanimoto y distancia de Tanimoto. La mayoría de ellas son sinónimos de similitud de Jaccard y distancia de Jaccard, pero algunas son matemáticamente diferentes. Muchas fuentes [12] citan un Informe técnico de IBM [5] como referencia fundamental.
En "Un programa informático para clasificar plantas", publicado en octubre de 1960, [13] se ofrece un método de clasificación basado en una relación de similitud y una función de distancia derivada. Parece que esta es la fuente más autorizada para el significado de los términos "similitud de Tanimoto" y "distancia de Tanimoto". La relación de similitud es equivalente a la similitud de Jaccard, pero la función de distancia no es la misma que la distancia de Jaccard.
En ese artículo, se proporciona una "relación de similitud" entre mapas de bits , donde cada bit de una matriz de tamaño fijo representa la presencia o ausencia de una característica en la planta que se está modelando. La definición de la relación es la cantidad de bits comunes, dividida por la cantidad de bits establecidos ( es decir, distintos de cero) en cada muestra.
Presentado en términos matemáticos, si las muestras X e Y son mapas de bits, es el i -ésimo bit de X , y son operadores bit a bit y , o respectivamente, entonces la relación de similitud es
Si cada muestra se modela como un conjunto de atributos, este valor es igual al coeficiente de Jaccard de los dos conjuntos. Jaccard no se cita en el artículo y parece probable que los autores no estuvieran al tanto de ello. [ cita requerida ]
Tanimoto continúa definiendo un "coeficiente de distancia" basado en esta relación, definido para mapas de bits con similitud distinta de cero:
Este coeficiente no es, deliberadamente, una métrica de distancia. Se ha elegido para permitir la posibilidad de que dos especímenes, que son bastante diferentes entre sí, sean ambos similares a un tercero. Es fácil construir un ejemplo que refute la propiedad de la desigualdad triangular .
A menudo se hace referencia a la distancia de Tanimoto, erróneamente, como sinónimo de la distancia de Jaccard . Esta función es una métrica de distancia adecuada. A menudo se afirma que la "distancia de Tanimoto" es una métrica de distancia adecuada, probablemente debido a su confusión con la distancia de Jaccard. [ aclaración necesaria ] [ cita necesaria ]
Si la similitud de Jaccard o Tanimoto se expresa sobre un vector de bits, entonces se puede escribir como
donde el mismo cálculo se expresa en términos de producto escalar vectorial y magnitud. Esta representación se basa en el hecho de que, para un vector de bits (donde el valor de cada dimensión es 0 o 1), entonces
y
Esta es una representación potencialmente confusa, porque la función tal como se expresa sobre vectores es más general, a menos que su dominio esté restringido explícitamente. Las propiedades de no necesariamente se extienden a . En particular, la función de diferencia no preserva la desigualdad triangular , y por lo tanto no es una métrica de distancia adecuada, mientras que es .
Existe un peligro real de que la combinación de la "Distancia de Tanimoto" definida utilizando esta fórmula, junto con la afirmación "La distancia de Tanimoto es una métrica de distancia adecuada", lleve a la falsa conclusión de que la función es, de hecho, una métrica de distancia sobre vectores o multiconjuntos en general, mientras que su uso en algoritmos de búsqueda de similitud o de agrupamiento puede no producir resultados correctos.
Lipkus [9] utiliza una definición de similitud de Tanimoto que es equivalente a , y se refiere a la distancia de Tanimoto como la función . Sin embargo, en el artículo se aclara que el contexto está restringido por el uso de un vector de ponderación (positivo) de modo que, para cualquier vector A que se considere, En estas circunstancias, la función es una métrica de distancia adecuada y, por lo tanto, un conjunto de vectores gobernados por dicho vector de ponderación forma un espacio métrico bajo esta función.
En las matrices de confusión empleadas para la clasificación binaria , el índice de Jaccard puede formularse en la siguiente fórmula:
donde TP son los verdaderos positivos, FP los falsos positivos y FN los falsos negativos. [14]